ORF和CDS的区别
ORF的英文展开是open reading frame(开放阅读框)。
CDS的英文展开是coding sequences (编码区)。
CDS:DNA转录成mRNA,mRNA经剪接等加工后翻译出蛋白质,所谓CDS就是与蛋白质序列一 一对应的DNA序列,且该序列中间不含其它非该蛋白质对应的序列,不考虑mRNA加工等过程中的序列变化,总之,就是与蛋白质的密码子完全对应.
ORF:理论上的氨基酸编码区,一般是在分析DNA核酸图谱中(主要是利用电脑程序)得到的。程序会自动在DNA序列中寻找启动因子(ATG或AUG),然后按每3个核酸一组,一直延伸寻找下去,直到碰到终止因子(TAA或TAG)。程序把这个区域当成ORF区,认为理论上可以编码一组氨基酸。
但问题是,在一个整体核酸序列中寻找ATG并不靠谱。因为寻找到的ATG很可能是两个氨基酸编码片段的尾和头的混合体。比如AACGCATGCAGC.
看上面这个小序列,如果以T为中心,会有三种编码组合的可能。即
(1)ATG(T在中心)电脑程序发现的启动因子的组合
(2)CAT(T在最右侧)
(3)TGC(T在最左侧)本例中实际核酸编码的组合。
这就是ORF三种框架的来源。实际上,DNA序列可以按六种框架阅读和翻译(每条链三种,对应六种不同的三联密码子)。
所以,我们说ORF只是理论上的编码区,与真实的情景可能并不一样。
而CDS是检查cDNA后得到的编码组合序列,和实际情景比较接近。
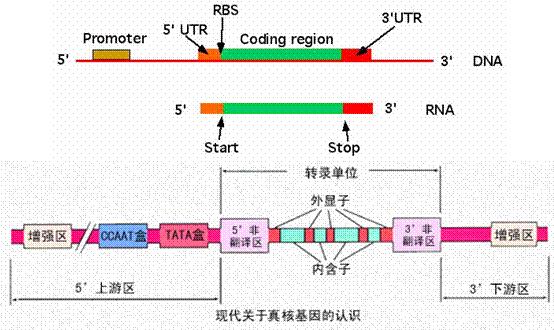
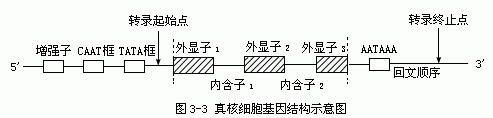
启动子与起始密码子、终止子与终止密码子有何区别?
启动子与起始密码子、终止子与终止密码子看起来似乎差不多,实际上却是两组截然不同的概念,根本就没有共同点。
简单地说,启动子和终止子都是一段特殊的DNA序列,属于基因的非编码区,分别位于编码区的上游和下游,负责调控基因的转录。而起始密码子和终止密码子都是mRNA上的三联体碱基序列,分别决定翻译的起始和终止。
启动子——DNA分子上能与RNA聚合酶结合并形成转录起始复合体的区域,在许多情况下,还包括促进这一过程的调节蛋白的结合位点。
强启动子(strong promoter),指对RNA聚合酶有很高亲和力的启动子,它能指导合成大量的mRNA。
起始密码子——蛋白质翻译过程中被核糖体识别并与起始tRNA(原核生物为甲酰甲硫氨酸tRNA,真核生物是甲硫氨酸tRNA)结合而作为肽链起始合成的信使核糖核酸(mRNA)三联体碱基序列。大部分情况下为AUG,原核生物中有时为GUG等。
终止子——转录过程中能够终止RNA聚合酶转录的DNA序列。使RNA合成终止。
终止密码子——蛋白质翻译过程中终止肽链合成的信使核糖核酸(mRNA)的三联体碱基序列。一般情况下为UAA、UAG和UGA,它们不编码氨基酸。
转录因子:转录因子(transcription factor)是一群能与基因5`端上游特定序列专一性结合,从而保证目的基因以特定的强度在特定的时间与空间表达的蛋白质分子。
转录因子的结合位点(transcription factor binding site,TFBS)是转录因子调节基因表达时,与基因模板链结合的区域。按照常识,转录因子(transcription factor,TF)的结合位点一般应该分布在基因的前端,但是,新的研究发现,人21和22号染色体上,只有22%的转录因子结合位点分布在蛋白编码基因的5'端。
UTR(Untranslated Regions)即非翻译区,是信使RNA(mRNA)分子两端的非编码片段。
5'-UTR从mRNA起点的甲基化鸟嘌呤核苷酸帽延伸至AUG起始密码子,3'-UTR从编码区末端的终止密码子延伸至多聚A尾巴(Poly-A)的前端。

参考:























 1万+
1万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








