Lefse分析
Lefse(LDA Effect Size)分析是一种用于发现和解释高维度数据的基因、通路和分类单元相关等生物标识的分析工具,通常可以进行两个或多个分组的比较,能够找出组与组之间有差异的生物标识。在微生物多样性分析中,Lefse可用来分析微生物的组间差异,如菌群差异等等。TUTUCLOUD云平台提供在线做Lefse分析的工具,可获得LDA值分布柱状图以及特征表。

TUTU网站工具使用
小编给大家介绍一个在线作图工具,lefse分析方法如下:
①登录网址:www.cloudtutu.com(推荐使用360或者谷歌浏览器)
②输入用户名和密码(小编已经为大家填好了,如果不显示可添加文末二维码添加小编获取),输入验证码后即可登录;
③登录后在全部工具那一栏找到lefse分析,点击进入;
④请按照界面右侧的说明书或者下文进行操作~
上传文件
※目前平台仅支持.txt(制表符分隔)文本文件或者.csv文件的文件上传。(平台可对不规范的数据格式进行部分处理,但还是请您尽量按照示例数据的格式调整数据,以便机器可以识别)
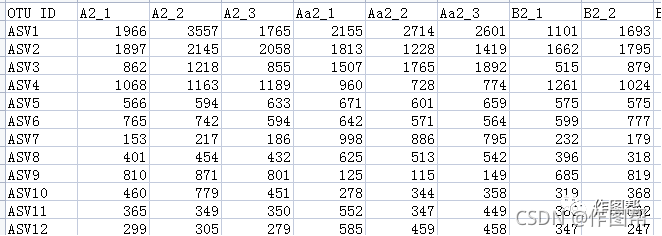
a)准备一个数据矩阵(形式参照示例数据,如微生物物种丰度表、基因表达量矩阵等等);
b)丰度文件表格需要带表头和列名,每一列为样本名,每一行为各种指标数据名,例如OTU、基因ID等等;
c)请提交txt(制表符分隔)文本文件或者.csv文件。操作方法为:全选excel中的所有内容(ctrl+A),复制到记事本中,将记事本文件另存后点击“上传”按钮上传该文件。
参数设置





 本文介绍了如何在TUTUCLOUD云平台上进行Lefse分析,用于揭示微生物多样性的组间差异。用户需上传制表符分隔或CSV格式的文件,并设置分组信息,平台提供PDF格式的矢量图输出。
本文介绍了如何在TUTUCLOUD云平台上进行Lefse分析,用于揭示微生物多样性的组间差异。用户需上传制表符分隔或CSV格式的文件,并设置分组信息,平台提供PDF格式的矢量图输出。
 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章

















 4746
4746

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








