随着高通量技术的发展,宏基因组学(metagenomics)已经成为研究微生物群落物种及功能的前沿科学,在肠道微生物、环境微生物等研究领域具有广泛应用。宏基因组学通过对微生物群落全部DNA进行高通量测序,将测序序列与公共数据库进行比对或从头组装出微生物基因组,从而识别微生物群落的物种和功能基因。
目前主流的宏基因组数据分析方法包括三种:基于测序结果进行组装的分析方法;基于reads直接和已知数据库进行比对的方法;基于分箱bin的方法。
常用到的分析工具见下图:
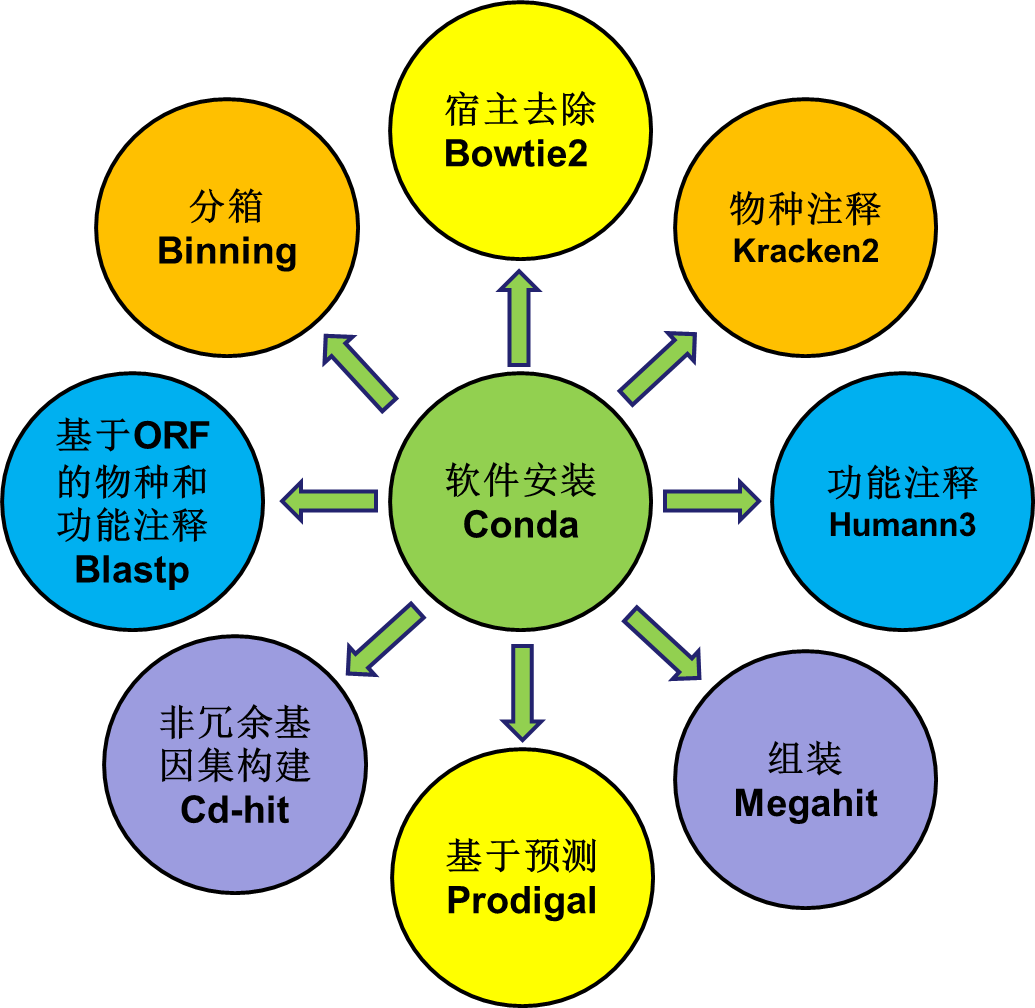
 本文介绍了宏基因组学的三种主流分析方法:基于组装,基于Reads的直接比对,以及基于Bin的分析。每种方法都有其优缺点,基于组装能得到ORF丰度信息,但分析时间长;基于Reads方法快速但无法覆盖未测序微生物;基于Bin分析则能组装出样本中菌株基因组。
本文介绍了宏基因组学的三种主流分析方法:基于组装,基于Reads的直接比对,以及基于Bin的分析。每种方法都有其优缺点,基于组装能得到ORF丰度信息,但分析时间长;基于Reads方法快速但无法覆盖未测序微生物;基于Bin分析则能组装出样本中菌株基因组。
随着高通量技术的发展,宏基因组学(metagenomics)已经成为研究微生物群落物种及功能的前沿科学,在肠道微生物、环境微生物等研究领域具有广泛应用。宏基因组学通过对微生物群落全部DNA进行高通量测序,将测序序列与公共数据库进行比对或从头组装出微生物基因组,从而识别微生物群落的物种和功能基因。
目前主流的宏基因组数据分析方法包括三种:基于测序结果进行组装的分析方法;基于reads直接和已知数据库进行比对的方法;基于分箱bin的方法。
常用到的分析工具见下图:


 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?


