一.MHTAN-DTI: Metapath-based hierarchical transformer and attention network for drug–target interaction prediction
基于元路径的层次变换器和注意力网络用于药物-靶标相互作用预测 二区 2023
元路径(Meta-path)是指通过不同类型的节点和边连接的路径。它是一种用于描述网络中节点之间关系的抽象概念。在DTI网络中,药物和靶标通常被表示为节点,而药物-靶标相互作用则表示为边。元路径描述了节点之间的连接模式,可以通过指定节点类型和边类型来定义。举个例子,假设我们有一个DTI网络,其中药物节点用"M"表示,靶标节点用"T"表示,药物-靶标相互作用边用"DT"表示。那么一个元路径可以是"M-DT-T",表示从药物节点到靶标节点的路径。
提出了一种基于元路径层次变换器和注意力网络的DTI预测方法,用于药物-靶标相互作用预测(MHTAN-DTI),应用元路径实例级变换器,单语义注意和多语义注意来生成药物和蛋白质的低维向量表示。元路径实例级转换器对元路径实例执行内部聚合,并对全局上下文信息建模以捕获长程依赖关系。单语义注意力学习某个元路径类型的语义,引入中心节点权重,并为不同的元路径实例分配不同的权重,以获得语义特定的节点嵌入。多语义注意力捕捉不同元路径类型的重要性,并进行加权融合以实现最终的节点嵌入。层次变换器和注意力网络削弱了噪声数据对DTI预测结果的影响,增强了MHTAN-DTI的鲁棒性和泛化能力。
1.模型
该模型首先融合蛋白质进化信息和药物结构信息,然后利用深度卷积神经网络(CNN)挖掘其隐藏特征,最后通过极限学习机(ELM)准确预测相关的DTIs。MSPEDTI首先融合了以位置特异性评分矩阵(PSSM)为特征的蛋白质序列信息和以分子指纹为特征的药物结构信息,然后使用深度学习CNN将它们自动提取为连续的、低维的、信息丰富的特征,从而避免了手动特征的乏味、稀疏、,以及高维度。最后,ELM分类器用于准确确定药物-靶标对是否相关。
1.药物表示
2.蛋白质表示
2.实验
1.数据集
二.Drug-Target Interaction Prediction Based on Transformer
会议 2022.8
1.模型
在表示药物化合物和蛋白质后,将它们转换为两个序列。然后使用 Transformer 架构来提取它们的特征:化合物和蛋白质向量首先分别输入到编码器中以生成它们的表示。之后,将两个生成的特征表示一起输入到解码器中,得到交互向量。鉴于化合物和蛋白质特征向量的顺序对 DTI 预测几乎没有影响(DTI预测模型具有一定的鲁棒性,它们能够在一定程度上适应不同特征的顺序。无论是将化合物特征放在前面还是将蛋白质特征放在前面,模型都能够从特征中提取出有用的信息,并进行准确的预测。),所以删除了变压器架构中的位置编码。修改了解码器的掩码操作,确保解码器可以应用到整个序列。还是六层编码器和解码器,八个注意力头

1.药物表示
SMILES
2.蛋白质表示
蛋白质序列通过预训练方法Word2vec转化为64维向量
2.实验
1.数据集

3.结果
三.MolTrans: Molecular Interaction Transformer for drug–target interaction prediction
二区 2021.5
1.模型
问题:1.整个分子作为输入会引入噪声并且影响预测效果,忽略了引起作用的相关子结构的影响
2.没有使用大量可用的未标记的数据
工作:1.提出了频繁连续子序列(FCS)挖掘的数据驱动方法,该方法适用于提取蛋白质和药物的高质量且尺寸合适的子结构。此外,MolTrans 还包括模拟真实生物DTI过程的生物启发交互模块。新的子结构识别使得能够通过交互模块中的显式映射来理解哪个子结构组合与结果更相关。
2.掘来自多个未标记数据源的数百万药物和蛋白质序列以提取药物和蛋白质的高质量子结构。使用 Transformer 来增强特征,该 Transformer 从未标记数据生成的大量序列子结构输出中捕获复杂信号。

1.利用大量未标记的数据,给定输入的药物和蛋白质数据,FCS挖掘模块首先使用专门的分解算法将其分解为一组明确的子结构序列(FCS首先初始化一个包含独特氨基酸标记或SMILES字符串字符的词汇表集合V。然后,根据这些标记,对整个药物/蛋白质语料库进行分词,得到一个被标记化的集合W。接下来,FCS扫描W,并根据所限定的阈值来识别出最频繁的连续标记(A,B)。然后,FCS将标记化集合W中的每个(A,B)更新为新的标记(AB),并将这个新的标记添加到词汇表集合V中。然后,这个扫描、识别、更新的过程重复进行,直到没有频繁的标记超过阈值θ,或者词汇表集合V的大小达到预定义的最大值ℓ。通过这个操作,频繁的子序列被合并为一个标记,而不够频繁的子序列则被分解为一组较短的标记。最终,对于一个药物/蛋白质,FCS的结果是一个由子结构组成的序列C={C1,…,Ck},其中每个Ci来自于词汇表集合V。)
2.将上一步的输出送入一个embedding模块,利用公式Eip=Econtip+Eposip,Ejd=Econtjd+Eposjd 得到一个可学习的嵌入表,再送入到编码器通过公式2 来获得每个子结构的增强的上下文嵌入E˜d和E˜p(也就是每个子结构不仅包含自身信息,还包括上下文相关信息).
3.交互作用:包括一个由两层组成的交互模块:
① 一个用于对药物子结构和蛋白质子结构相互作用进行建模的相互作用图I。Ii,j=F(Eip~,Ejd~)。F 是衡量对间相互作用的函数,可以是和、平均值、点积(作者使用)等。如果一对子结构确实相互作用,它们将在相互作用图中相应的子结构对位置上具有较高的相互作用分数。
② 一个用于提取邻域相互作用的 CNN 层。因为蛋白质和药物的乡里给亚结构在相互作用方面会相互影响。通过方程O=CNN(I)产生张量O,即输出表征。
4. 然后,解码器模块将张量馈送到分类器,以通过等式P=σ(Wo⋅FLATTEN(O)+bo)(FLATTEN()函数返回一维数组)输出 DTI 概率 P。
1.药物表示
SMILES
2.蛋白质表示
氨基酸序列
2.实验
将数据集中药物和蛋白质子结构的最小出现次数设置为 500,得到 23532 个药物子结构和 16693 个蛋白质子结构。对于 transformer 编码器,我们为药物和蛋白质使用两层 transformer 编码器。输入嵌入的大小为 384,我们为每个中间维度为 1536 的 transformer 编码器设置了 12 个注意头。我们将药物的最大序列长度设置为 50,将蛋白质的最大序列长设置为 545,以覆盖数据集中 95% 的序列。对于CNN,我们使用三个内核大小为三的过滤器。对于优化超参数,我们使用学习率为 0.00001 的Adam优化器,batch_size 设置为64,并允许它运行30个 epochs。dropout 设为0.1。
1.数据集
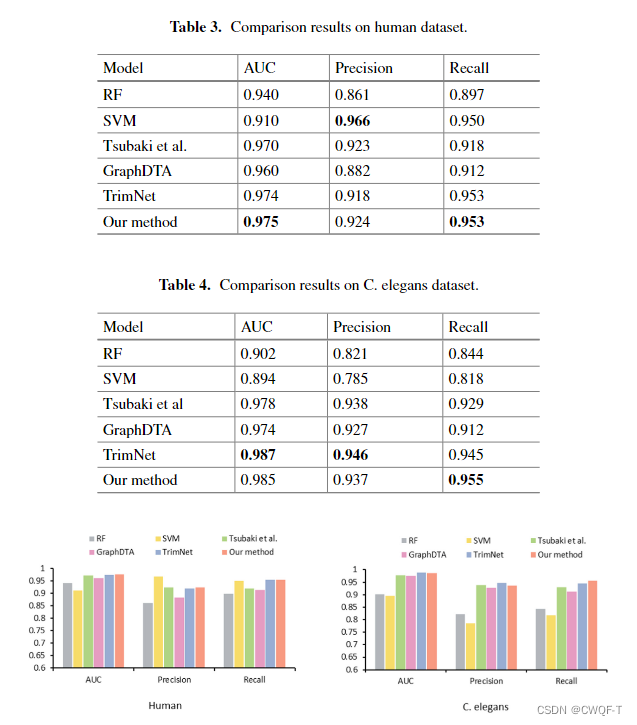
3.结果
 在小数据集上的结果:在 5%、10%、20% 和 30% 的数据集上训练每种方法,并对其余数据集进行预测
在小数据集上的结果:在 5%、10%、20% 和 30% 的数据集上训练每种方法,并对其余数据集进行预测

四.Multi-TransDTI: Transformer for Drug–Target Interaction Prediction Based on Simple Universal Dictionaries with Multi-View Strategy
基于多视角策略的简单通用词典的药物-靶点相互作用预测 2022.05
当前的嵌入方法没有考虑序列中每个字符之间的关系,这通常会导致高维嵌入矩阵并产生部分数据噪声。此外,在关注全局结构的同时,有效获取重要的局部残基仍然具有挑战性。
该模型具有基于简单通用蛋白质和药物字典(SUPD和SUDD)的多视图策略,以更好地嵌入
1.模型

首先,模型使用简单通用蛋白质字典(SUPD)和简单通用药物字典(SUDD)将每个SMILES和氨基酸序列转换为它们的编码标记:所有的DTI数据被表示为集合S = {<p1,d1,0>, <p2,d5,1>, <p4,d3,1>, ……, <pi,di,0>},其中每个三元组是一个正样本或负样本,S的总数量是三元组的总数。通过使用SUPD和SUDD,对于S中的每个药物-靶点对,将对应的pi和di的序列转换为编码标记Vpi和Vdi,最大蛋白质序列的长度被设置为m = 800,最大药物序列的长度被设置为v = 100。改变后的S表示为S = {<Vp1,Vd1,0>, <Vp2,Vd5,1>, <Vp4,Vd3,1>,……, <Vpi,Vdi,0>}。
接着将Vpi同时输入到嵌入层和Transformer模块,而Vdi则送入嵌入层。嵌入层是一个查找表,包含可训练和优化的嵌入向量值,这些值在训练过程中通过损失进行优化。嵌入向量的初始值采用'tensorflow'中的'glorot normal'形式进行初始化。
接下来,得到两个矩阵MVpi和MVdi。然后,通过一维卷积操作在MVpi和MVdi上进行特征提取,以充分提取蛋白质和药物的特征信息。之后,对编码的蛋白质和药物进行全局最大池化操作,以过滤出重要的局部残基。最后,将提取的关键特征进行拼接,用于最终的预测。

SUPD:作者提出的对蛋白质编码嵌入的字典
简单通用蛋白质嵌入字典(Simple Universal Protein Embedding Dictionary,SUPD)是用于编码蛋白质的嵌入字典。在生成SUPD的过程中,蛋白质序列由从A到Z的大写字母组成,共有26个字母。因此,我们生成了18728个可能的编码子序列,计算公式如下:26+26×26+26×26×26=18728。在这里,我们只计算了三阶连续子序列,而不是四阶子序列,原因有两点:首先,四阶子序列会生成大约5万个潜在的蛋白质子序列,这不仅会大幅增加蛋白质字典的大小,还会增加蛋白质的编码复杂性;其次,三阶子序列足以压缩至少一半的蛋白质序列长度,并有利于特征提取。
第二步是从第一步生成的所有二阶和三阶子序列中筛选有价值的子序列。这里不需要筛选一阶子序列,主要是因为在蛋白质序列被二阶和三阶子序列编码后,剩余部分必须是一些单个氨基酸残基,而每个一阶子序列都可以发挥其重要作用。在筛选过程中,我们根据这些子序列在我们的BindingDB数据集的所有蛋白质序列中的频率,主要删除不重要的子序列。只要一个蛋白质序列中的二阶或三阶子序列的数量大于或等于7,我们就将该子序列视为有价值的子序列。最终,我们获得了26个一阶子序列,340个二阶子序列,108个三阶子序列,以及一个长度为474的简单通用蛋白质嵌入字典。据我们所知,这是第一次使用这种方法对蛋白质序列进行编码。通过SUPD,我们不仅可以压缩嵌入矩阵的维度并大大提高效率,还可以全面考虑不同的氨基酸残基。
SUDD:作者提出的对药物进行嵌入的字典
在训练、验证和测试集中计算所有 SMILES 字符串中的单个字符,删除重复的字符并生成唯一的嵌入字典。最终,获得了一个长度为41的独特药物嵌入词典。通过SUDD,将药物SMILES编码到相应的嵌入矩阵中。首先根据SUDD对SMILES中的每个字符进行编码,然后为每个字符生成嵌入向量。每个嵌入的维度是一个变量参数,但实验设置为 10。最后,将这些载体放在一起形成药物的嵌入矩阵。
1.药物表示
SMILES
2.蛋白质表示
氨基酸序列
2.实验
1.数据集
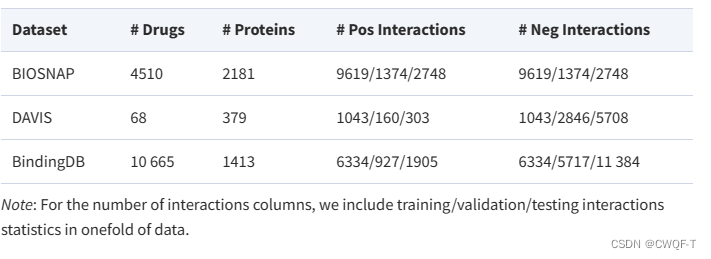
使用Moltrans中的原始BindingDB数据集在删除 6256 个重复样本和四个不可以使用摩根指纹表示药物的正样本之后剩下的6571个正样本和 从19761 个负样本中随机选择的6571个负样本。将所有样本按照 7:1.5:1.5 的比例分为训练集、验证集和测试集。最终,训练集中包括了 9200 个样本,在验证集中包括了 1970 个样本,在测试集中包括了 1972 个样本。每组正负样本的比例为1:1。同时,随机选择了50%和30%的训练集来构建50%和30%的数据集,以便在小数据集上评估我们的模型。所有模型仅由训练集训练;然后,通过验证集确定其中的最优模型。最终,通过将所选模型应用于测试集来获得所有实验结果。


3.结果
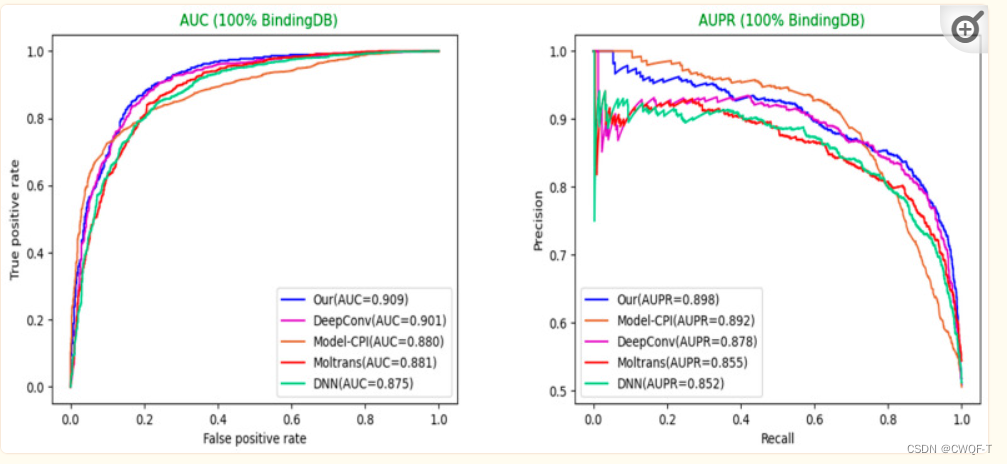

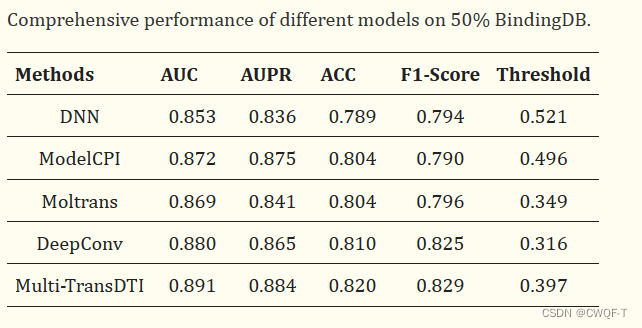
























 611
611

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








