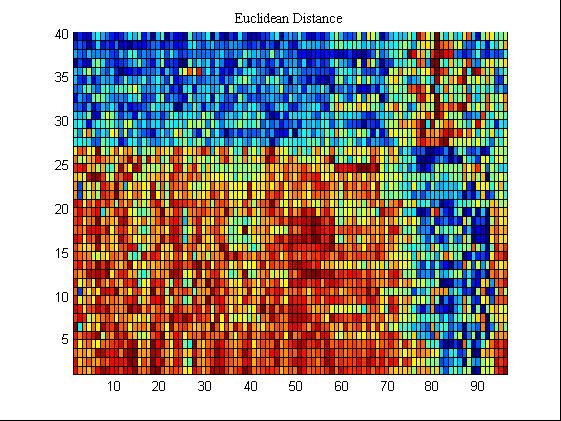
%% 方法一:Euclidean Distance
ED=EuclideanDistance(X,Y);
%
%
%选取40个特征基因
[ED,J]=sort(ED,1,'ascend');%对判决向量进行排序
POS=J(1:40);
disp('方法一:Euclidean Distance
disp(POS');
figure
X1=X(POS,:);
LX1=size(X1,1);
FLAG=zeros(LX1,1);
for i=1:LX1
end
[FLAG,JJ]=sort(FLAG,1,'descend');
pcolor(X1(JJ,:));
title('Euclidean Distance','FontName','Times New Roman','FontSize',10);
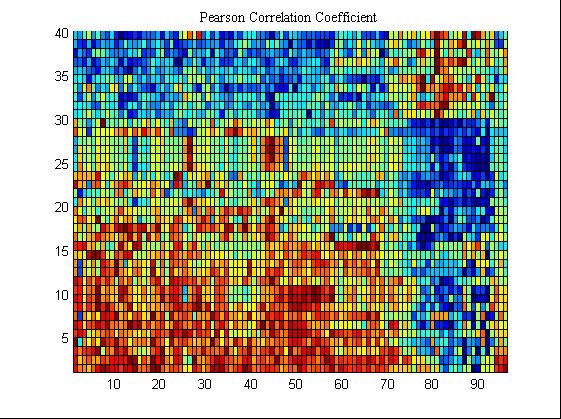
%% 方法二:Pearson's Correlation Coefficient
PR=Pearson(X,Y);
%
%
%选取40个特征基因
[PR,J]=sort(PR,1,'ascend');%对判决向量进行排序
POS=J(1:40);
disp('方法二:Pearson Correlation Coefficient
disp(POS');
figure
X1=X(POS,:);
LX1=size(X1,1);
FLAG=zeros(LX1,1);
for i=1:LX1
end
[FLAG,JJ]=sort(FLAG,1,'descend');
pcolor(X1(JJ,:));
title('Pearson Correlation Coefficient','FontName','Times New Roman','FontSize',10);
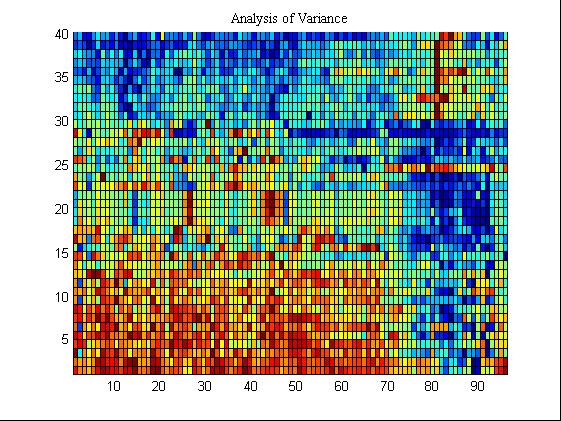
%% 方法三:Analysis of Variance
FR=VarianceAnalysis(X,Y);
%
%
%选取40个特征基因
[FR,J]=sort(FR,1,'descend');%对判决向量进行排序
POS=J(1:40);
disp('方法三:Analysis of Variance
disp(POS');
figure
X1=X(POS,:);
LX1=size(X1,1);
FLAG=zeros(LX1,1);
for i=1:LX1
end
[FLAG,JJ]=sort(FLAG,1,'descend');
pcolor(X1(JJ,:));
title('Analysis of Variance','FontName','Times New Roman','FontSize',10);
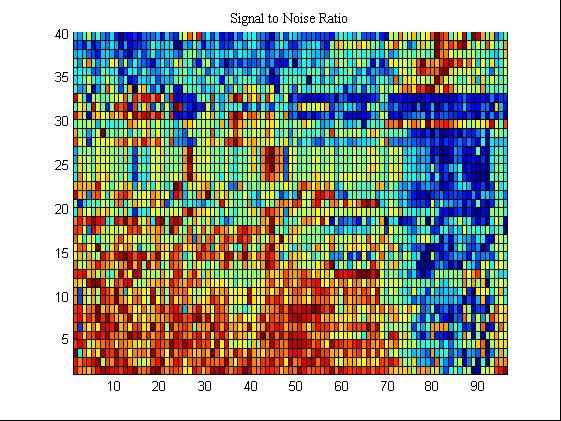
%% 方法四:Signal to Noise Ratio
SNR=SignalNoiseRatio(X,Y);
%
%
%选取40个特征基因
[SNR,J]=sort(SNR,1,'descend');%对判决向量进行排序
POS=J(1:40);
disp('方法四:Signal to Noise Ratio
disp(POS');
figure
X1=X(POS,:);
LX1=size(X1,1);
FLAG=zeros(LX1,1);
for i=1:LX1
end
[FLAG,JJ]=sort(FLAG,1,'descend');
pcolor(X1(JJ,:));
title('Signal to Noise Ratio','FontName','Times New Roman','FontSize',10);
%% 方法五:t-test
TV=Ttest(X,Y);
%
%
%选取40个特征基因
[TV,J]=sort(TV,1,'descend');%对判决向量进行排序
POS=J(1:40);
disp('方法五:t-test
disp(POS');
figure
X1=X(POS,:);
LX1=size(X1,1);
FLAG=zeros(LX1,1);
for i=1:LX1
end
[FLAG,JJ]=sort(FLAG,1,'descend');
pcolor(X1(JJ,:));
title('t-test','FontName','Times New Roman','FontSize',10);
%% 方法六:Correlation-based Selection
CR=Correlation(X,Y);
%
%
%选取40个特征基因
[CR,J]=sort(CR,1,'descend');%对判决向量进行排序
POS=J(1:40);
disp('方法六:Correlation-based Selection
disp(POS');
figure
X1=X(POS,:);
LX1=size(X1,1);
FLAG=zeros(LX1,1);
for i=1:LX1
end
[FLAG,JJ]=sort(FLAG,1,'descend');
pcolor(X1(JJ,:));
title('Correlation-based Selection','FontName','Times New Roman','FontSize',10);
转载自http://blog.sina.com.cn/s/blog_4b425443010190o3.html





 本文介绍了六种用于从基因表达谱数据中识别肿瘤相关基因的算法,包括欧氏距离、皮尔逊相关系数等方法,并展示了每种算法选择的前40个特征基因。
本文介绍了六种用于从基因表达谱数据中识别肿瘤相关基因的算法,包括欧氏距离、皮尔逊相关系数等方法,并展示了每种算法选择的前40个特征基因。
















 1238
1238

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








