在机器学习和数据挖掘中,我们经常需要对数据的大量特征进行降维处理,减少训练的运算量和运算时间,而主成分分析(PCA)就是用来进行降维操作的算法。
主成分分析(PCA)的描述如下:
PCA(Principal Component Analysis),即主成分分析方法,是一种使用最广泛的数据降维算法。PCA的主要思想是将n维特征映射到k维上,这k维是全新的正交特征也被称为主成分,是在原有n维特征的基础上重新构造出来的k维特征。PCA的工作就是从原始的空间中顺序地找一组相互正交的坐标轴,新的坐标轴的选择与数据本身是密切相关的。其中,第一个新坐标轴选择是原始数据中方差最大的方向,第二个新坐标轴选取是与第一个坐标轴正交的平面中使得方差最大的,第三个轴是与第1,2个轴正交的平面中方差最大的。依次类推,可以得到n个这样的坐标轴。通过这种方式获得的新的坐标轴,我们发现,大部分方差都包含在前面k个坐标轴中,后面的坐标轴所含的方差几乎为0。于是,我们可以忽略余下的坐标轴,只保留前面k个含有绝大部分方差的坐标轴。事实上,这相当于只保留包含绝大部分方差的维度特征,而忽略包含方差几乎为0的特征维度,实现对数据特征的降维处理。
我们选用基于SVD分解协方差矩阵实现PCA算法来实现
由于算法实现过程中需要进行实对称矩阵的特征向量分解,所以我先用java实现基于雅克比(Jacobi)方法的特征向量分解。
实现如下:
定义特征向量分解结果类
package com.algorithm;
import org.ejml.data.DenseMatrix64F;
public class EDInfo {
private DenseMatrix64F values;
private double[] vectors;
public EDInfo(DenseMatrix64F values,double[] vectors) {
this.values = values;
this.vectors = vectors;
}
public DenseMatrix64F getValues() {
return values;
}
public void setValues(DenseMatrix64F values) {
this.values = values;
}
public double[] getVectors() {
return vectors;
}
public void setVectors(double[] vectors) {
this.vectors = vectors;
}
}
然后是特征值倒序方法
public static int[] argsort(double[] input) {
int[] rs = new int[input.length];
for(int i=0;i<input.length;i++){
rs[i] = i;
}
for(int i=0;i<input.length-1;i++) {
for(int j=i+1;j<input.length;j++) {
if(input[i] < input[j]) {
double tmp = input[i];
int tmpIndex = rs[j];
input[i] = input[j];
input[j] = tmp;
rs[j] = rs[i];
rs[i] = tmpIndex;
}
}
}
return rs;
}
然后便是雅克比特征分解的主方法
public static EDInfo JacobiCount(DenseMatrix64F src,double diff,int iter) {
DenseMatrix64F values = new DenseMatrix64F(src.numRows,src.numCols);
for(int i=0;i<src.numRows;i++) {
for(int j=0;j<src.numCols;j++) {
if(i == j) {
values.set(i, j, 1);
}else {
values.set(i, j, 0);
}
}
}
int nCount = 0;
while(true)
{
double dbMax = Double.MIN_VALUE;
int nRow = 0;
int nCol = 1;
for(int i=0;i<src.numRows;i++) {
for(int j=0;j<src.numCols;j++) {
if(i != j && Math.abs(src.get(i, j)) > dbMax) {
dbMax = Math.abs(src.get(i, j));
nRow = i;
nCol = j;
}
}
}
if(dbMax < diff)
break;
if(nCount > iter)
break;
nCount++;
double dbApp = src.get(nRow, nRow);
double dbApq = src.get(nRow, nCol);
double dbAqq = src.get(nCol, nCol);
double dbAngle = 0.5*Math.atan2(-2*dbApq,dbAqq-dbApp);
double dbSinTheta = Math.sin(dbAngle);
double dbCosTheta = Math.cos(dbAngle);
double dbSin2Theta = Math.sin(2*dbAngle);
double dbCos2Theta = Math.cos(2*dbAngle);
src.set(nRow, nRow, dbApp*dbCosTheta*dbCosTheta +
dbAqq*dbSinTheta*dbSinTheta + 2*dbApq*dbCosTheta*dbSinTheta);
src.set(nCol, nCol, dbApp*dbSinTheta*dbSinTheta +
dbAqq*dbCosTheta*dbCosTheta - 2*dbApq*dbCosTheta*dbSinTheta);
src.set(nRow, nCol, 0.5*(dbAqq-dbApp)*dbSin2Theta + dbApq*dbCos2Theta);
src.set(nCol, nRow,src.get(nRow, nCol));
for(int i = 0; i < src.numRows; i ++)
{
if((i!=nCol) && (i!=nRow))
{
dbMax = src.get(i, nRow);
src.set(i, nRow,src.get(i, nCol)*dbSinTheta+dbMax*dbCosTheta);
src.set(i, nCol,src.get(i, nCol)*dbCosTheta-dbMax*dbSinTheta);
}
}
for (int j = 0; j < src.numRows; j ++)
{
if((j!=nCol) && (j!=nRow))
{
dbMax = src.get(nRow, j);
src.set(nRow, j,src.get(nCol, j)*dbSinTheta+dbMax*dbCosTheta);
src.set(nCol, j,src.get(nCol, j)*dbCosTheta-dbMax*dbSinTheta);
}
}
for(int i = 0; i < src.numRows; i ++)
{
dbMax = values.get(i,nRow);
values.set(i, nRow,values.get(i,nCol)*dbSinTheta+dbMax*dbCosTheta);
values.set(i,nCol,values.get(i,nCol)*dbCosTheta-dbMax*dbSinTheta);
}
}
double[] eig = new double[src.numRows];
for(int i=0;i<src.numRows;i++) {
eig[i] = src.get(i, i);
}
int[] sortInx = argsort(eig);
DenseMatrix64F tmpValues = new DenseMatrix64F(src.numRows,src.numCols);
for(int i=0;i<src.numRows;i++) {
for(int j=0;j<src.numRows;j++) {
tmpValues.set(i, j, values.get(j,sortInx[i]));
}
eig[i] = src.get(sortInx[i],sortInx[i]);
}
for(int i = 0; i < src.numRows; i ++)
{
double dSumVec = 0;
for(int j = 0; j < src.numRows; j ++)
dSumVec += tmpValues.get(j, i);
if(dSumVec<0)
{
for(int j = 0;j < src.numRows; j ++)
tmpValues.set(j, i,tmpValues.get(j, i)*-1);
}
}
return new EDInfo(tmpValues,eig);
}
接下来便是pca的主方法
public static DenseMatrix64F runPCA(DenseMatrix64F src,int k) {
DenseMatrix64F rs = new DenseMatrix64F(src.numRows,k);
//计算输入矩阵每个元素和特征值平均的差值矩阵
DenseMatrix64F norm_X = new DenseMatrix64F(src.numRows,src.numCols);
for(int i =0;i<src.numCols;i++) {
double tmp=0;
for(int j=0;j<src.numRows;j++) {
tmp+=src.get(j, i);
}
tmp /=src.numRows;
for(int j=0;j<src.numRows;j++) {
norm_X.set(j,i, src.get(j, i)-tmp);
}
}
//计算协方差矩阵
DenseMatrix64F norm_X_T = new DenseMatrix64F(src.numCols,src.numRows);
CommonOps.transpose(norm_X, norm_X_T);
DenseMatrix64F scatter_matrix = new DenseMatrix64F(src.numCols,src.numCols);
CommonOps.mult(norm_X_T,norm_X,scatter_matrix);
//特征向量分解
EDInfo ed = Jacobi.JacobiCount(new DenseMatrix64F(scatter_matrix),0.001,1000);
//选取前k个特征
DenseMatrix64F feature = new DenseMatrix64F(k,src.numCols);
for(int i=0;i<k;i++) {
for(int j=0;j<src.numCols;j++) {
feature.set(i, j, ed.getValues().get(j, i));
}
}
DenseMatrix64F feature_T = new DenseMatrix64F(src.numCols,k);
CommonOps.transpose(feature, feature_T);
CommonOps.mult(norm_X,feature_T,rs);
return rs;
}
现在我们来测试一下
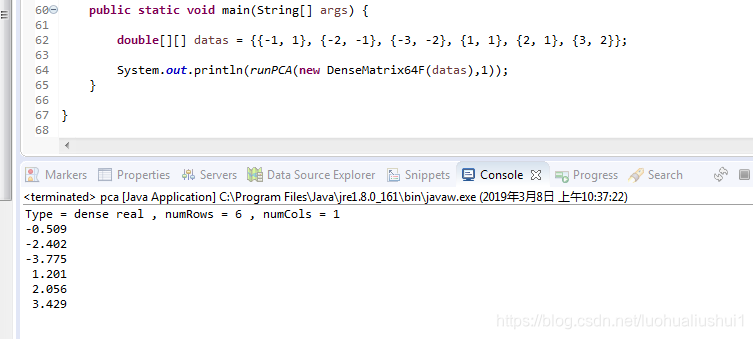
double[][] datas = {{-1, 1}, {-2, -1}, {-3, -2}, {1, 1}, {2, 1}, {3, 2}};
System.out.println(runPCA(new DenseMatrix64F(datas),1));
结果如下:








 本文深入探讨了主成分分析(PCA)算法,一种广泛应用于数据降维的技术。PCA通过重构数据集的特征空间,减少冗余信息,提高计算效率。文章详细介绍了PCA的工作原理,包括如何寻找数据的主成分,并通过实例演示了基于SVD分解协方差矩阵实现PCA的过程。
本文深入探讨了主成分分析(PCA)算法,一种广泛应用于数据降维的技术。PCA通过重构数据集的特征空间,减少冗余信息,提高计算效率。文章详细介绍了PCA的工作原理,包括如何寻找数据的主成分,并通过实例演示了基于SVD分解协方差矩阵实现PCA的过程。

















 1301
1301

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










