今天在实验钙调素蛋白的动力学模拟时遇到一个问题:在pdb文件中修改钙离子后,用VMD打开后,钙离子位置发生变化?
在pdb中钙离子的残基名为CA,然而在力场gromos54a7文件夹下rtp中钙离子残基名为CA2+,所以要做把CA替换为CA2+
PDBID:2BBM
# 2BBM 未修改
ATOM 2700 2HD2 LEU B 26 17.951 -18.980 -17.260 1.00 7.95 H
ATOM 2701 3HD2 LEU B 26 17.818 -20.520 -16.416 1.00 7.62 H
TER 2702 LEU B 26
HETATM 2703 CA CA A 181 11.240 1.243 10.109 1.00 1.14 CA2+
HETATM 2704 CA CA A 182 19.123 4.066 0.604 1.00 0.99 CA2+
HETATM 2705 CA CA A 183 -15.000 -10.457 -0.757 1.00 0.94 CA2+
HETATM 2706 CA CA A 184 -21.362 0.710 -5.410 1.00 0.93 CA2+
TER 2707 CA A 184
# 2BBM-1.pdb 修改1
ATOM 2700 2HD2 LEU B 26 17.951 -18.980 -17.260 1.00 7.95 H
ATOM 2701 3HD2 LEU B 26 17.818 -20.520 -16.416 1.00 7.62 H
TER 2702 LEU B 26
HETATM 2703 CA CA2+ A 181 11.240 1.243 10.109 1.00 1.14 CA2+
HETATM 2704 CA CA2+ A 182 19.123 4.066 0.604 1.00 0.99 CA2+
HETATM 2705 CA CA2+ A 183 -15.000 -10.457 -0.757 1.00 0.94 CA2+
HETATM 2706 CA CA2+ A 184 -21.362 0.710 -5.410 1.00 0.93 CA2+
TER 2707 CA A 184
# 2BBM-2.pdb 修改2
ATOM 2700 2HD2 LEU B 26 17.951 -18.980 -17.260 1.00 7.95 H
ATOM 2701 3HD2 LEU B 26 17.818 -20.520 -16.416 1.00 7.62 H
TER 2702 LEU B 26
HETATM 2703 CA CA2+A 181 11.240 1.243 10.109 1.00 1.14 CA2+
HETATM 2704 CA CA2+A 182 19.123 4.066 0.604 1.00 0.99 CA2+
HETATM 2705 CA CA2+A 183 -15.000 -10.457 -0.757 1.00 0.94 CA2+
HETATM 2706 CA CA2+A 184 -21.362 0.710 -5.410 1.00 0.93 CA2+
TER 2707 CA A 184
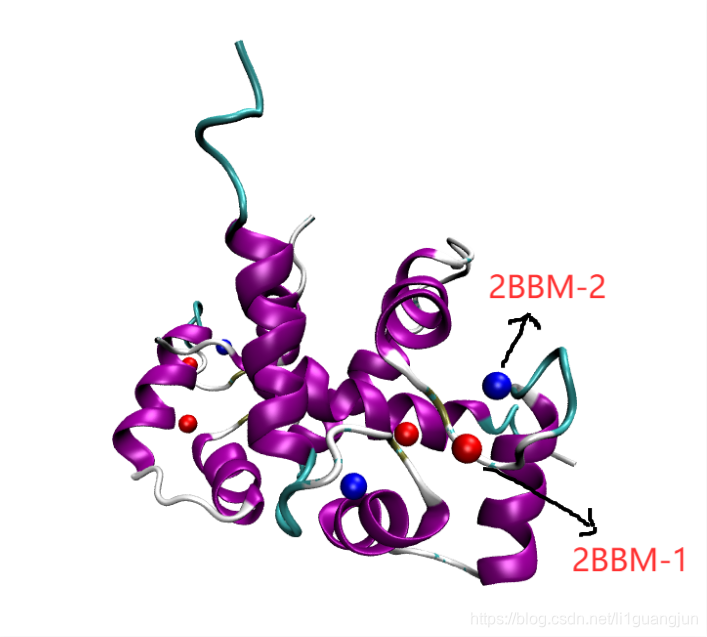
钙离子以球状表示,蓝色表示2BBM-2.pdb,红色表示2BBM-1.pdb,经过比对,2BBM-2.pdb与2BBM.pdb完全重合,钙离子位置没有偏差。
2BBM-1.pdb和2BBM-2.pdb相比:A链和B链没有对齐
又测试了3CLN,修改pdb文件如下所示:
# 3CLN 未修改
ATOM 1125 O ALA A 147 -1.938 22.966 5.190 1.00 63.20 O
ATOM 1126 CB ALA A 147 -1.738 26.270 6.293 1.00 63.07 C
TER 1127 ALA A 147
HETATM 1128 CA CA A 149 -13.638 53.352 31.500 1.00 12.29 CA2+
HETATM 1129 CA CA A 150 -17.797 50.156 20.859 1.00 15.64 CA2+
HETATM 1130 CA CA A 151 15.035 17.711 17.954 1.00 28.17 CA2+
HETATM 1131 CA CA A 152 13.784 23.887 8.345 1.00 29.20 CA2+
TER 1132 CA A 152
# 3CLN-1 修改1
ATOM 1125 O ALA A 147 -1.938 22.966 5.190 1.00 63.20 O
ATOM 1126 CB ALA A 147 -1.738 26.270 6.293 1.00 63.07 C
TER 1127 ALA A 147
HETATM 1128 CA CA2+ A 149 -13.638 53.352 31.500 1.00 12.29 CA
HETATM 1129 CA CA2+ A 150 -17.797 50.156 20.859 1.00 15.64 CA
HETATM 1130 CA CA2+ A 151 15.035 17.711 17.954 1.00 28.17 CA
HETATM 1131 CA CA2+ A 152 13.784 23.887 8.345 1.00 29.20 CA
TER 1132 CA A 152
# 3CLN-2 修改2
ATOM 1125 O ALA A 147 -1.938 22.966 5.190 1.00 63.20 O
ATOM 1126 CB ALA A 147 -1.738 26.270 6.293 1.00 63.07 C
HETATM 1127 CA CA2+A 149 -13.638 53.352 31.500 1.00 12.29 CA2+
HETATM 1128 CA CA2+A 150 -17.797 50.156 20.859 1.00 15.64 CA2+
HETATM 1129 CA CA2+A 151 15.035 17.711 17.954 1.00 28.17 CA2+
HETATM 1130 CA CA2+A 152 13.784 23.887 8.345 1.00 29.20 CA2+
TER 1131 CA A 152
得到的结果:
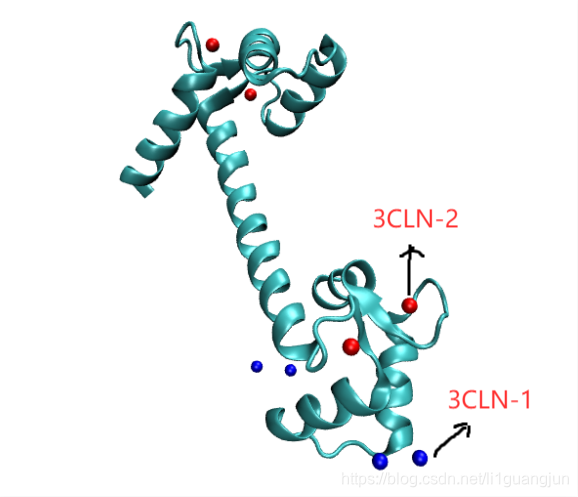
蛋白残基链和杂原子链在pdb文件中要对齐,才不会出现钙离子位置发生偏差。在做模拟时,要比对原始文件和转换文件中结构的差别,别一开始就错了。





 本文探讨了在进行钙调素蛋白动力学模拟时遇到的问题,即修改pdb文件中钙离子名称后,在VMD中显示的位置发生变化。通过对比不同pdb文件发现,确保蛋白残基链与杂原子链对齐是关键。
本文探讨了在进行钙调素蛋白动力学模拟时遇到的问题,即修改pdb文件中钙离子名称后,在VMD中显示的位置发生变化。通过对比不同pdb文件发现,确保蛋白残基链与杂原子链对齐是关键。
















 1569
1569

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








