对于读者的日常学习知识和习惯来说,gongzhonghao是更为高效的平台。
scanning below, come on!

https://gitee.com/jaychouwong/gongzhonghao/blob/master/关注公众号请扫码.png
大概是因为KEGG数据库更新了,因此clusterProfiler也要使用最新版本的(4.6.2)
对于更新包呢,试了一些方法,未果,因此直接去Bioconductor - clusterProfiler下载打包好的
选择的是source包:
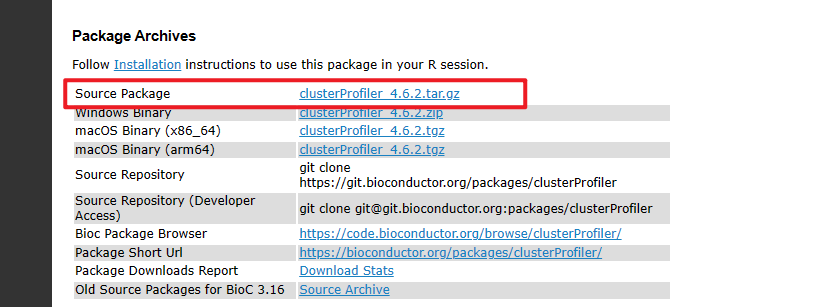
使用 R CMD INSTALL clusterProfiler_4.6.2.tar.gz进行安装
当然,会提示你依赖的一些包也需要更新,于是乎用同样的方法安装了 DOSE_3.24.2、HDO.db_0.99.1、GOSemSim_2.24.0。
其中GOSemSim_2.24.0用source包还报错了。。。于是乎选择了windows binary包直接解压到R library(.libPaths())里即可:
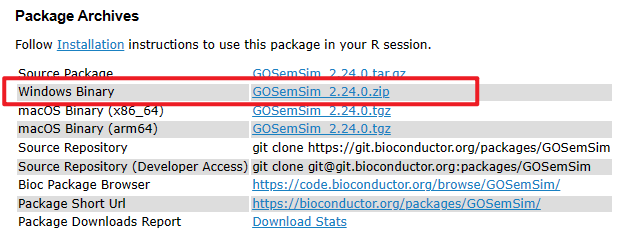
或许以上包都可以用这种binary包直接安装
安装好依赖包后,再安装clusterProfiler就能成功了,enrichKEGG也是成功运行了。。。
总之就是一个大无语





















 1796
1796

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








