R包介绍
为什么开发这个包
-
你在下载和分析GEO数据库或者使用GEOquery包时是否遇到以下问题?
- getGEO总是下载文件失败
- 网页中series文件或者补充文件下载总是失败
- 无法确定表达矩阵是否已经log化
- 不会进行探针ID到基因SYBBOL的注释转换
- GPL注释文件下载总是中断
- GPL注释文件不包含基因SYMBOL
- GPL注释文件的SYMBOL存在一对多和多对一的情况
- GPL注释文件仅能查看但没有下载按钮
- 下载的表达谱基因包含日期
-
如果有以上这些问题, 那么我开发的R包fastGEO大概率能解决你的问题!
实现的功能
- 一行代码实现对芯片表达谱数据的下载、标准化、样本间矫正、探针注释、去除重复基因、恢复日期基因等等操作
- 最终你会直接得到行名是基因列名是样本的标准化之后的表达谱, 以及对应的样本临床信息
- 探针注释是fastGRO开发的主要目的, 可以实现对多种情况的GPL平台进行下载和处理
- 对于文件下载失败的问题提供多种解决办法
适用场景
- 首先, GEO网站在国外, 下载网速受限制是难以避免的, 会科学上网会更好一点, 但偶尔还是下载失败, 需要多尝试
- fastGEO下载表达谱是针对于芯片数据, 不支持高通量数据, 因为NGS数据不存放在series里, 而是在补充文件里
- 探针注释能够成功的前提是GPL平台要提供了注释文件, 并且文件包含基因ID信息
- SYMBOL/ENSEMBL/ENTREZ/REFSEQ等等都可以
- 如果没有提供基因信息, 那也不能无中生有
- 对于任何测序类型, 都支持一行代码获取样本临床信息
功能演示
- 安装在下一节介绍
# detach("package:fastGEO", unload = TRUE)
# 加载R包
library(fastGEO)
========================================
Welcome to fastGEO (v1.6.1)
- 作者: 生信摆渡
- 微信: bioinfobaidu1
- 帮助文档: help(package = fastGEO)
========================================
# 查看帮助文档, 在RStudio里可以点击查看每一个函数的帮助文档
help(package = fastGEO)
Documentation for package 'fastGEO'
Information on package 'fastGEO'
Description:
Package: fastGEO
Title: Quickly download and annotate GEO data chip
datasets
Version: 1.6.1
Authors@R: person("Jiahao", "Wang", , "wjhyyds@sina.cn", role
= c("aut", "cre"), comment = c(ORCID =
"YOUR-ORCID-ID"))
Description: You can use the fastqGEO to easily download GEO
chip expression profiles and clinical data and
automate probe-gene annotation.
License: `use_mit_license()`, `use_gpl3_license()` or
friends to pick a license
Encoding: UTF-8
Roxygen: list(markdown = TRUE)
RoxygenNote: 7.3.2
Depends: GEOquery (>= 2.72.0), limma (>= 3.60.4), AnnoProbe
(>= 0.1.0), stringr (>= 1.5.1), httr (>= 1.4.7),
fastR (>= 1.3.0), rvest (>= 1.0.4), R (>= 2.10)
Imports: data.table, readr
Suggests: org.Hs.eg.db, org.Mm.eg.db, clusterProfiler
LazyData: true
NeedsCompilation: no
Packaged: 2025-07-09 14:09:48 UTC; Jia
Author: Jiahao Wang [aut, cre] (YOUR-ORCID-ID)
Maintainer: Jiahao Wang <wjhyyds@sina.cn>
RemoteType: local
RemoteUrl: W:\02_Study\R_build\build\fastGEO\version\1.6.1\fastGEO_1.6.1.tar.gz
Built: R 4.4.1; ; 2025-07-09 14:09:49 UTC; windows
Index:
anno_GEO Annotate GEO Expression Matrix
anno_GEO_AnnoProbe Annotate GEO Data Using AnnoProbe Package
anno_GEO_online Annotate GEO Data Using Online Annotation
anno_NGS Annotate NGS Expression Matrix
anno_obj GEO Platform Annotation Reference Object
data_date_to_gene Convert table for date to gene convertion
date_to_gene Convert Date-Based IDs to Gene Symbols
download_GEO Download and Process GEO Dataset
download_GEO_file Download GEO Data Files
extract_GPL_anno Extract Gene Symbol Annotations from GEO
Platform (GPL) Data
getGEO2 Download and Process GEO Dataset
get_GEO_pheno Get GEO Phenotype Data
read_GPL Read GEO Platform (GPL) File
read_GPL_file Read and Process GPL Annotation File
read_GPL_url Download and Process GPL Annotation from GEO
Website
run_fastGEO_app 启动 fastGEO 的 Shiny 应用
- 每一个函数的帮助文档可以使用
?函数名进行自行查看, 比如?download_GEO
download_GEO
- 主函数, 输入GSE号, 实现自动化下载和处理数据
# 查看参数
args(download_GEO)
function (GSE, out_dir = "00_GEO_data", anno_file = NULL, method = "max",
timeout = 3600 * 24, download_method = "curl", symbol_name = NULL)
NULL
- GSE: GSE号
- out_dir: 文件保存目录, 默认为当前目录的00_GEO_data文件夹, 可自行修改
- anno_file: 注释文件, 现在已经内置了注释文件, 可以不用指定
- method: 重复基因的处理方法
- max: 默认, 取最大表达量做为该基因的表达水平, 相对较快且合理
- mean: 取均值, 太慢
- remove: 直接去除重复基因, 最快, 但不推荐
- sum: 求和, 用的比较少, 暂不推荐
- download_method: 下载文件的方式, 如果存在本地curl则用本地的, 不存在则使用getGEO的curl
- symbol_name: 注释文件中含有基因信息的那一列的列名
# 这种下载文件的方式可能也比较慢, 看运气
# 如果下载失败, 看下面的download_GEO_file函数
obj = download_GEO("GSE294904", out_dir = "test/00_GEO_data_GSE294904")
INFO [2025-07-09 23:06:31] Step1: Download GEO data ...
INFO [2025-07-09 23:06:31] Querying dataset: GSE294904 ...
- Use local curl
- Found 1 GPL
- Found 6 samples, 34 metas.
- Writting sample_anno to test/00_GEO_data_GSE294904/GSE294904_sample_anno.csv
- Execute log(expM + 1) ...
- Normalize between arrays ...
- Successed, file save to test/00_GEO_data_GSE294904/GSE294904_GPL570.RData.
INFO [2025-07-09 23:06:37] Step2: Annotate probe GPL570 ...
INFO [2025-07-09 23:06:37] Use built-in annotation file ...
- All porbes matched!
- All porbes annotated!
INFO [2025-07-09 23:06:37] Removing duplicated genes by method: max ...
INFO [2025-07-09 23:06:39] Done.
# 结果包含以下文件
cbind(dir("test/00_GEO_data_GSE294904/"))
A matrix: 7 × 1 of type chr
GSE294904.RData
GSE294904_GPL570.RData
GSE294904_GPL570_expM.csv
GSE294904_GPL570_Normalization.pdf
GSE294904_GPL570_sample_anno.csv
GSE294904_GPL570_sample_anno.xlsx
GSE294904_series_matrix.txt.gz
GSE294904_GPL570.RData是最终保存的文件, 也是返回的obj的内容, 可以load进行使用sample_anno是样本临床信息, 可打开快速浏览和筛选样本
# 保存的对象
names(obj)
[1] "GPL" "expM" "expM_raw" "sample_anno"
obj$GPL
[1] "GPL570"
# 完美的表达谱
hd(obj$expM)
dim: 22881 × 6
GSM8928113 GSM8928114 GSM8928115 GSM8928116 GSM8928117
A1BG 7.664415 7.834532 8.045193 8.715155 8.702467
A1BG-AS1 4.182379 6.486369 3.594159 4.888389 5.456329
A1CF 3.311157 6.515058 6.397253 7.288225 6.671245
A2M 5.580024 6.934858 5.895105 6.041618 6.338498
A2M-AS1 2.398807 5.313860 5.330637 5.697162 5.843399
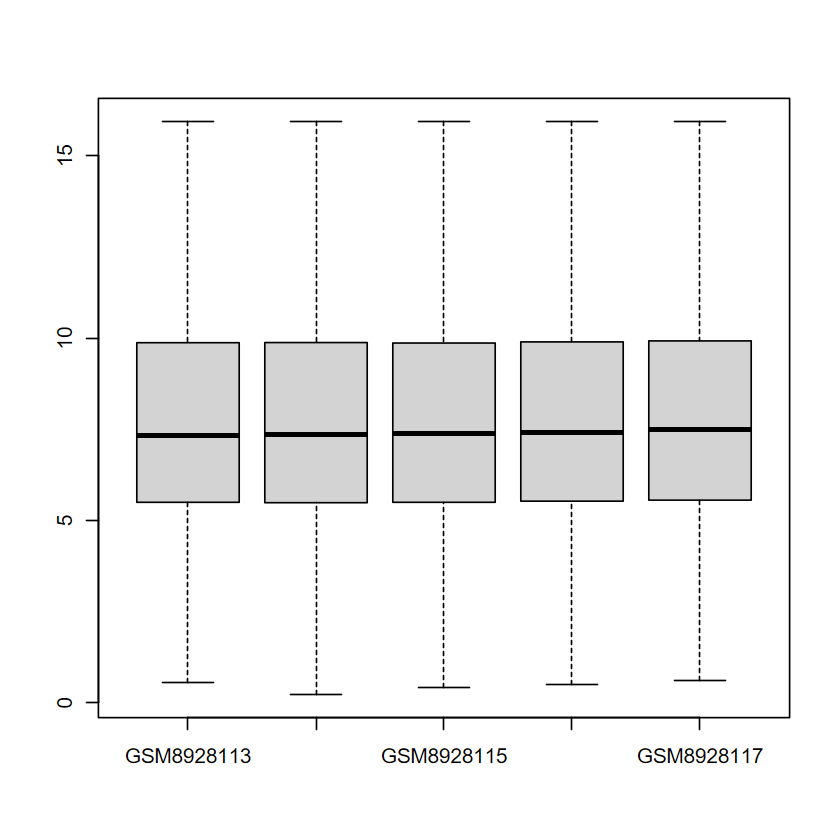
boxplot(obj$expM[, 1:5])
# 样本信息
hd(obj$sample_anno)
dim: 6 × 34
title geo_accession status submission_date last_update_date
GSM8928113 NCI-ADR-RES control GSM8928113 Public on Apr 30 2025 Apr 17 2025 Apr 30 2025
GSM8928114 NCI-ADR-RES treated GSM8928114 Public on Apr 30 2025 Apr 17 2025 Apr 30 2025
GSM8928115 GCS control GSM8928115 Public on Apr 30 2025 Apr 17 2025 Apr 30 2025
GSM8928116 GCS treated GSM8928116 Public on Apr 30 2025 Apr 17 2025 Apr 30 2025
GSM8928117 asGCS control GSM8928117 Public on Apr 30 2025 Apr 17 2025 Apr 30 2025
download_GEO_file
- 专门用来下载文件的
- 如果download_GEO下载series文件失败, 可先尝试这种方式, 然后再运行download_GEO
- 还可以用来下载所有的补充文件或其中一个
# 如果download_GEO下载文件失败, 可下面这样专门下载series文件
download_GEO_file("GSE294904", out_dir = "test/00_GEO_data_GSE294904/")
All file already downloaded in test/00_GEO_data_GSE294904/
# 显示已经下载好了, 因为上面的download_GEO我下载成功了, 嘿嘿
# 我们可以换个输出目录再演示一下
download_GEO_file("GSE294904", out_dir = "test/00_GEO_data_GSE294904-2/")
Will download 1 file(s) ...
Succeed.
# 结果包含以下文件
cbind(dir("test/00_GEO_data_GSE294904-2/"))
[,1]
[1,] "GSE294904_series_matrix.txt.gz"
- 有时候还是比较慢的, 很离谱, 不过直接在浏览器下载却出奇的快
- 所以也支持跳转到浏览器进行下载, 逆天!
- 设置 method = “browser” 就行了
- 同时也可以设置输出目录, 程序会自动粘贴输出目录, 然后在浏览器里直接复制这个目录, 进行保存文件
- 所以浏览器的文件下载要设置每次询问下载位置
download_GEO_file("GSE294904", out_dir = "test/00_GEO_data_GSE294904-3/", method = "browser")
Found 1 GPL(s):
- GSE294904_series_matrix.txt.gz
Will be download 'GSE294904_series_matrix.txt.gz' in browser after 3s ...
# 很快啊, 几秒就完成了
cbind(dir("test/00_GEO_data_GSE294904-3/"))
[,1]
[1,] "GSE294904_RAW.tar"
[2,] "GSE294904_series_matrix.txt.gz"
# 也可以用来下载补充文件
download_GEO_file(GSE = "GSE294904",
file = "GSE294904_RAW.tar",
out_dir = "test/00_GEO_data_GSE294904-3/",
method = "browser")
Found 1 GPL(s):
- GSE294904_RAW.tar
Will be download 'GSE294904_RAW.tar' in browser after 3s ...
get_GEO_pheno
- 快速获取样本临床信息
# 芯片数据
sample_anno = get_GEO_pheno("GSE294904", out_dir = "test/00_GEO_data_GSE294904")
INFO [2025-07-09 23:42:44] Querying dataset: GSE294904 ...
- Use local curl
- Found 1 GPL
- Found 6 samples, 34 metas.
- Writting sample_anno to test/00_GEO_data_GSE294904/GSE294904_sample_anno.csv
- Execute log(expM + 1) ...
- Normalize between arrays ...
- Successed, file save to test/00_GEO_data_GSE294904/GSE294904_GPL570.RData.
hd(sample_anno)
dim: 6 × 34
title geo_accession status submission_date last_update_date
GSM8928113 NCI-ADR-RES control GSM8928113 Public on Apr 30 2025 Apr 17 2025 Apr 30 2025
GSM8928114 NCI-ADR-RES treated GSM8928114 Public on Apr 30 2025 Apr 17 2025 Apr 30 2025
GSM8928115 GCS control GSM8928115 Public on Apr 30 2025 Apr 17 2025 Apr 30 2025
GSM8928116 GCS treated GSM8928116 Public on Apr 30 2025 Apr 17 2025 Apr 30 2025
GSM8928117 asGCS control GSM8928117 Public on Apr 30 2025 Apr 17 2025 Apr 30 2025
# 其他测序类型也支持
sample_anno = get_GEO_pheno("GSE121950", out_dir = "test/00_GEO_data_GSE121950")
INFO [2025-07-09 23:43:10] Querying dataset: GSE121950 ...
- Use local curl
- Found 1 GPL
- Found 6 samples, 41 metas.
- Writting sample_anno to test/00_GEO_data_GSE121950/GSE121950_sample_anno.csv
- Can't found expression profile, please see
https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE121950
- Return sample annotation!
hd(sample_anno)
dim: 6 × 41
title geo_accession status submission_date
GSM3450401 villus_induced abortion_rep1 GSM3450401 Public on Nov 27 2018 Oct 29 2018
GSM3450402 villus_induced abortion_rep2 GSM3450402 Public on Nov 27 2018 Oct 29 2018
GSM3450403 villus_induced abortion_rep3 GSM3450403 Public on Nov 27 2018 Oct 29 2018
GSM3450404 villus_recurrent spontaneous abortion_rep1 GSM3450404 Public on Nov 27 2018 Oct 29 2018
GSM3450405 villus_recurrent spontaneous abortion_rep2 GSM3450405 Public on Nov 27 2018 Oct 29 2018
last_update_date
GSM3450401 Nov 27 2018
GSM3450402 Nov 27 2018
GSM3450403 Nov 27 2018
GSM3450404 Nov 27 2018
GSM3450405 Nov 27 2018
getGEO2
-
用于下载和预处理数据的函数
-
不过这个函数不需要直接使用, 因为download_GEO和get_GEO_pheno会进行自动调用
-
这里主要讲解下过程和原理
-
getGEO2有默认的下载方式, 其中curl是更推荐的一种方式, 但仍存在超时的情况
-
所以我这里优先使用本地的curl进行下载, 如果你还没有安装则需安装一下, 并添加系统环境变量
-
下载完后也会自动保存样本临床信息
-
自动判断是否需要进行log2(expM + 1)处理
-
自动进行样本间矫正
-
最终返回临床信息和矫正后的表达谱, 但此时探针ID还没有进行注释
# 相当于 download_GEO 的中间步骤
obj = getGEO2("GSE294904", out_dir = "test/00_GEO_data_GSE294904")
INFO [2025-07-09 23:52:33] Querying dataset: GSE294904 ...
- Use local curl
- Found 1 GPL
- Found 6 samples, 34 metas.
- Writting sample_anno to test/00_GEO_data_GSE294904/GSE294904_sample_anno.csv
- Execute log(expM + 1) ...
- Normalize between arrays ...
- Successed, file save to test/00_GEO_data_GSE294904/GSE294904_GPL570.RData.
obj$GPL
'GPL570'
hd(obj$expM)
dim: 54675 × 6
GSM8928113 GSM8928114 GSM8928115 GSM8928116 GSM8928117
1007_s_at 9.978914 10.098814 10.987668 11.046282 10.315491
1053_at 11.623185 11.606150 11.331921 11.334088 10.947126
117_at 6.778689 6.949338 4.286260 3.549599 4.958118
121_at 10.248425 10.474015 10.292886 10.556959 9.458525
1255_g_at 2.812666 3.389816 3.084875 5.967788 2.155590
hd(obj$sample_anno)
dim: 6 × 34
title geo_accession status submission_date last_update_date
GSM8928113 NCI-ADR-RES control GSM8928113 Public on Apr 30 2025 Apr 17 2025 Apr 30 2025
GSM8928114 NCI-ADR-RES treated GSM8928114 Public on Apr 30 2025 Apr 17 2025 Apr 30 2025
GSM8928115 GCS control GSM8928115 Public on Apr 30 2025 Apr 17 2025 Apr 30 2025
GSM8928116 GCS treated GSM8928116 Public on Apr 30 2025 Apr 17 2025 Apr 30 2025
GSM8928117 asGCS control GSM8928117 Public on Apr 30 2025 Apr 17 2025 Apr 30 2025
read_GPL 系列
- 下载完之后就需要进行探针ID注释了, 这里是最麻烦的一步, 因为探针注释文件格式千变万化 -_-
- 所以第一步就是下载和读取GPL注释文件
- 这几个函数也是提供给大家, 可以自行构建注释表, 因为总注释表我更新肯定不及时, 而且平台太多了, 总是遇到没有注释过的平台
read_GPL
- 直接读取本地的GPL文件, 这种情况前提是你要能下载下来
- 先不做处理
# https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GPL570
# 下载至 test/GPL570/GPL570-55999.txt
GPL570_raw = read_GPL("test/GPL570/GPL570-55999.txt")
hd(GPL570_raw)
dim: 54675 × 16
ID GB_ACC SPOT_ID Species Scientific Name Annotation Date
1 1007_s_at U48705 Homo sapiens Oct 6, 2014
2 1053_at M87338 Homo sapiens Oct 6, 2014
3 117_at X51757 Homo sapiens Oct 6, 2014
4 121_at X69699 Homo sapiens Oct 6, 2014
5 1255_g_at L36861 Homo sapiens Oct 6, 2014R
extract_GPL_anno
- 从读取好的注释数据框里提取探针ID列和包含基因名的列
- 这是最麻烦的一点, 这一列有很多不同的写法, 我们总结了以下几类列名
colnames_SYMBOL = c("Gene symbol", "Gene Symbol", "GENE_SYMBOL", "GeneSymbol", "Symbol", "SYMBOL", "gene_assignment")
colnames_ENTREZ = c("ENTREZ_GENE_ID")
colnames_REFSEQ = c("GB_ACC")
colnames_ENSEMBL = c("GENE_ID", "SPOT_ID")
colnames_candicate = c(colnames_SYMBOL, colnames_ENTREZ, colnames_REFSEQ, colnames_ENSEMBL)
- 只要存在这些列就说明是可以注释的, 只需要使用
bitr函数进行ID转换就行了
load2("./test/00_GEO_data_GSE294904/GSE294904.RData")
Loading objects:
GSE294904
GPL = GSE294904$GPL
GPL
[1] "GPL570"
expM_raw = GSE294904$expM_raw
hd(expM_raw)
dim: 54675 × 6
GSM8928113 GSM8928114 GSM8928115 GSM8928116 GSM8928117
1007_s_at 10.014355 10.135966 11.008001 11.071630 10.268858
1053_at 11.633831 11.610780 11.346647 11.354608 10.919310
117_at 6.661878 6.857272 4.313413 3.496718 5.036547
121_at 10.278252 10.507507 10.323595 10.588387 9.398323
1255_g_at 2.666673 3.210481 3.041658 5.902110 2.303889
GPL570_anno = extract_GPL_anno(GPL570_raw)
Found annotation column: Gene Symbol
hd(GPL570_anno)
dim: 54675 × 2
ID SYMBOL
1 1007_s_at DDR1
2 1053_at RFC2
3 117_at HSPA6
4 121_at PAX8
5 1255_g_at GUCA1A
- 这样我们就得到了ID转换表了, 已经成功一大半了
read_GPL_file
- 从本地GPL读取注释文件(read_GPL实现)
- 随后使用extract_GPL_anno提取探针ID列和包含基因名的列
- 返回包含两列的注释表: ID列和SYMBOL列
# 读取和提取一步到位
anno_tb = read_GPL_file("./test/GPL570/GPL570-55999.txt", out_dir = "test/GPL570")
Processing: GPL570 ...
Found annotation column: Gene Symbol
hd(anno_tb[[1]])
dim: 54675 × 2
ID SYMBOL
1 1007_s_at DDR1
2 1053_at RFC2
3 117_at HSPA6
4 121_at PAX8
5 1255_g_at GUCA1A
- 成功生成注释表后, 将自动与总注释表进行合并
- 这也是这里返回的是列表的原因, 因为总的注释数据就是一个列表, 每个元素就是一个平台的注释数据框
- 合并完之后会自动保存到fastGEO的安装目录下的data目录
- 下次就不需要再为这个平台准备注释文件了, 因为download_GEO 默认首先会从这里读取总注释数据
- 所以自己遇到我还没整理的平台可以运行一下这几个函数, 成功后再次运行 download_GEO可能就好了
read_GPL_url
- 很多小伙伴会遇到一个问题, 就是GPL注释文件虽然可以查看, 但是不能下载
- 而且查看久了浏览器会卡死, 因为几万行的数据浏览器是扛不住的
- 后来我发现这个查看的网页的注释内容是固定且完整的, 所以将网页下载下来并提取注释表格就行了
- 嘿, 我真他娘的天才!还真可以!
- 你只需要快速将网页的链接粘贴下来, 然后就能关闭网页了, 不然等会浏览器就崩溃了
- 拿到链接之后直接扔给
read_GPL_url就能获取处理完之后的注释表格了 - 所以这里包含从HTML提取注释表格并执行
extract_GPL_anno两个步骤
url = "https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?view=data&acc=GPL10379&id=5393&db=GeoDb_blob45"
anno_tb = read_GPL_url(url, out_dir = "test/GPL1037")
Processing: GPL10379 ...
File downloaded successfully!
Found annotation column: GeneSymbol
New annotation file save to: C:/software/R-4.4.1/library/fastGEO/data/anno_obj.RData
hd(anno_tb[[1]])
dim: 52378 × 2
ID SYMBOL
11 100138926_TGI_at SLC16A1
12 100131045_TGI_at
13 100134882_TGI_at IFNG
14 100149534_TGI_at RAB14
15 100160054_TGI_at YTHDC2
anno_GEO 系列
- 那问题又来了, 如果平台没有提供GPL注释文件或不包含基因名信息怎么办?
- 好在我发现还有其他的获取注释文件的方法
- anno_GEO_AnnoProbe: Jimmy Zeng写的R包, 整理了很多R包的注释信息, 尤其是将一些只提供碱基序列的探针给比对了一遍
- anno_GEO_online: 少数情况下, 有些GSE自带注释文件, 可以直接在线下载读取
- 所以我们有三种注释的方式, 自己准备注释文件(或者我的包已经自带的)、AnnoProbe和online三种
anno_GEO_AnnoProbe
- 使用AnnoProbe包获取注释信息
anno_GEO_online
- 使用GSE里的在线注释文件获取注释信息
anno_GEO
- 准备好注释文件后就可以对表达谱进行注释了
- 由于存在多个探针对应同一个基因的情况, 所以需要进行去重
- 前面有讲过, 通过method参数指定重复基因的处理方法
- max: 默认, 取最大表达量做为该基因的表达水平, 相对较快且合理
- mean: 取均值, 太慢
- remove: 直接去除重复基因, 最快, 但不推荐
- sum: 求和, 用的比较少, 暂不推荐
anno_NGS
- 这个是独立的功能, 用于对行名是ENSEMBL ID的二代测序表达谱的注释
- 二代测序表达谱数据一般存放在补充文件
date_to_gene
- 最后还有一个功能, 就是我发现有些表达谱的基因名是日期, 不排除是Excel搞的鬼: R语言-将日期恢复为基因
- 所以最后再检查以下是否包含日期名, 如果有则自动进行转换
run_fastGEO_app
- 最后还有个更逆天的功能, 你可以在浏览器执行这些操作
- 其实就是使用Shiny基于我的这些函数搭建的一个分析平台
- 其实我并不会Shiny的语法, 是ChatGPT帮我写的, 我也不太会改, 所有现在还是有点bug
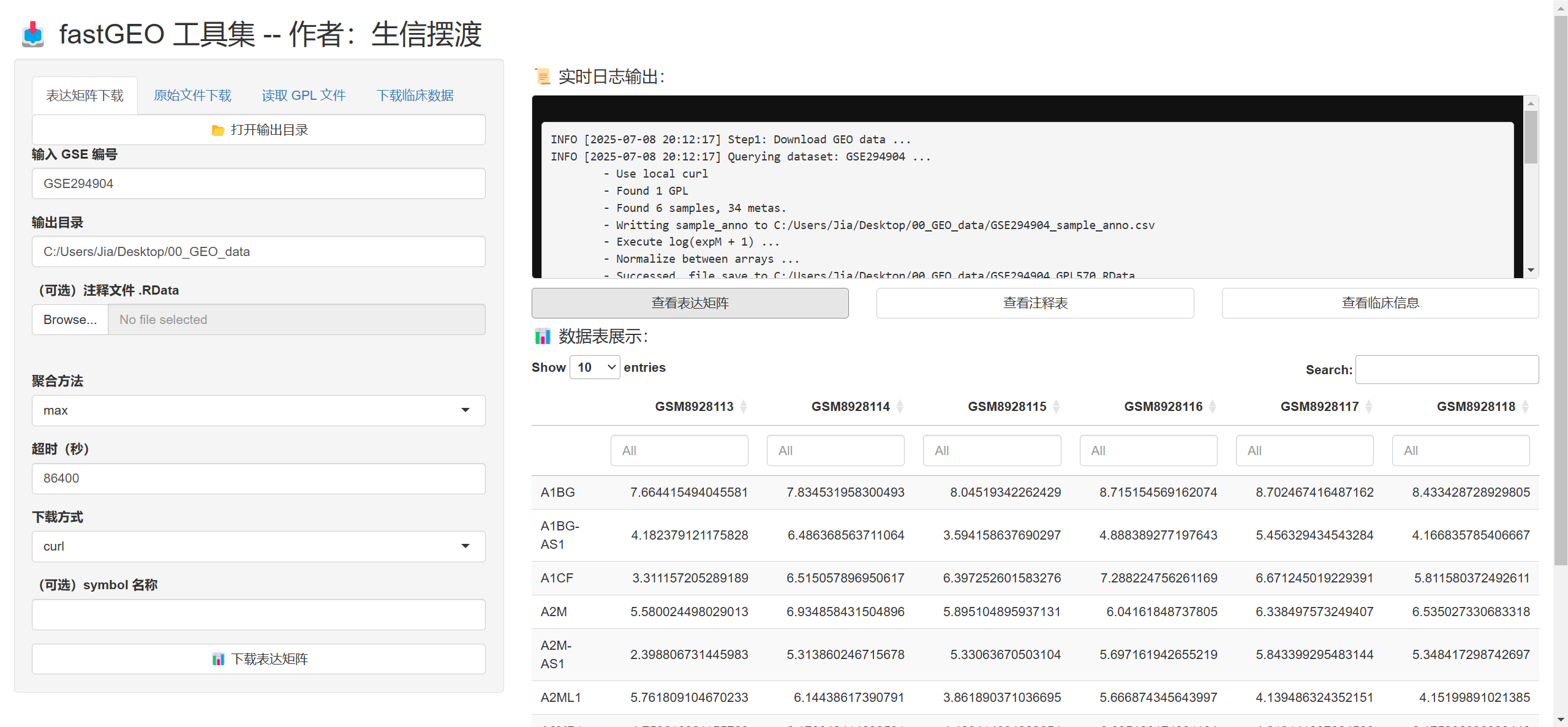
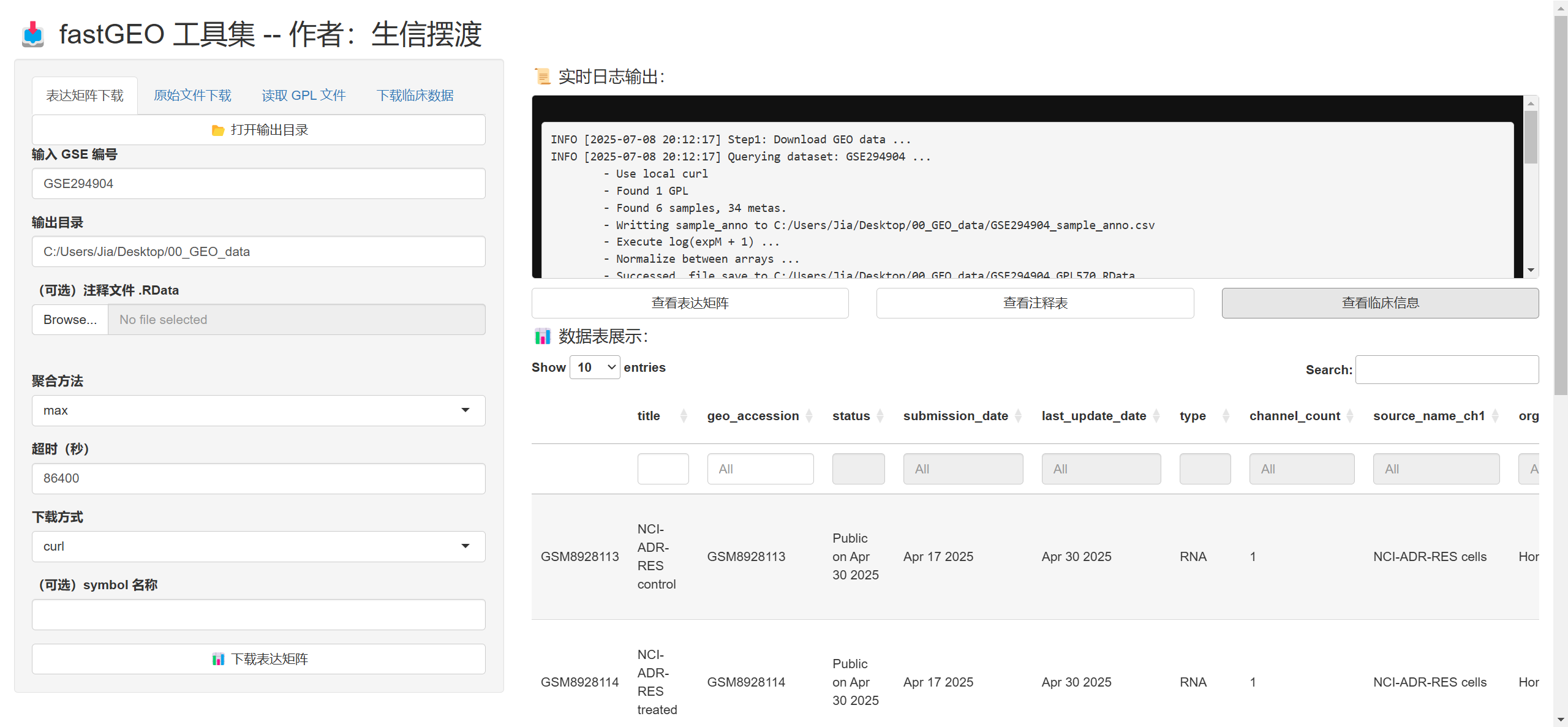
- 大家可以体验一下😅, 目前还是要在R里面操作, 更灵活一点, 这个是搞着玩的暂时就是
# 执行下面的函数打开Shiny App
# 我这里就不运行了, 只展示截图
# run_fastGEO_app()
安装依赖R包
必须安装的
- 执行基础的功能所必须的
# if (!require("BiocManager", quietly = TRUE))
# install.packages("BiocManager")
# BiocManager::install("GEOquery")
# BiocManager::install("limma")
# devtools::install_github("jmzeng1314/AnnoProbe")
# install.packages("data.table", repos="https://rdatatable.gitlab.io/data.table")
# devtools::install_github("hadley/readr")
# install.packages("stringr")
# install.packages("httr")
# install.packages("rvest")
# install.packages("shiny")
# install.packages("DT")
建议安装的
- 这些可能不太容易安装, 尽量安装一下
# BiocManager::install("org.Hs.eg.db")
# BiocManager::install("org.Mm.eg.db")
# BiocManager::install("clusterProfiler")
安装我的工具包 fastR
- 有很多函数是依赖我的这个工具包的
- 当前文件夹没有安装包的需要付费获取安装包:
- 直接添加微信转账50, 发送压缩包
- 以前是20, 老用户依然可免费获取
- 很多资料后面都会慢慢涨价, 早买会便宜很多!
- 购买后可进入交流群,后续版本更新和问题答疑都在群里进行

# 自动寻找当前目录下的压缩包
file_fastR = grep("^fastR.*.gz", dir(), value = TRUE)
file_fastR
‘fastR_1.3.0.tar.gz’
# 如果你正在使用要安装或更新的R包, 需要先取消加载, 取消占用
# detach("package:fastGEO", unload = TRUE)
# 强制进行安装更新
# install.packages(file_fastR, repos = NULL, upgrade = FALSE, force = TRUE)
安装 fastGEO
# ## 如果以前安装过, 可能本地存在注释文件, 如果有则需要备份
# ## 重新安装前保存预先存在的注释文件
# anno_file = paste0(.libPaths(), "/fastGEO/data/anno_obj.RData")
# anno_file_temp = paste0("temp-", basename(anno_file))
# anno_file_exists = FALSE
# if(file.exists(anno_file)){
# file.copy(anno_file, anno_file_temp)
# anno_file_exists = TRUE
# }
# anno_file_exists
TRUE
# ## 安装fastGEO
# file_fastGEO = grep("^fastGEO.*.gz", dir(), value = TRUE)
# install.packages(file_fastGEO, repos = NULL, upgrade = FALSE, force = TRUE)
# ## 重新拷贝注释文件
# if(anno_file_exists){
# file.copy(anno_file_temp, anno_file)
# unlink(anno_file_temp)
# }
- 安装完之后按照上一节的内容自行测试就行了




















 1012
1012

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








