本文来源公众号“arXiv每日学术速递”,仅用于学术分享,侵权删,干货满满。
原文链接:QuantUNet:基于Brevitas库量化U-Net,结合自定义损失函数,助力医学肿瘤分割在资源受限设备实时低功耗部署 !

医学影像分割,特别是在肿瘤分割方面,是一项关键任务,在医学成像中具有重要意义。U-Net作为一种广泛采用的卷积神经网络(CNN)架构被用于这一目的。
然而,U-Net对计算和内存资源的需求较高,这给在诸如可穿戴医疗系统等资源受限设备上的部署带来了挑战。本文通过引入QuantUNet解决了这些挑战,QuantUNet是专门为低功耗设备(如现场可编程门阵列FPGA)上的高效部署而优化的U-Net的量化版本。
使用Brevitas [7],这是一个用于量化感知训练的PyTorch库,作者对U-Net模型进行了量化,将其精度降低到平均4.24位,同时验证准确率仍达到94.25%,比浮点基准模型低1.89个百分点。量化后的模型大小减少了8倍,使其适用于可穿戴医疗设备中的实时应用。
作者采用了一个自定义损失函数,该函数结合了二元交叉熵(BCE)损失、Dice损失和位宽损失函数,以同时优化分割精度和模型大小。利用这个自定义损失函数,作者将找到最佳模型位宽与准确性的组合所需的训练次数从假设的6^23次显著减少到了一次,即通过上述损失函数组合找到了最优解。
该模型使用整数运算的特点突显了其在FPGA和其他特定AI加速硬件上的部署潜力。本文通过使深度学习模型能够在资源受限的设备上部署,推动了医学影像分割领域的发展,为可穿戴健康护理应用提供了实时、低功耗的诊断解决方案。
1 Introduction
医学图像分割,尤其是肿瘤分割,是医学影像领域的一项关键任务。U-Net,一种卷积神经网络(CNN)架构,因其在处理生物医学图像方面的有效性而被广泛应用。然而,U-Net 对计算和内存需求较高,这为其在资源受限设备(如手机或嵌入式系统)上的部署带来了挑战。
量化是一种减少神经网络权重和激活值精度的过程,为解决这个问题提供了有希望的方案。通过量化模型,作者可以显著减小其大小和计算需求,使其更适合在真实场景中部署,特别是在可穿戴医疗设备中的应用。
可穿戴医疗设备越来越多地用于连续健康监测、早期诊断和治疗领域。这些设备通常在对能量使用效率要求极高的环境中运行,因为用户的生命可能依赖于设备能在长时间内可靠工作。
例如,一种能够进行实时癌症诊断的可穿戴设备,比如一种由AI辅助的超声扫描仪以实现早期肿瘤检测,可以为用户提供即时洞察,而这原本可能需要临床影像检查。如果这种设备出现故障,可能会延误关键性的诊断,从而导致病情恶化或错失治疗机会。然而,深度学习模型如U-Net的计算需求可能是其有效部署在这些设备中的一个重大障碍。
本研究中对U-Net模型进行量化的主要动机是能够在嵌入式设备上部署该模型,特别是Field-Programmable Gate Arrays (FPGA)或其它类型的神经网络加速器。FPGA在执行量化模型时具有高度效率,因为它能够比浮点运算更高效地进行整数算术运算。研究表明,在FPGA上进行整数操作相比浮点操作可以在性能上提高一个数量级,并且需要显著更少的内存。这种效率对于可穿戴医疗设备至关重要,因为功耗和电池寿命是关键因素。
通过量化U-Net模型,作者旨在减少其计算和内存开销,使其能够在低功耗嵌入式系统上运行,而不牺牲肿瘤分割的准确性。这种方法不仅能提高设备的效率,还能延长电池寿命,确保在较长的时间内保持可靠的运行。
本文的主要贡献如下:
-
Baseline 浮点U-Net:作者使用浮点U-Net模型建立了一个 Baseline ,用于肿瘤分割。
-
Brevitas量化U-Net:作者使用Brevitas[7]对整个U-Net模型进行了量化,Brevitas是一个用于量化感知训练的PyTorch库。
-
位宽优化:作者将位宽设为可训练参数,并结合自定义损失函数和梯度下降法来找到保留浮点模型准确性的最小位宽。
2 Related Works
量化感知训练(QAT)已被广泛研究,作为一种减轻量化通常引起的准确率损失的方法。QAT 在训练过程中模拟量化的影响,使模型能够适应较低的精度。多项研究表明,在显著减小模型规模和计算需求的同时,QAT 能够保持模型的准确率。近期在 QAT 方面的研究探索了将位宽[3, 8]作为可训练参数的想法,允许模型在训练过程中动态调整权重和激活的精度。这种方法可以通过针对不同层甚至单个神经元优化位宽来实现更高效的模型。此外,量化技术也在医学影像领域得到探索,因为在该领域的深度学习模型往往面临严重的计算挑战。
3 Dataset
本研究使用的数据集是乳腺超声图像数据集[10, 1],这是一个公开可用的数据集。该数据集包含被分为三类的乳腺超声图像:正常、良性以及恶性。它特别设计用于肿瘤分割和分类等任务,因此非常适合用于评估U-Net架构在医学图像分析中的性能。
3.1 Dataset Description
该数据集包含总共780张图像,按类别分布如下:
正常:未见肿瘤或异常表现的图像。 良性:包含良性肿瘤的图像,良性肿瘤是非癌性的且通常较为温和。 恶性:包含恶性肿瘤的图像,恶性肿瘤是癌性的并需要立即医疗关注。
每张图像以PNG格式提供,并附有一个相应的 Mask ,用于标示感兴趣区域(ROI),例如肿瘤区域。这些 Mask 是二值图像,肿瘤区域用白色 Token ,背景用黑色表示。该数据集还包含元数据,如患者信息和肿瘤特征等,可用于进一步分析。
3.2 Dataset Preprocessing
数据集从结构化的目录加载,图像及其相应的 Mask 按类别整理到子目录中,分别为良性、恶性和正常。灰度图像被调整为固定分辨率的 (128 \times 128) 像素,并且图像像素值及其 Mask 的像素值都通过除以 255 归一化到范围 ([0,1]),使其适合神经网络输入。对于没有相应 Mask 的图像(例如正常情况),生成空白 Mask 以保持一致性,并为每幅图像- Mask 对分配一个标签 ((0) 表示良性,(1) 表示恶性,(2) 表示正常)。使用分层方法将数据集划分为训练集(70%)、验证集(15%)和测试集(15%),以确保所有子集中的类别分布保持一致,从而实现良性、恶性和正常情况的均衡表示。最后,图像和 Mask 转换为 PyTorch 张量,数据类型为 float32,并添加通道维度,灰度图像的形状变为 [1,128,128],使其准备好用于深度学习模型训练。
3.3 Dataset Challenges
该数据集提出了几个典型的医学影像任务挑战:
类别失衡:三类图像(正常、良性、恶性)之间的分布不均匀。通过过采样少数类别或在训练过程中应用类别加权损失函数等方式来解决这一问题。
噪声和伪影:由于成像模态的特性,超声图像中常常含有噪声和伪影。预处理步骤,例如归一化和数据增强,有助于减轻这些问题。
数据集规模小:仅拥有780张图像,对于深度学习任务来说数据集相对较小。通过使用数据增强和迁移学习来克服这一限制。
4 Baseline U-Net Model
本文的研究基准模型采用的是U-Net架构[9],这是一种专门为生物医学图像分割设计的卷积神经网络(CNN)。U-Net架构的特点在于其编码器-解码器结构以及跳接连接,这使得它能够有效地捕捉医学图像中的局部和全局特征。该架构由一个编码器、一个瓶 Neck 分和一个解码器组成,并随后跟随一个用于分割的单一输出头。表1提供了本文研究中所使用的U-Net架构概述。
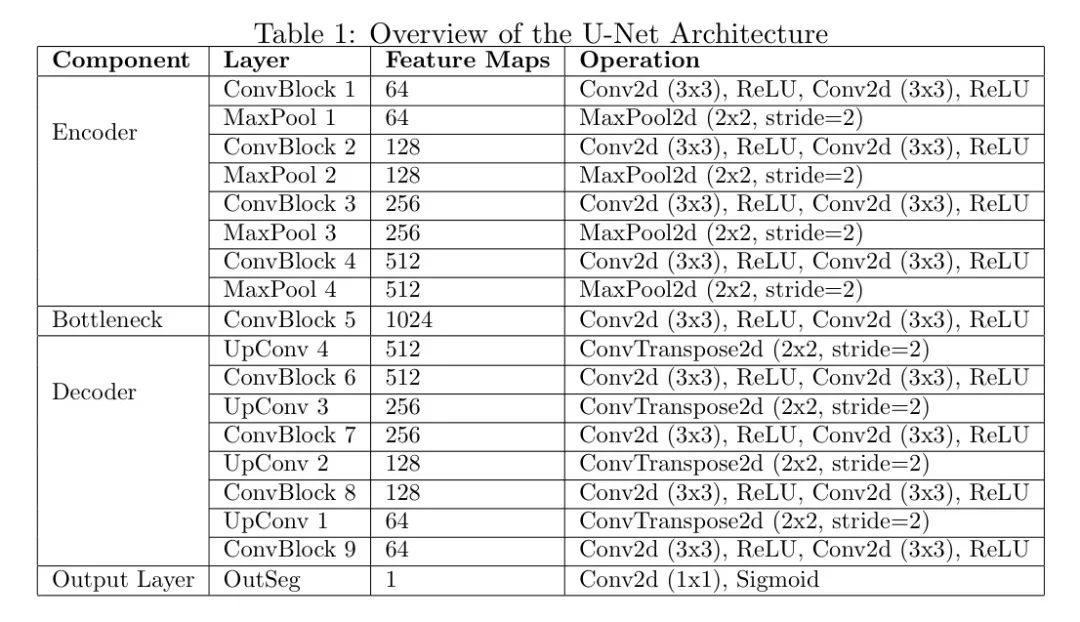
编码器由四个卷积块组成,每个块包含两层卷积层,后面跟着ReLU激活函数。每个块中的特征图数量在每次迭代时翻倍,从第一个块的64增加到第四个块的512。在每个卷积块之后,应用一个 Kernel 大小为2、步幅为2的池化操作以降低特征图的空间维度,同时保留重要特征。这种逐步下采样使编码器能够从输入图像中提取分层特征。
Bottleneck 层充当编码器和解码器之间的桥梁。它使用具有1024个特征图的卷积块处理最低分辨率的特征图。该层捕捉输入图像的最抽象和高层特征,然后将这些特征传递给解码器进行重建。
解码器由四个上卷积块组成,每个块都使用转置卷积层(也称为反卷积层)对特征图进行上采样。上采样的特征图通过 Short-Cut 连接与编码器中的相应特征图相连,从而使解码器能够整合高阶和细粒度的细节。每次拼接后都接着是一个卷积块来细化特征图,在逐步减少特征图数量的同时增加其空间维度。这种对称结构确保解码器能够准确重建输入图像的空间细节。
分割输出是由一个 1x1 卷积层生成的,该层将特征图的数量减少到 1,随后使用 Sigmoid 激活函数。此输出表示一个概率图,其中每个像素值指示其属于肿瘤区域的可能性。U-Net 架构旨在有效捕捉医学图像中的局部和全局特征,使其非常适合用于肿瘤分割任务。
4.1 Loss Function and Training
U-Net模型专门用于图像分割任务的训练,并采用结合了二元交叉熵损失(BCE Loss)和Dice损失的联合损失函数。训练过程由Trainer类管理,该类统筹训练和验证循环,计算性能指标并保存表现最佳的模型。训练框架的关键组件描述如下。
4.1.1 Loss Function

4.1.2 Dice Coefficient
为了评估分割性能,采用了Dice系数作为评价指标。Dice系数量化了预测的二值 Mask 与Ground Truth Mask 之间的重叠程度,其定义如下:

更高的Dice系数表明分割性能更好。
4.1.3 Training and Validation
训练过程包括两个主要循环:训练循环和验证循环。在训练循环中,模型使用综合损失函数(L_total)进行优化。验证循环在单独的验证数据集上评估模型的性能,并计算Dice系数作为主要指标。在整个训练过程中,记录并跟踪关键指标,包括训练损失、验证损失和Dice系数。
5 QuantU-Net
QuantU-Net 是原 U-Net 架构的一种优化版本,通过将权重和激活量化到较低的位宽来减少计算复杂度和内存使用。这种量化是通过使用 Brevitas 库实现的,该库使得可以在资源受限的设备上高效部署深度学习模型。在本节中,作者描述了 QuantU-Net 与原 U-Net 之间的关键差异。
Quant U-Net用Brevitas的量化版本替代了原始U-Net中的标准层。Brevitas层旨在提供对量化的精细控制,允许用户指定各种参数来确定权重和激活值的量化方式。这些参数对于平衡模型效率和准确性至关重要。影响量化过程的主要参数是位宽参数。较低的位宽可以减少内存使用和计算成本,但可能会影响模型的准确性。较高的位宽则可以保持准确性但会增加资源需求。
在本文中,作者定义最优模型为一个QuantU-Net模型,其平均位宽最小化,从而在资源使用量最少的同时,与浮点数对应模型相比,准确率下降微乎其微(例如,低于2%)。鉴于作者的U-Net模型包含23个卷积层,每层的位宽可以在2到8位之间变化,对最优配置进行 exhaustive 搜索需要评估 6^23 种可能的组合,这在计算上是不可行的。因此,作者选择将位宽本身设为可训练参数。结合下面所示的损失函数,QuantU-Net能够找到最优模型。
5.1 QuantU-Net Loss Function
QuantU-Net 在原 U-Net 实现的基础上引入了一种位宽正则化损失,以优化在有限精度硬件上进行高效推理的模型。QuantU-Net 模型采用了一种复合损失函数,该函数结合了分割损失、Dice损失和位宽正则化损失,以优化模型性能和量化效率。分割损失和 Dice 损失的计算方法与浮点原版相同。与原始版本相比,QuantU-Net 的损失函数通过引入一个位宽损失函数来区分自身,该函数返回整个模型的平均位宽。当此函数用于损失函数中时,它会对较高的位宽进行惩罚,以促进使用较低精度权重和激活值,从而减小模型大小和计算复杂度。总损失表达式为:

在训练过程中,模型通过梯度下降方法优化以最小化总损失,确保其在实现高分割准确性的同时,维持低位宽精度,以便能够在资源受限设备上高效部署。位宽正则化损失尤其关键,因为它有助于平衡模型性能与硬件效率。正如早期研究发现的那样,以低位宽初始化模型,并在必要时通过损失函数调整位宽,可以生成适用于实际穿戴式医疗成像应用的QuantU-Net模型。
6 Experimental Set-up
本研究的实验是在Google Colab环境中进行的,该环境提供了Tesla T4 GPU以加速计算。此环境配备了一块带有16 GB GPU内存的Tesla T4 GPU,使其能够高效地训练和推理深度学习模型。CPU由2颗Intel(R) Xeon(R)处理器组成,运行速度为2.20 GHz,每颗处理器具有1个核心和2个线程,总共提供了2个逻辑处理器。系统大约有12.68 GB的可用RAM,在执行时有7.63 GB空闲,并且不使用交换内存,因为交换分区大小设置为0 kB。
7 Results
QuantU-Net 模型在训练了 40 个epoch 后,其结果总结在表2 中。单精度 U-Net 模型达到了 96.14% 的准确率,作为基准模型提供了有力的对比参考。通过第 10 个 epoch,Quant U-Net 由于其量化权重和激活值,实现了验证 Dice 系数为 0.4947,尽管精度降低,但仍展示了竞争力的分割性能。模型的平均位宽稳定在约 4.24 位,使得模型大小约为单精度 U-Net 模型的八分之一。
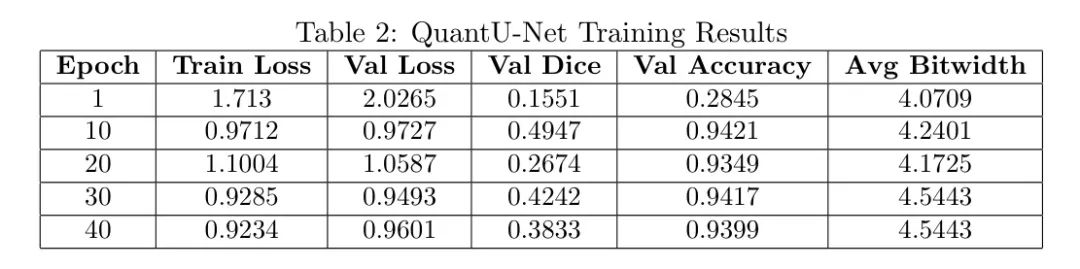
QuantU-Net 的验证精度始终保持在较高水平,在第 39 轮达到 94.25%,这仅比浮点 U-Net 模型的精度 96.14% 低 1.89%。这一微小的精度下降是一个显著的成就,考虑到模型尺寸和计算复杂性的大幅减少。在整个模型中的平均位宽从第一轮的 4.0709 位略微增加到第 30 轮的 4.5443 位,反映了模型适应以维持精度的同时优化为较低精度。
模型层之间的位宽分布保持稳定,大多数层被量化为4或5位。如图1所示,在训练过程中,各层的位宽动态变化。 Bottleneck 层和上卷积层等层调整了其位宽以保持准确性并尽量减少精度损失。 Bottleneck 层对于特征提取至关重要,在早期轮次中被量化为3位,并在后期增加到4位,这表明模型更优先考虑关键层的准确性。
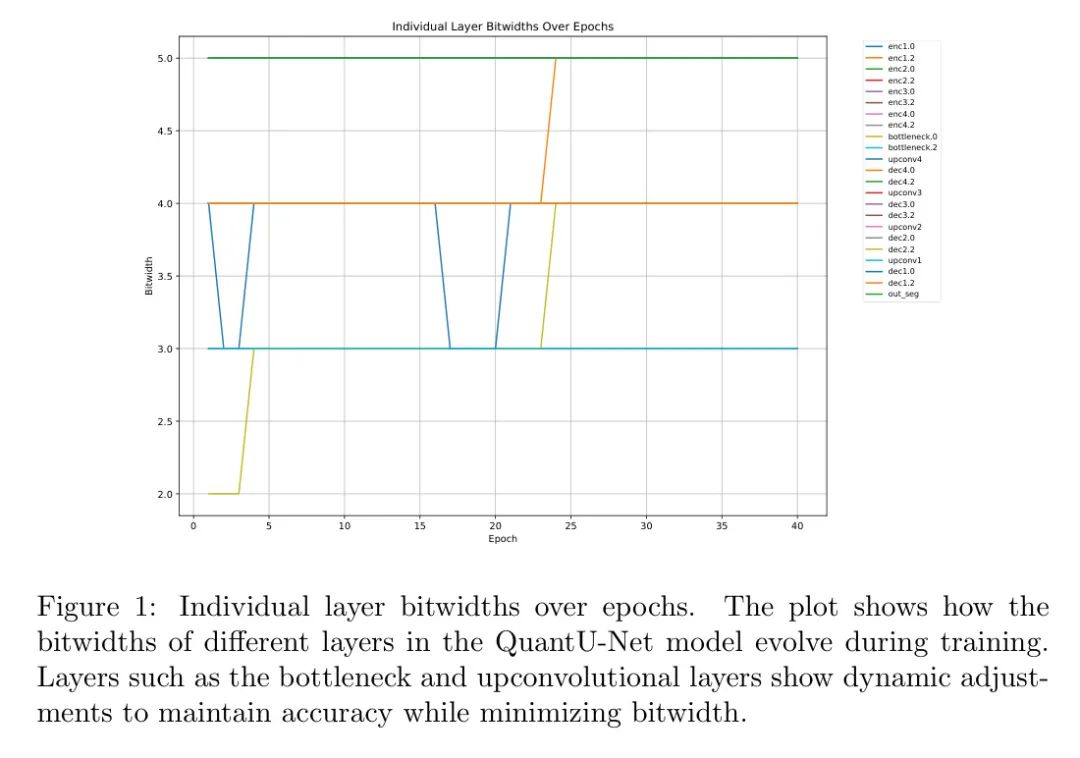
图2展示了验证精度和平均位宽随 Epoch 变化的折中关系。左侧 y 轴显示了验证精度,而右侧 y 轴则显示了模型上的平均位宽。该图表表明,在平均位宽收敛至约 4.24 位的过程中,精度保持稳定,突显了量化方法的有效性。
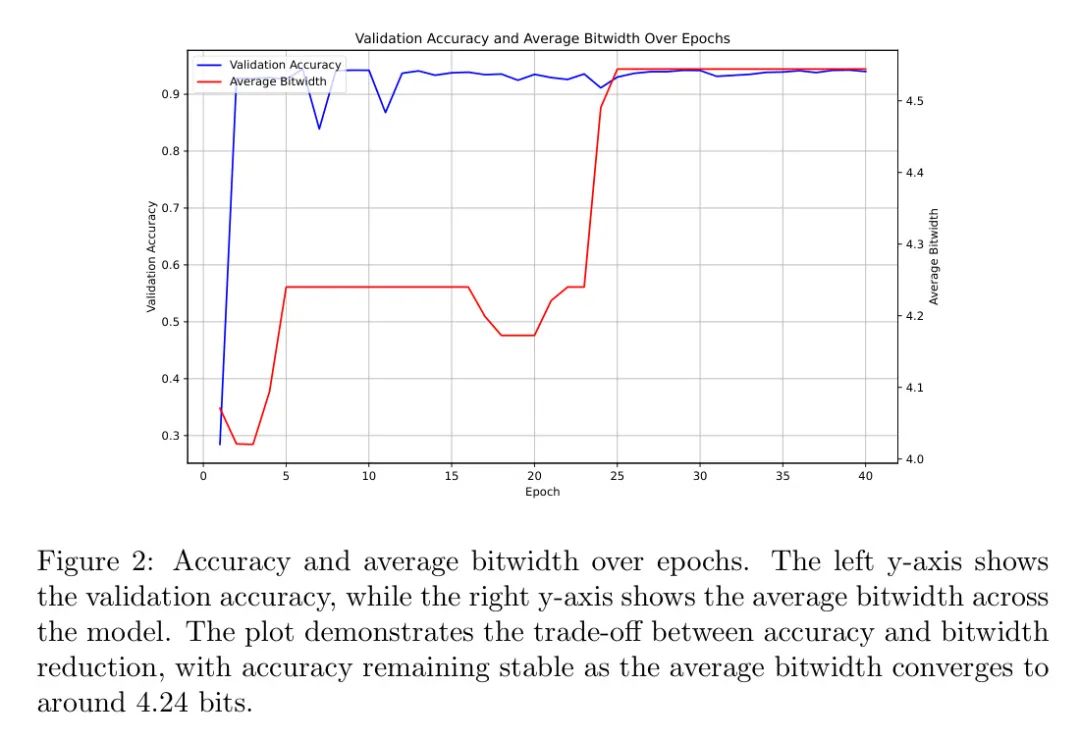
此外,图3显示了位宽正则化损失和分割损失在训练周期中的关系。随着模型优化以实现更低的精度,位宽损失逐渐降低,而分割损失保持稳定,这表明尽管位宽减少,模型仍维持了准确性。
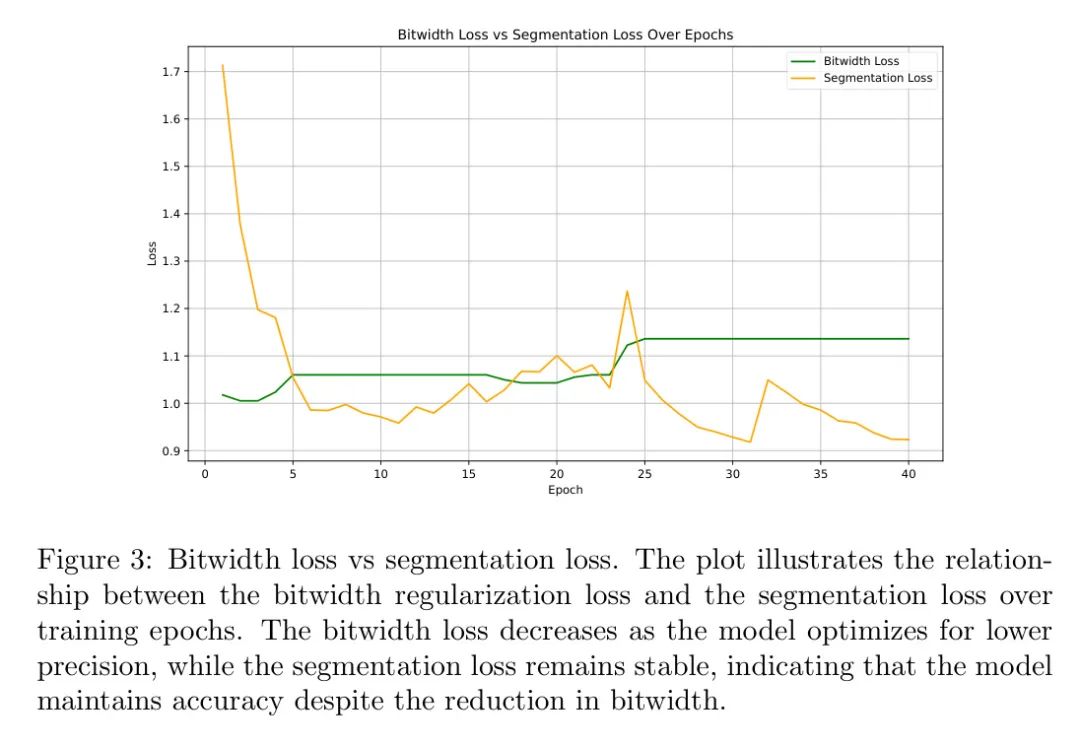
所有这些结果都在单次训练会话中获得,与通过穷举搜索寻找最优位宽所需的想象中的次训练会话形成了对比。这一效率突显了所提出位宽优化方法的有效性,该方法利用梯度下降和自定义损失函数动态确定最小可能的位宽,而不牺牲准确性。
8 Discussion
研究结果表明,QuantU-Net 成功平衡了模型效率和分割精度之间的trade-off。浮点U-Net模型的准确性达到了96.14%,而Quant U-Net的验证准确性为94.25%,但其模型大小大约小了8倍。这一模型大小的减少直接来自于将权重和激活量化到平均4.24位,显著减少了内存使用量和计算成本。
在QuantU-Net中使用整数操作相较于 Baseline 模型中的浮点操作,为在FPGA和其他AI加速器上的部署打开了巨大的可能性。FPGA特别适合运行量化模型,因为它们能够比浮点操作更高效地执行定点算术运算。
从96.14%下降到94.25%的最小准确率下降证实了量化感知训练(QAT)在保持模型性能的同时减少精度的有效性。位宽正则化损失起到了关键作用,引导模型尽可能使用较低的位宽而不牺牲准确率。这一点在 Bottleneck 层尤其明显,在后期epoch中,位宽略有增加以维持特征提取的质量。
一个值得注意的观察是在验证Dice系数上出现的变化,该系数在第10个epoch达到0.4947的最大值,但在后续epoch中有所波动。这种变化性表明,虽然模型的准确率较高,但在稳定性方面仍有改进的空间。未来的工作可以探索自适应量化技术,根据输入数据的复杂度动态调整位宽,这可能同时提高准确率和稳定性。
模型尺寸的减小以及使用整数操作使得Quant UNet非常适用于部署在资源受限设备上,例如可穿戴医疗设备和嵌入式系统。在FPGA和其他AI加速器上的潜在效率提升进一步增强了该模型在实际应用场景中的适用性,在这些场景中低功耗和实时性能至关重要。
9 Conclusion
本研究对U-Net架构在肿瘤分割中的应用进行了全面分析,解决了在资源受限设备上部署深度学习模型所面临的计算挑战。通过利用Quantization Aware Training(QAT)并借助Brevitas进行位宽优化,QuantU-Net模型在降低计算复杂度和内存使用量的同时,保持了竞争力的分割精度。
研究结果表明,QuantU-Net有效地平衡了模型效率与准确性的trade-off,使其适用于穿戴式医疗设备和嵌入式系统的部署。位宽优化方法不仅减少了模型大小,还为连续健康监测应用中提高能源效率铺平了道路。
尽管取得了这些进展,但仍存在挑战,特别是在量化效率与最小化精度退化的平衡方面。未来的工作可以探索自适应量化技术以及混合模型以进一步优化性能。此外,可以在更大规模和更多样化的数据集上验证QuantU-Net模型,并将其部署在各种硬件平台上(如ASIC和其他神经网络加速器),以评估其延迟、功耗和内存效率,从而增强其实用性并适用于实际临床场景。
本研究通过展示量化感知U-Net模型在高效和精确肿瘤分割方面的潜力,为医疗图像分割领域做出了贡献,铺平了通往实时、低功耗及可穿戴诊断解决方案的道路。
10 Acknowledgements参考
[0]. QuantU-Net: Efficient Wearable Medical Imaging Using Bitwidth as a Trainable Parameter .
THE END !
文章结束,感谢阅读。您的点赞,收藏,评论是我继续更新的动力。大家有推荐的公众号可以评论区留言,共同学习,一起进步。























 1547
1547

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








