作业链接
0.作业题目
- 从ucsc的xena浏览器里面下载感兴趣癌症,比如肝癌的表达矩阵(counts值)
- 然后根据样本名字拿到有配对的几十个病人的癌症和正常对照数据(部分癌症数据并没有对照)
- 接着提取感兴趣基因(比如TP53)的表达量
- 最后套用上面的绘图代码即可!
1.数据下载
下载网址

然后找到LIHC
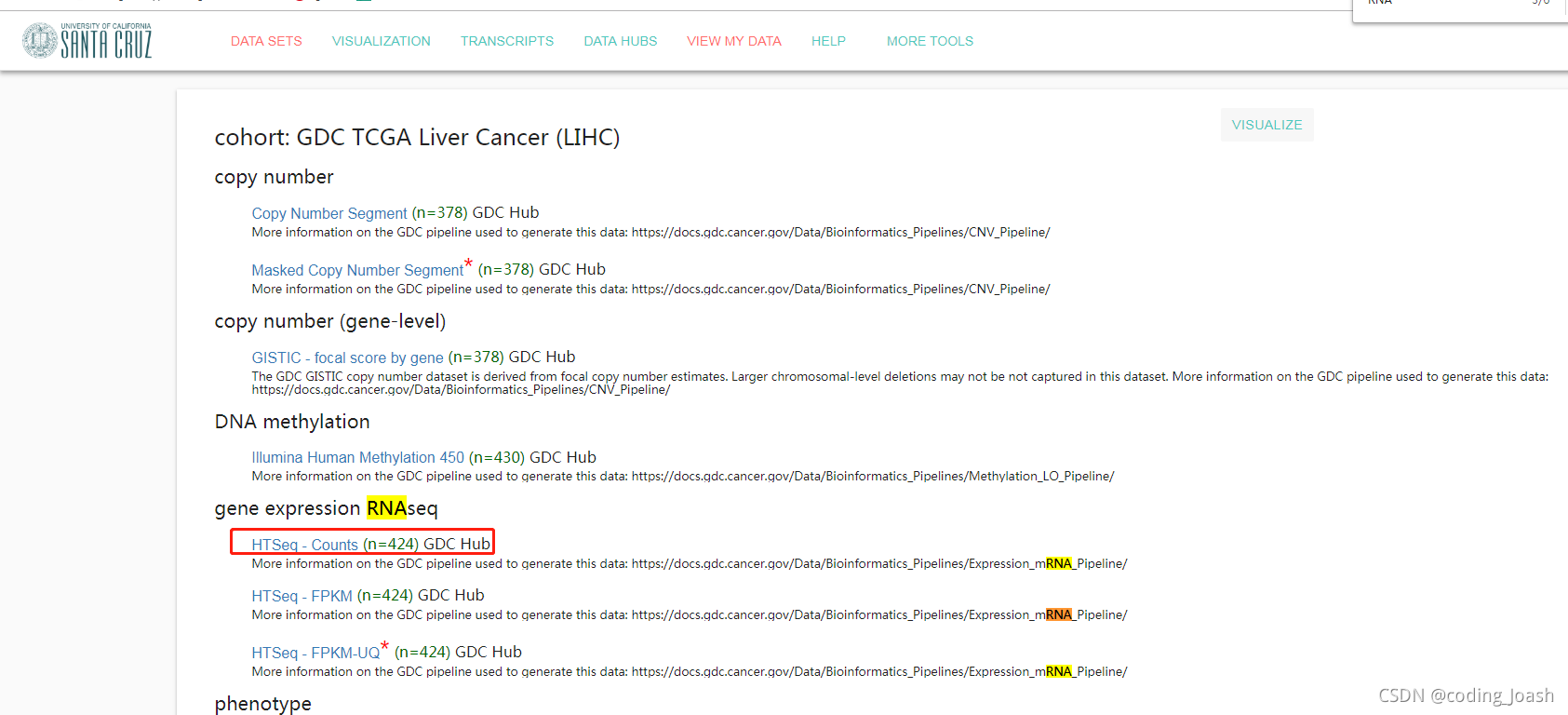
点击进去下载即可

2.数据提取以及简单统计
提取TP53的表达量数据
#TP53的ensemble id 为ENSG00000141510
zcat TCGA-LIHC.htseq_counts.tsv.gz | grep -E 'Ensembl_ID|ENSG00000141510' >TP53_tcga_expression.txt
library(dplyr)
tp53_tcga = read.table('TP53_tcga_expression.txt',header = T,check.names = F)
rownames(tp53_tcga) = 'TP53'
tp53_tcga = tp53_tcga[,-1]
统计正常样品 和 肿瘤 样品个数
> table(colnames(tp53_tcga) %>% sub('TCGA-\\w+-\\w+-','',.) )
 肝癌TP53基因表达分析与可视化
肝癌TP53基因表达分析与可视化





 该博客介绍了如何从UCSC Xena浏览器下载肝癌(LIHC)的表达矩阵,提取TP53基因的表达数据,并统计正常和肿瘤样本数量。通过筛选,获取了50个具有配对肿瘤和正常样本的病人数据,进一步提取并对比了这些样本中TP53基因的表达量,最后使用ggpubr包进行可视化展示。
该博客介绍了如何从UCSC Xena浏览器下载肝癌(LIHC)的表达矩阵,提取TP53基因的表达数据,并统计正常和肿瘤样本数量。通过筛选,获取了50个具有配对肿瘤和正常样本的病人数据,进一步提取并对比了这些样本中TP53基因的表达量,最后使用ggpubr包进行可视化展示。
 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章

















 703
703

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








