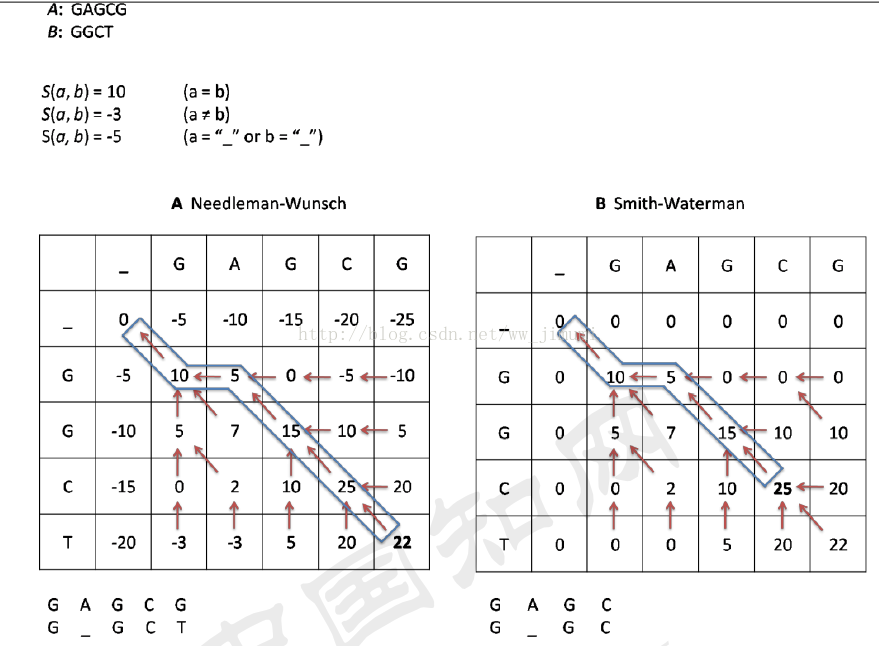
1. 加分规则
S(a,b) = 10 (a=b)
S(a,b) = -3(a!=b)
S(a,b) = -5(a='_' or b='_')
通常DNA和RNA,Protein都有打分矩阵,Protein常用打分矩阵BLOSUM62
2. 动态规划算法:
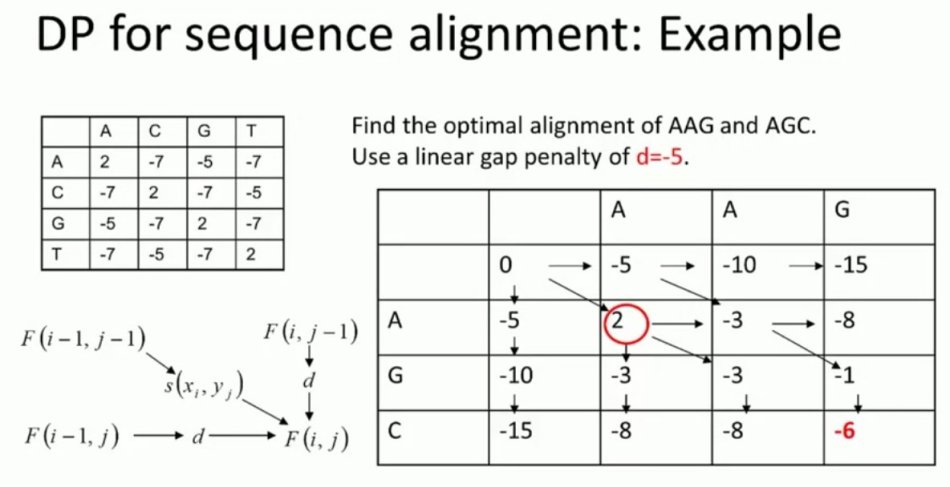
全局比对算法Needleman-Wunsch思路:从第一个到最后一个,一个个比,一条序列比个遍,然后得出最优解

example:
局部比对Smith-Waterman算法:

example:

综合比较:
首先,在初始化阶段,第一行和第一列全填充为 0(而且第一行和第一列的指针均为空)。
第二,在填充表格时,如果某个得分为负,那么就用 0 代替,只对得分为正的单元格添加返回指针。
最后,在回溯的时候,从得分最高的单元格开始,回溯到得分为 0 的单元格为止。除此之外,回溯的方式与 Needleman-Wunsch 算法完全相同





 本文探讨了DNA序列比对中的两种动态规划算法——Needleman-Wunsch全局比对算法和Smith-Waterman局部比对算法。讲解了加分规则,并使用BLOSUM62打分矩阵。通过实例解析算法的执行过程,包括初始化、表格填充和回溯策略,帮助理解如何寻找最优解。
本文探讨了DNA序列比对中的两种动态规划算法——Needleman-Wunsch全局比对算法和Smith-Waterman局部比对算法。讲解了加分规则,并使用BLOSUM62打分矩阵。通过实例解析算法的执行过程,包括初始化、表格填充和回溯策略,帮助理解如何寻找最优解。
















 1万+
1万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








