核心思路
- 需求常见词:【每组
xxx的第一个yyy的zzz】【每组xxx的最后一个】
【每组xxx的前n个】【每组最xx的前n个】 - 公式:
row_number() over(partition by 分组字段 order by 排序字段) as rn,再筛选rn<=N名
例题一:跨越物流
create table emp(empno string ,ename string,hiredate string,sal int ,deptno string);
insert overwrite table emp values
('7521', 'WARD', '1981-2-22', 1250, 30),
('7566', 'JONES', '1981-4-2', 2975, 20),
('7876', 'ADAMS', '1987-7-13', 1100, 20),
('7369', 'SMITH', '1980-12-17', 800, 20),
('7934', 'MILLER', '1982-1-23', 1300, 10),
('7844', 'TURNER', '1981-9-8', 1500, 30),
('7782', 'CLARK', '1981-6-9', 2450, 10),
('7839', 'KING', '1981-11-17', 5000, 10),
('7902', 'FORD', '1981-12-3', 3000, 20),
('7499', 'ALLEN', '1981-2-20', 1600, 30),
('7654', 'MARTIN', '1981-9-28', 1250, 30),
('7900', 'JAMES', '1981-12-3', 950, 30),
('7788', 'SCOTT', '1987-7-13', 3000, 20),
('7698', 'BLAKE', '1981-5-1', 2850, 30);
select * from emp;
题目描述
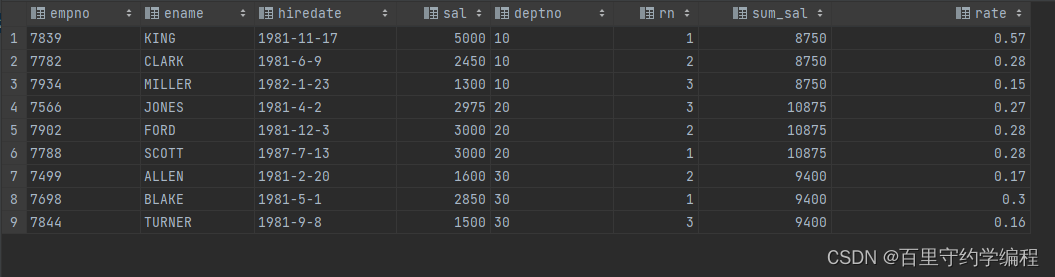
求出每个部门工资最高的前三名员工,并计算这些员工每个员工的工资占所属部门总工资的百分比。
分析:最终结果中需要包含员工信息,然后还需要包含三个新增的列(伪列),排名、部门总工资、员工占总工资的百分比
with t1 as (
select
*,
row_number() over (partition by deptno order by sal desc) as rn,
-- sum窗口函数没有order by时,即分组后的指定列值的总和,有order by时,则是指定列值的前缀累加和
sum(sal) over (partition by deptno) as sum_sal
from emp)
select
*,
round(sal / sum_sal,2) as rate
from t1
where rn <= 3;
例题二:小米电商
订单表名字:t_order.
字段:user_id, order_id, ctime(10位时间戳),city id,sale_num,sku_id(商品)
问题:20201201至今每日订单量top10的城市及其订单量(订单量对order id去重)`
create table t_order (user_id string,
order_id string,
ctime string,
city_id string,
sale_num int ,
sku_id string) ;
第一步:对数据做相关预处理,如时间戳转为日期,过滤出指定时间范围的订单,对订单id去重统计订单量
with t1 as (select to_date(ctime) cdate, city_id, count(distinct order_id) cnt
from t_order
where to_date(ctime) >= '2020-12-01'
and to_date(ctime) <= `current_date`()
group by to_date(ctime), city_id),
第二步:对日期分组,对销售量排序
t2 as (select *, row_number() over (partition by cdate order by cnt desc) rn from t1)
第三步:过滤出top10,保留所需结果字段
select cdate, city_id, cnt
from t2
where rn <= 10;





















 1232
1232

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








