快速体验
- 打开 InsCode(快马)平台 https://www.inscode.net
- 输入框输入如下内容
帮我开发一个单细胞RNA测序聚类分析系统,用于生物医学研究人员分析细胞异质性。系统交互细节:1.支持上传单细胞RNA测序数据 2.自动选择高变异基因 3.可视化聚类结果 4.提供NMI/ARI评估指标。注意事项:需处理数据高维稀疏特性。 - 点击'项目生成'按钮,等待项目生成完整后预览效果
单细胞RNA测序技术近年来在生物医学领域掀起研究热潮,但如何从海量数据中准确识别细胞亚群一直是技术难点。scDFN这篇论文提出的深度融合网络方法,为解决这一挑战提供了创新思路。
-
技术背景与创新点 传统单细胞聚类方法面临三大难题:高维度带来的计算负担、基因表达数据稀疏性、以及细胞间复杂关系的表征困难。scDFN通过双编码器架构创新性地解决了这些问题:自动编码器提取基因表达特征,改进的图自动编码器捕获细胞间拓扑关系,再通过跨网络融合机制实现信息互补。
-
核心架构解析 模型包含三大模块:数据处理模块采用Scanpy进行基因筛选和KNN构图;信息编码模块通过ZINB分布处理数据稀疏性;信息融合模块创新性地引入三重自监督策略,同时优化四种损失函数。这种设计既保留了细胞特性又捕捉了群体结构。
-
实验验证亮点 在32个数据集测试中,scDFN的NMI和ARI指标平均达到0.8131和0.7518,尤其在大规模聚类数据集表现突出。例如在49个聚类的TASIC数据上,它能清晰区分仅含15个细胞的微小亚群,这得益于其创新的潜在空间构建方式。
-
实际应用价值 对于生物医学研究者,scDFN提供了三大实用优势:处理百万级细胞的扩展能力、抗批次效应的鲁棒性、以及无需标记数据的自监督特性。这些特点使其特别适合肿瘤异质性、胚胎发育等复杂场景的研究。
-
技术演进方向 虽然当前模型在部分噪声数据集表现有待提升,但论文指出的改进方向很有启发性:引入注意力机制增强特征选择、开发动态图构建算法、优化多模态数据融合策略等,都是值得关注的未来发展方向。
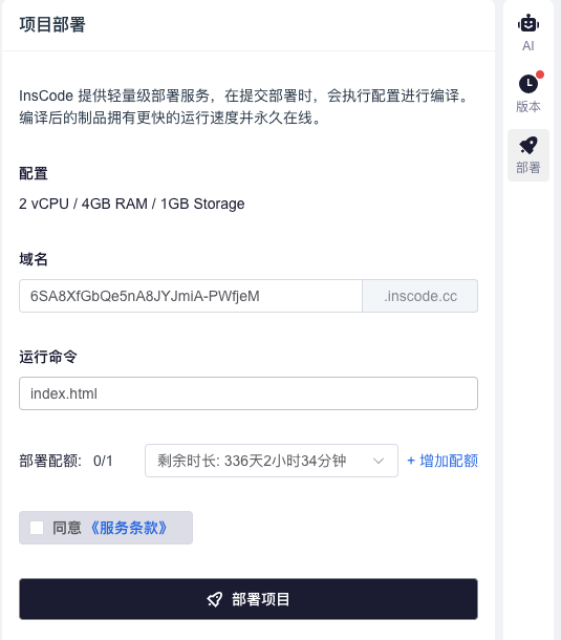
想快速体验单细胞分析流程?InsCode(快马)平台的AI生成功能可以一键构建分析环境,无需配置繁琐的生物信息学工具链。实际操作中发现其可视化结果生成非常便捷,特别适合想快速验证idea的研究者。平台还支持将分析流程部署为可分享的网页应用,方便团队协作查阅聚类结果。
创作声明:本文部分内容由AI辅助生成(AIGC),仅供参考





















 2221
2221

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










