X-Net:用于从3D腹部CT扫描中分割肝脏和肿瘤的多分支UNet网络
Neurocomputing 2021
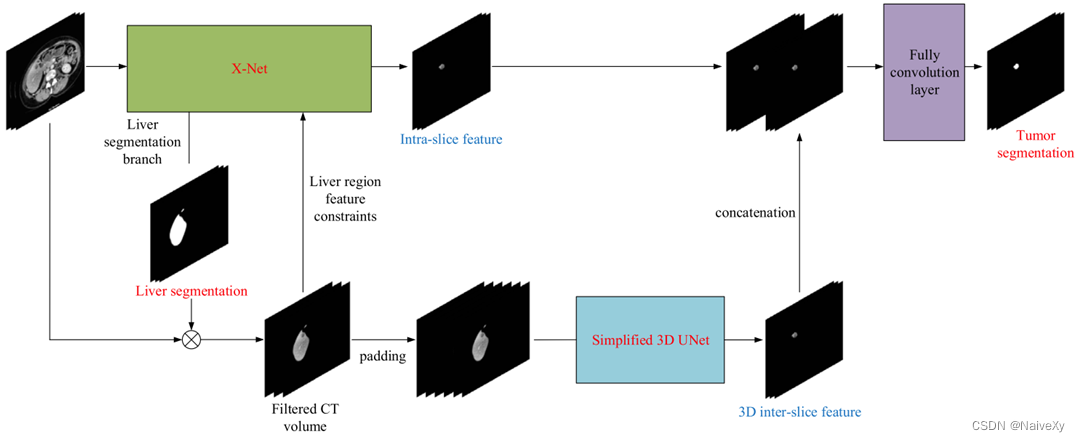
分割框架由以下部分组成:
1) 通过将肝脏区域解码和肝内金字塔特征提取的分支集成到DenseUNet结构中,设计了一个2D端到端网络;
2) 给定CT体积的每个切片都由修改后的DenseUNet处理,所以称为Dense X-Net,用于探索肝脏和肿瘤的切片内上下文信息;
3)堆叠的肝脏体积由具有简化卷积和反卷积核的 3D UNet处理以生成切片间肿瘤特征图,然后堆叠的内切片特征和切片间特征连接起来,用于具有完全卷积层的最终肿瘤分割;
4) 提出轮廓损失函数并与修改后的Dice损失函数一起采用作为联合损失函数,以更好地约束形状分割的肝脏和肿瘤。
Contributions:
1.提出了一种多分支深度神经网络来同时提取单个CT切片中的肝脏和肿瘤特征,其中在DenseUNet中添加一个额外的反卷积分支用于肝脏区域估计,并将估计肝脏的金字塔特征图用作肿瘤分割的互补特征。
2. 修改了传统的 3D UNet,以有效地同时利用切片内特征和切片间上下文信息,其中考虑了CT体积中切片之间的空间相干性,并简化了3D卷积和反卷积核以进行预测肝脏区域和肿瘤。
3. 提出了一种基于主动轮廓理论的形状损失函数,以反映预测结果与Ground Truth在区域轮廓方面的差异。我们还修改了传统的Dice损失函数,以便更好地考虑没有肿瘤或肝脏的负样本。然后使用联合损失函数来有效优化网络参数。
4. 我们在整个网络的训练过程中采用多任务学习策略,使得肝脏分割和肿瘤分割的分支能够得到更高效率的优化。
X-Net用于从单个CT切片中提取切片内特征
1.Backbone Dense UNet for tumor segmentation
我们提出的片内特征提取模块的主干是图中绿色矩形部分标记的2D DenseUNet。DenseUNet结合了密集连接路径和类UNet连接的优点,从而充分考虑了多尺度,来自输入图像的非常深的特征,参数量相对较少。然而,目前的DenseUNet方法将每个腹部切片处理为一个统一的图像,缺乏反映区域特征差异,即“肝脏与其他器官之间的差异”和“肿瘤与肝脏组织之间的差异”。为了进一步提高网络在表示腹部器官、肝脏和肿瘤之间视觉差异和关系的能力,我们在原始DenseUNet中添加了一个用于肝脏区域分割的分支,并利用分割后的肝脏区域来抑制肿瘤特征,命名为X-Net。
2. Dense UNet-branch for liver mask segmentation
图中的红色矩形说明了肝脏Mask分割分支的结构。来自输入CT体积的每个CT切片首先被送入4个连续的下采样密集块,以获得不同尺度的特征图。在对肿瘤特征图进行上采样之前,第4个下采样层通过4个连续的反卷积层进行肝脏Mask估计处理,然后与相应的低级Dense block进行快捷连接。
3.用于抑制肿瘤分割的分割肝脏区域的特征金字塔
上面讨论的网络检测到的肝脏Mask用于提取肿瘤分割的互补特征。图中的蓝色矩形部分显示了肝脏特征提取网络的结构以及与用于肿瘤分割的主干网络的特征图连接。输入切片由肝脏Mask过滤,以获得仅包含肝脏区域的图像。之后,图像被发送到一个4层特征金字塔网络(FPN)模块,以获得4个不同尺度的肝脏区域的特征图。最后,将每个特定的特征图与对应的下采样密集块连接起来,并将前一个上采样层的上采样输出作为当前上采样层的输入。
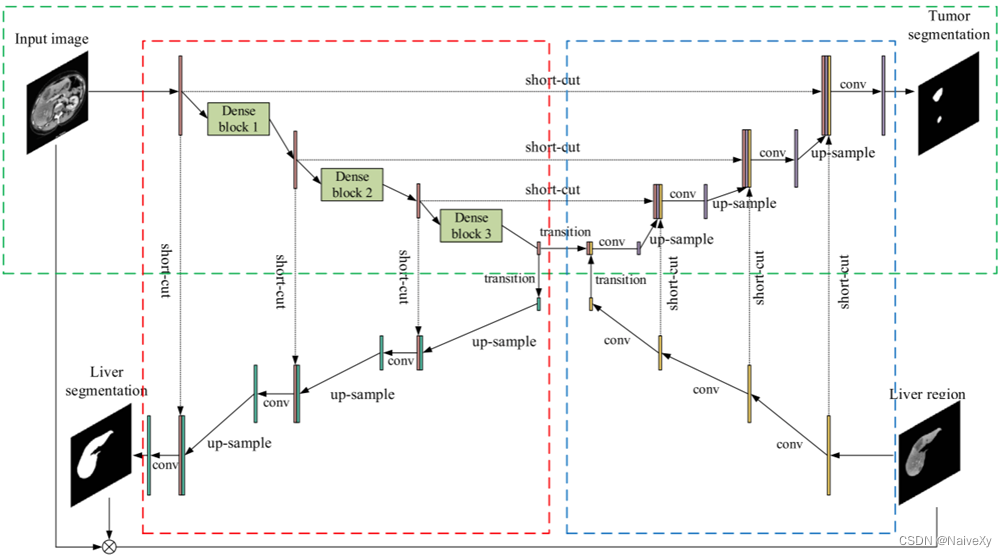
X-Net结构示意图:绿色矩形部分是主干DenseUNet分支,红色矩形部分是肝脏区域分割分支,蓝色矩形部分是肿瘤特征提取和分割分支。
用于切片间特征提取的 3D UNet
使用提出的 X-Net,我们可以表示每个CT切片中肝组织和肿瘤之间的差异和关系。然而,仅使用2D网络并不能完全提取CT体积沿z轴的空间信息,而3D卷积神经网络在实际应用中的计算成本相当大。为了更好地利用CT切片之间的上下文信息,我们提出了一种简化的3D-UNet来从堆叠的肝脏区域体积中提取切片间特征。
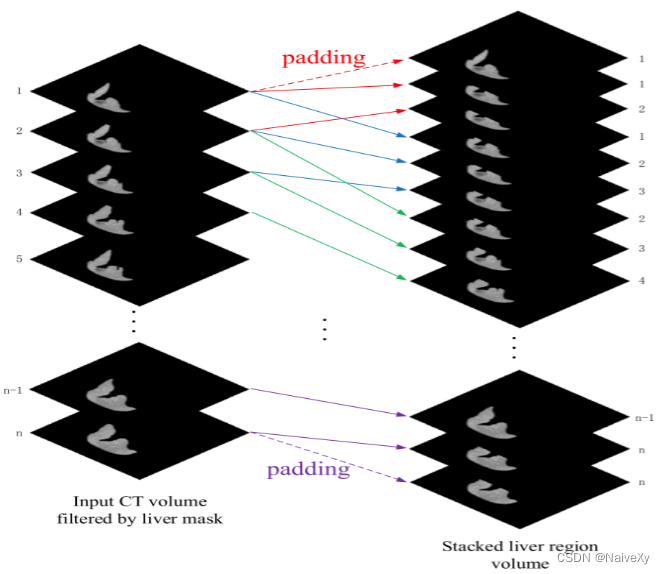
1.堆叠肝脏区域体积生成
由于我们专注于提取肝脏区域内的切片间特征,我们通过其输出肝脏Mask从X-Net分支过滤每个切片。然后使用H-DenseUNet中介绍的堆叠方法填充过滤后的CT体积。如右图所示,沿z维度的每三个相邻切片堆叠在一起,因此变换后的CT体积可以反映原始CT体积中每个切片之间的上下文信息。
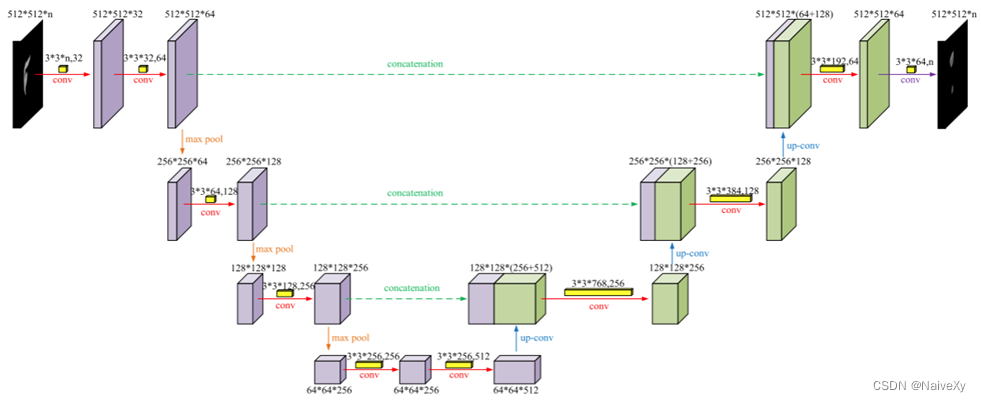
2.通过简化的3D-UNet提取切片间特征
用堆叠的CT体积作为输入,提出了一个3D卷积神经网络来提取表示相邻切片之间关系和差异的特征。网络的结构如下图。鉴于当前切片中某个邻域的切片间上下文信息主要由具有相同的邻域决定在相邻切片的中心坐标上,我们通过将大小为m*m*n的卷积核替换为大小为3*3*n的卷积核来简化传统3D UNet。它可以反映像素级的相关性和沿z轴的差异,在很大程度上抑制了来自远处相邻像素的影响,大大减轻了计算负担。因此,可以显着提高网络训练的效率。
3.片内和片间特征融合
使用相同的CT切片堆叠方法,X-Net从CT体积中的切片中提取的切片内特征图,其中肝脏区域内的特征已经被特征金字塔对应物突出显示,堆叠形成一个具有上下文信息的特征量。将片间特征量和片内特征量连接在一起。
区域和轮廓联合损失函数
肝脏和肿瘤预测分割的区域特征和轮廓特征共同用于抑制网络训练。由于肝脏和肿瘤的分割结果是3D体积,为了方便,将3D体积损失视为从分割体积的每个切片计算的2D切片损失的总和。
1.区域损失
对于区域损失部分,我们采用改进的Dice损失函数来衡量预测分割结果与Ground Truth之间的差异。传统的Dice损失函数仅用于那些图像中存在肿瘤和肝脏的阳性样本。对于那些负样本,即图像中没有肿瘤或肝脏,使用预测结果的L1范数作为Dice loss。
2.轮廓损失
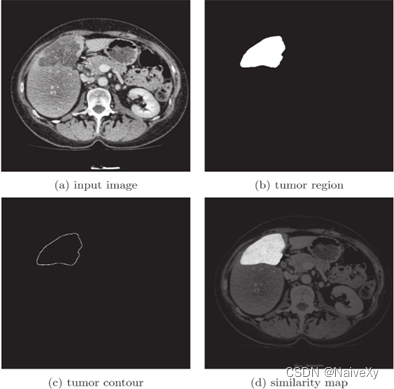
区域损失不能完全描述分割的形状,因此我们提出轮廓损失作为补充来训练网络。如右图所示,输入图像根据分割的轮廓(Ground Truth或预测)转换为轮廓相似度图。
(a)输入图像 (b)网络模型预测的肿瘤区域
(c)通过主动轮廓方法从分割的肿瘤区域中检测到的轮廓
(d)计算的相似度图跟随轮廓损失
对于LiTS数据集,使用dice per case score(DC)和dice global score (DG)来评估肝脏和肿瘤分割的性能,类似于最初2017 LiTS Challenge;DC是通过平均每个volume的Dice得分来定义的,而DG是指将volumes作为整体计算的Dice得分。
对于3DIRCAb 数据集,我们遵循H-DenseUNet中使用的指标来评估分割性能:体积重叠误差(VOE)、相对体积差(RVD)、平均对称表面距离(ASD)、均方根对称表面距离(RMSD)和Dice得分。前4个评价指标的值越小,分割性能越好。Dice得分与用于LiTS数据集的每个volume的Dice分数相同。





 本文提出了一种名为X-Net的多分支深度学习网络,用于从3D腹部CT扫描中精确分割肝脏和肿瘤。该网络结合了2D DenseUNet和3D UNet的优势,同时考虑了切片内和切片间的上下文信息。通过在DenseUNet中增加一个分支来估计肝脏区域,并利用肝脏特征来抑制肿瘤分割,提高了分割精度。此外,还引入了轮廓损失函数和修改后的Dice损失函数作为联合损失,以优化形状分割。实验结果显示,X-Net在肝脏和肿瘤分割任务上表现优越。
本文提出了一种名为X-Net的多分支深度学习网络,用于从3D腹部CT扫描中精确分割肝脏和肿瘤。该网络结合了2D DenseUNet和3D UNet的优势,同时考虑了切片内和切片间的上下文信息。通过在DenseUNet中增加一个分支来估计肝脏区域,并利用肝脏特征来抑制肿瘤分割,提高了分割精度。此外,还引入了轮廓损失函数和修改后的Dice损失函数作为联合损失,以优化形状分割。实验结果显示,X-Net在肝脏和肿瘤分割任务上表现优越。



















 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








