clc;clear
cd('C:\Users\29628\Desktop\nanjing\2')%选择你自己的文件夹
[A,R] = geotiffread('sentinel-2-0000000000-0000000000') ;%选择你的tif文件
img_r = A(:,:,4); % 获取红光波段
img_r(isnan(img_r) == 1) = 0;
R = mat2gray(img_r,[0,1]);
img_g = A(:,:,3); % 获取绿光波段
img_g(isnan(img_g) == 1) = 0;
G = mat2gray(img_g,[0,1]);
img_b = A(:,:,2); % 获取蓝光波段
img_b(isnan(img_b) == 1) = 0;
B = mat2gray(img_b,[0,1]);
img_nir = A(:,:,8); % 获取近红外波段
img_nir(isnan(img_nir) == 1) = 0;
NIR = mat2gray(img_nir,[0,1]);
NDVI = (NIR-R)./(NIR+R);
NDVI(isnan(NDVI) == 1) = 0;
NDVI = rescale(NDVI);
dr = stretchlim(NDVI);%自动选取范围,较手动选取会准
imshow(NDVI,dr)
colormap gray
在网上看了很多类似的,但复制过来都不行,自己就写了一个。以我的三脚猫功力解释一下这个代码读取数据时的NAN其实不管也行,但最后的图不太好看,就换成0了。NDVI是直接拿一个矩阵除以另一个矩阵就行了(开始还想着写俩很大的循环来着)目前只计算了NDVI,别的植被指数计算可以类比,就不写在这了。
有问题的地方是在输出NDVI的时候,如果不指定波段,会得到如下的结果(左边是matlab的,右边是ENVI计算的NDVI)会发现左边的灰了很多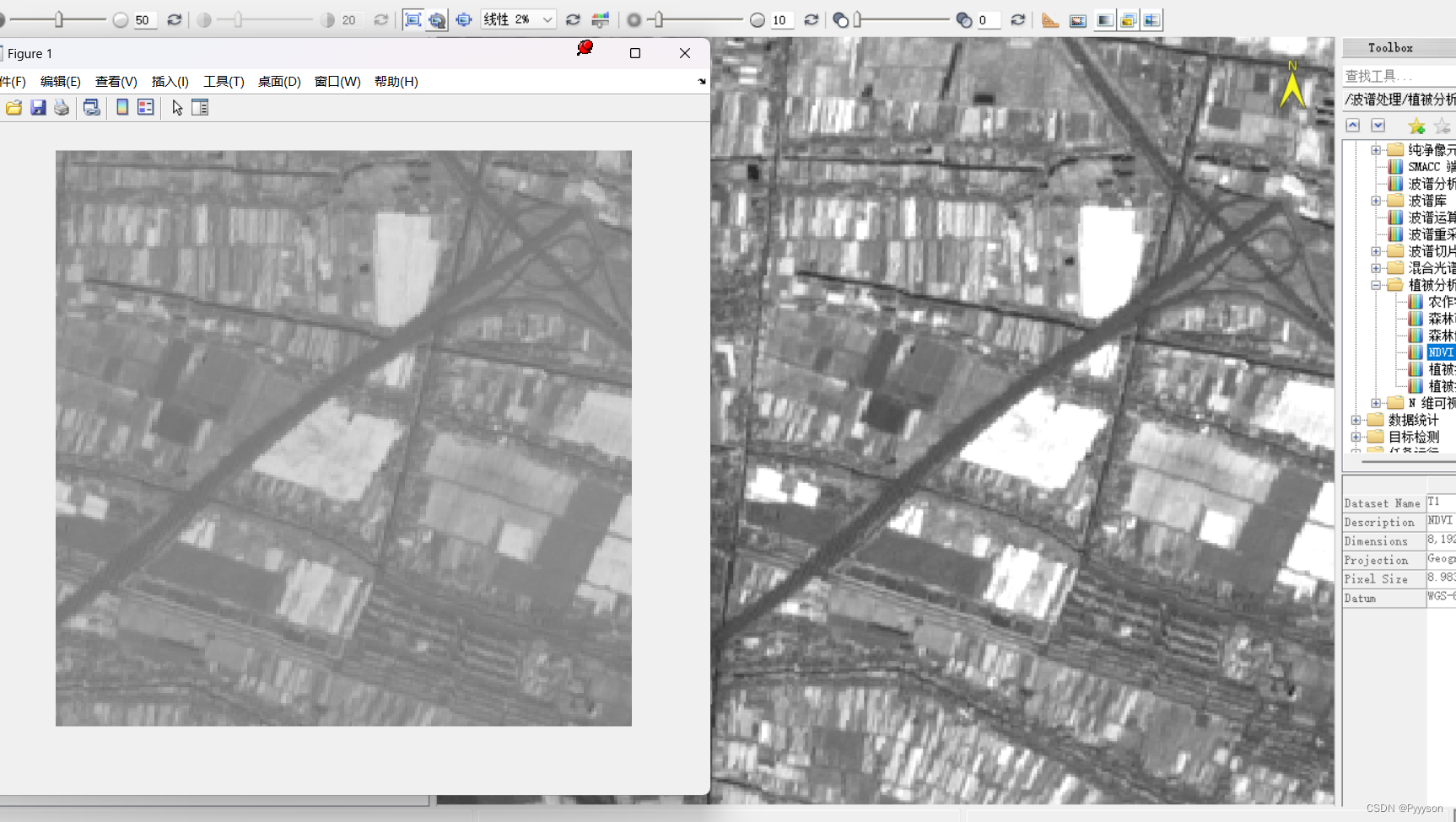
这是因为envi会自动调整对比度亮度这种,而matlab只是将原始结果展示出来,所以需要使用stretchlim函数为其自动指定范围,最后能得到以下的图(这一步花了我好长好长时间..)
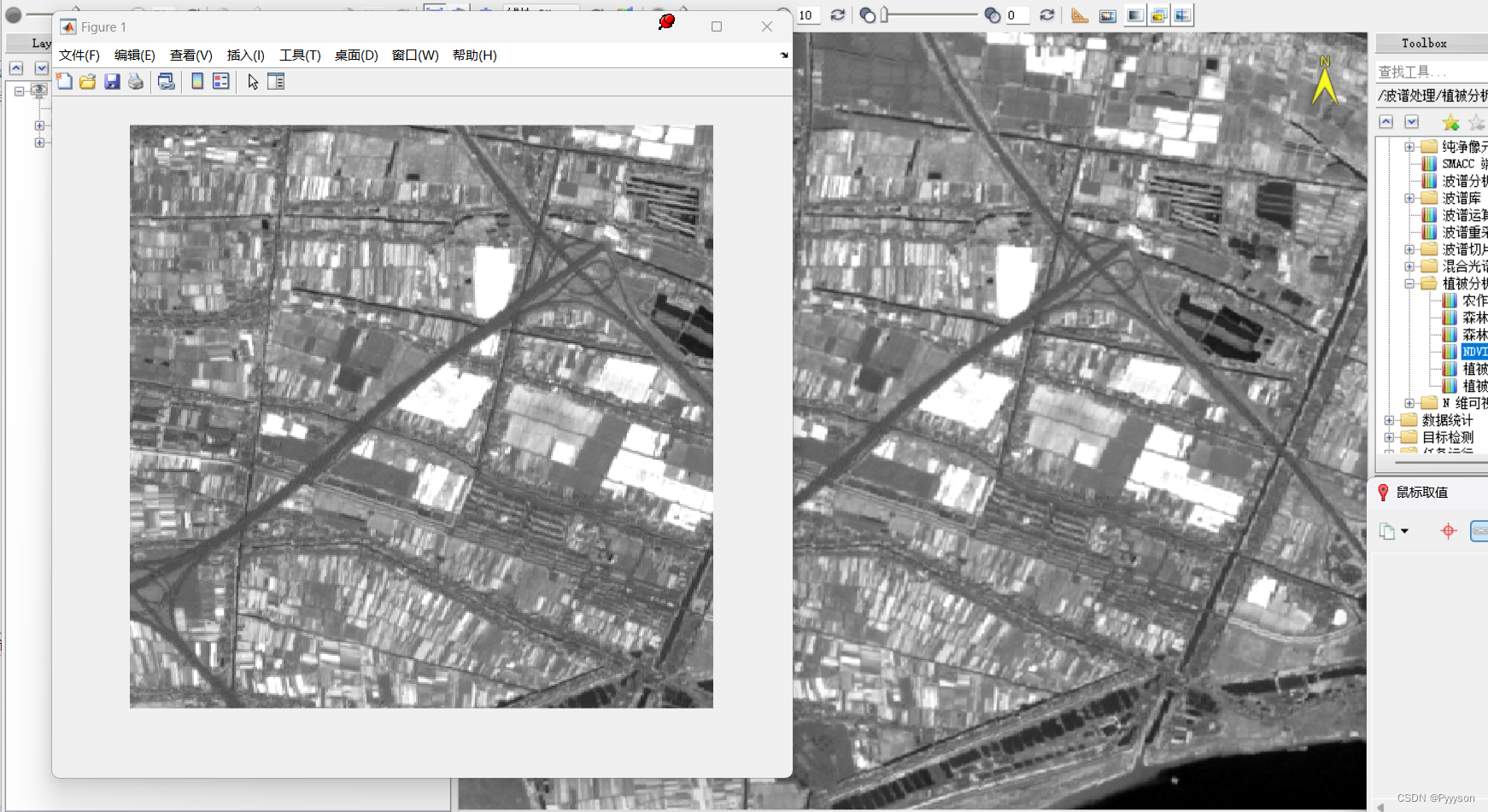




 该代码示例展示了如何在MATLAB中读取Geotiff图像,提取不同光谱波段,计算NDVI,并解决显示效果不理想的问题。通过使用mat2gray和stretchlim函数改善图像对比度。文章指出MATLAB默认显示与ENVI处理的NDVI图像存在差异,需调整对比度以获得更清晰的结果。
该代码示例展示了如何在MATLAB中读取Geotiff图像,提取不同光谱波段,计算NDVI,并解决显示效果不理想的问题。通过使用mat2gray和stretchlim函数改善图像对比度。文章指出MATLAB默认显示与ENVI处理的NDVI图像存在差异,需调整对比度以获得更清晰的结果。
















 706
706










