最近在做一个膝关节损伤检测项目,采用的是Stanford大学的MRNet-v1.0公开数据集,该数据集包含了1130例膝关节的轴向、冠状面、矢状三个方向的磁共振(MRI)切片数据。下载的数据格式均为数组(.npy),而我需要的数据格式为图像(.jpg\.png)于是写了一个npy2img的脚本。有需要的自取,但引用需备注出处。
#by Chongqing University LiJie 2025.8.1
import numpy as np
from PIL import Image
import os # 新增:用于文件路径和目录操作
# 批量转换配置
input_dir = 'D:/2DVision/MRNet-v1.0/valid/axial/' # 输入.npy文件所在目录
output_root = 'D:/2DVision/MRNet-v1.0/valid_img/axial/' # 输出图片根目录
# 获取目录下所有.npy文件
npy_files = [f for f in os.listdir(input_dir) if f.endswith('.npy')]
if not npy_files:
print("未找到任何.npy文件")
exit()
# 遍历每个.npy文件进行批量处理
for npy_file in npy_files:
# 构建完整文件路径
npy_path = os.path.join(input_dir, npy_file)
# 提取文件名(不含扩展名)作为子目录名
file_name = os.path.splitext(npy_file)[0]
# 创建输出子目录(每个.npy文件的切片单独存放)
output_dir = os.path.join(output_root, file_name)
os.makedirs(output_dir, exist_ok=True) # 自动创建目录,已存在则忽略
# 加载.npy文件数据
imgs_data = np.load(npy_path)
print(f"正在处理文件: {npy_file},数据形状: {imgs_data.shape}")
# 处理并保存当前文件的所有切片
for slice_idx in range(imgs_data.shape[0]):
slice_data = imgs_data[slice_idx, :, :]
# 保存路径:输出子目录/切片索引.png
save_path = os.path.join(output_dir, f"{slice_idx}.png")
Image.fromarray(slice_data).save(save_path)
print(f"文件 {npy_file} 处理完成,共生成 {imgs_data.shape[0]} 张切片\n")
print("所有.npy文件批量转换完成!")
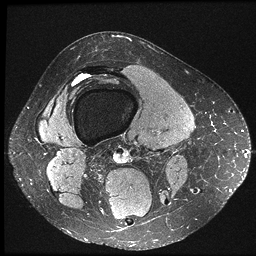
数据转换后的效果如下:






















 3026
3026

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








