准备
首先,我是从GRETNA的Github官网上下载的GRETNA-master这个版本。
其次,安装的方法就是解压,然后把解压的文件夹添加到matlab的路径中,注意路径不要有中文,特殊符号或者空格。
第三,我没有使用GRETNA来做数据的预处理。作为参考我的预处理是通过SPM12和DPARSF来完成的。最后使用的文件夹FunImgARWSDCF中每个被试一个文件夹,一个文件夹中一个Filtered_4DVolume.nii文件。
开始使用
在matlab的命令窗口输入gretna打开GRETNA界面。
第一个尝试:
一开始我用了DPARSF来对SPM预处理到smooth阶段的数据进行去线性漂移detrend,回归协变量,以及头动校正和滤波Filter的操作。然后使用滤波后的数据放入GRETNA进行网络的建立,但是出现了错误。修bug不如重写,于是我后面改了方法,不过这一步尝试还是可以先看看。
选择第一个FC Matrix Construct,开始创建功能连接的矩阵。我没有使用自带的模板,而是下载了一个BN_Atlas_246_3mm.nii这个模板,分成皮层和皮层下共246个区域。
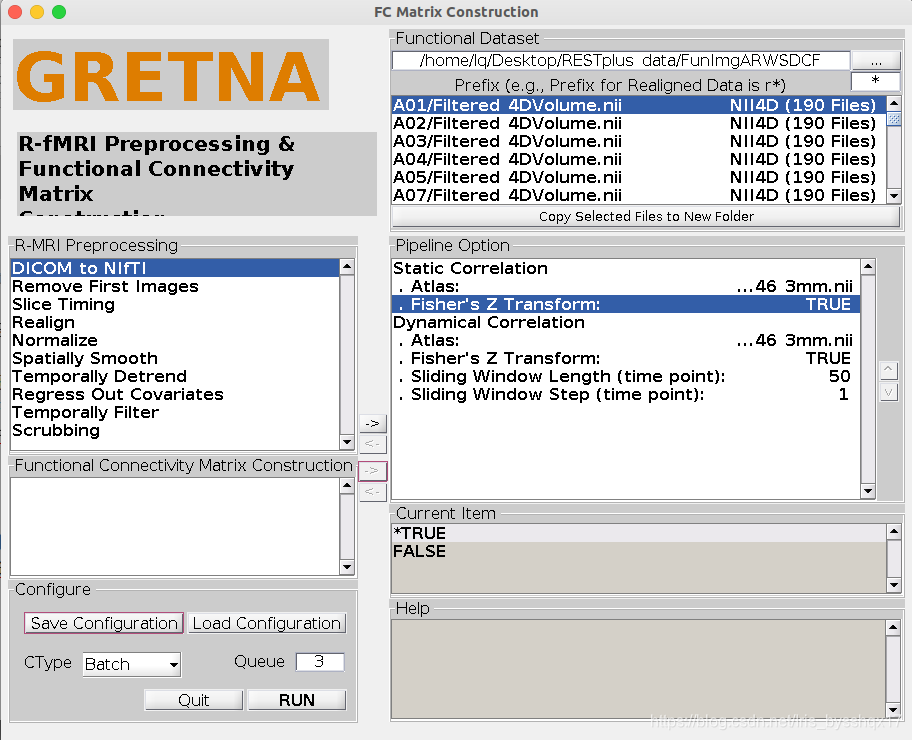 注意:然后我RUN了一下,出了第一个错误“/bin/bash: at: Command not found”,原因是没有找到at这个命令,于是
注意:然后我RUN了一下,出了第一个错误“/bin/bash: at: Command not found”,原因是没有找到at这个命令,于是sudo apt-fast install at安装一下。其中apt-fast时apt-get的一种快速的版本,安装方法可以自行搜索。如果不使用apt-fast,也可以直接sudo apt-get install at来安装at。
第二个尝试:
由于直接使用DPARSF的Filtered数据再GRETNA中出现了错误,为了免去麻烦,我直接使用了GRETNA中的数据预处理pipeline来进行数据预处理。
数据结构:
workingdir文件夹下有一个功能像文件夹FunImg一个结构像文件夹Struc。
功能像文件夹FunImg文件夹下每个被试一个文件夹,包含删去前10个时间点的原始Nifti格式文件。
结构像文件夹Struc文件夹中同样每个被试一个文件夹,包含一个结构像Nifti格式文件和一个在SPM12中修正过AC位置和图像朝向的mat文件。
注意:文件夹的命名不是固定的,但是文件夹的结构最好是这样子的。结构像是否需要修改朝向需要仔细查看。
步骤1:数据预处理:
1.时





 本文详细介绍了如何使用GRETNA进行fMRI数据处理,包括数据预处理、功能连接网络构建、网络属性分析及比较。通过实例展示了GRETNA在静态和动态功能网络构建中的应用,并进行了全局和局部属性的统计分析,结果表明在所选指标上未观察到显著差异。
本文详细介绍了如何使用GRETNA进行fMRI数据处理,包括数据预处理、功能连接网络构建、网络属性分析及比较。通过实例展示了GRETNA在静态和动态功能网络构建中的应用,并进行了全局和局部属性的统计分析,结果表明在所选指标上未观察到显著差异。
 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章

















 2万+
2万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








