download网址:https://sourceforge.net/projects/itk-snap/
网页界面:
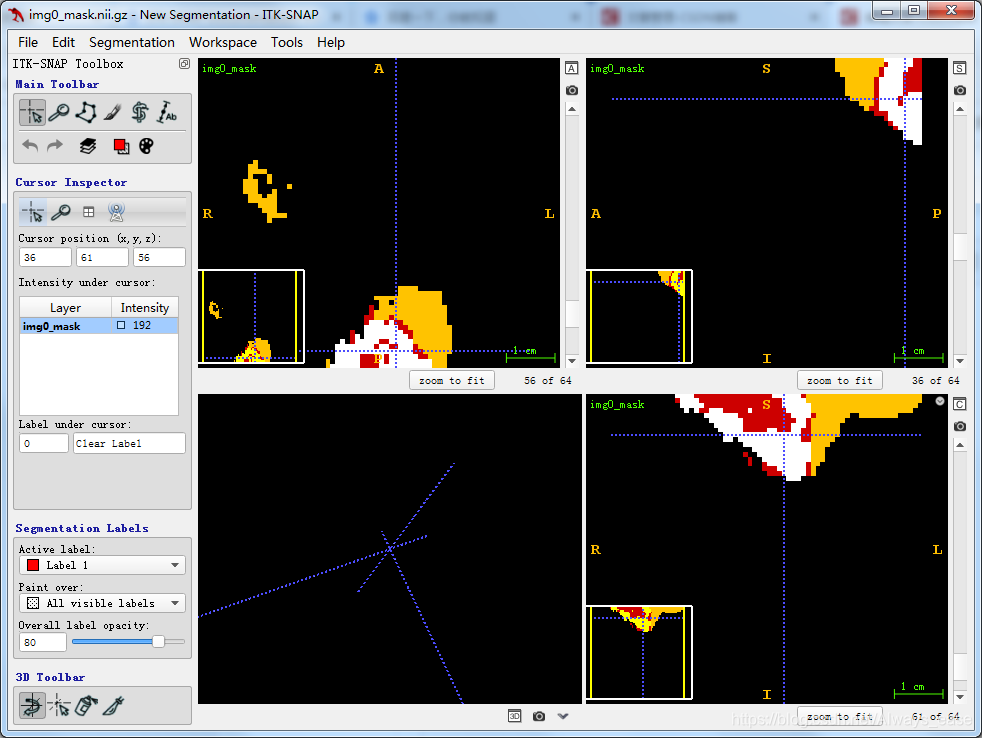
显示3d的操作方式
1)保存原始图像、预测图像、mask的nii文件
def colorize(n, images, logits, targets):
outputs = torch.argmax(logits, dim=1, keepdim=False).type(torch.uint8)
targets = targets.type(torch.uint8)
outputs = np.array(outputs[0,...]) # choose one to show
targets = np.array(targets[0,...])
img = sitk.GetImageFromArray(outputs * 64)
sitk.WriteImage(img, root + 'img' + str(n) + '_src.nii.gz')
out = sitk




 ITK-SNAP是一款强大的3D图像处理软件,可用于显示和操作3D图像。用户可以下载该软件并按照指导进行操作,如保存nii文件、导入主图像和segmentation,通过继续更新进行图像处理。
ITK-SNAP是一款强大的3D图像处理软件,可用于显示和操作3D图像。用户可以下载该软件并按照指导进行操作,如保存nii文件、导入主图像和segmentation,通过继续更新进行图像处理。
 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章

















 2427
2427

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








