数据准备。测序完成后得到md5.txt用于质量检测数据。以及R1forward,R2reverse双侧rawdata。上传至rawdata工作路径
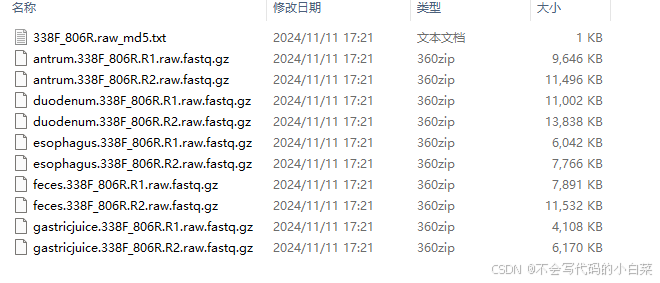
编写两个文件,第一个无文件格式的sample;第二个是txt格式的new.txt。所有编写以“tab”键输入空格。上传至工作路径
1.第一列为样本名称。第二列为前端数据的绝对路径。第三列为后端数据的绝对路径
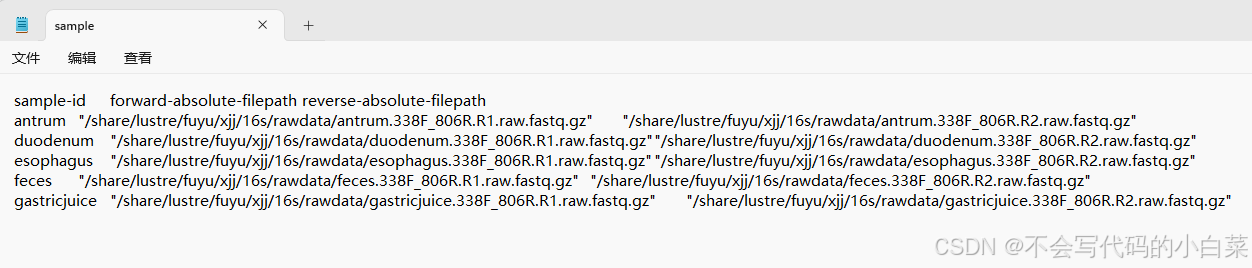
2.第一列为样本名称,应与1中的对应。中间输入三个“Tab”空格,然后第四列name为病人姓名。第五列site为样本的部位。第六列name-site为姓名+部位组合
上传完成后下面开始正式的Linux系统操作。
一.进入工作路径,进入qiime环境
cd /share/lustre/fuyu/xjj/16s/rawdata
mamba activate qiime2-2023.5
二.检查文件的完整性
md5sum -c "/share/lustre/fuyu/xjj/16s/rawdata/338F_806R.raw_md5.txt"
三. 数据导入
qiime tools import --type 'SampleData[PairedEndSequencesWithQuality]' --input-path sample --output-path paired-demux.qza --input-format PairedEndFastqManifestPhred33V2
四. 去除引物
qiime cutadapt trim-paired --i-demultiplexed-sequences paired-demux.qza --p-front-f CCTAYGGGRBGCASCAG --p-front-r GGACTACHVGGGTWTCTAAT --o-trimmed-sequences paired-end-demux.qza --verbose
若数据为单端数据,则将三四步骤替换为这段代码(单端为cleandata)
qiime tools import --type 'SampleData[SequencesWithQuality]' --input-path sample --output-path single-end-demux.qza --input-format SingleEndFastqManifestPhred33V2
五. 数据可视化
qiime demux summarize --i-data paired-end-demux.qza --o-visualization paired-end-demux.qzv
六.质量过滤
qiime dada2 denoise-single --i-demultiplexed-seqs paired-end-demux.qza --p-trim-left 0 --p-trunc-len 0 --p-n-threads 20 --o-representative-sequences representative-sequences.qza --o-table table.qza --o-denoising-stats denoising-stats.qza
七.将六的数据可视化
qiime feature-table summarize --i-table table.qza --o-visualization table.qzv
qiime metadata tabulate --m-input-file denoising-stats.qza --o-visualization denoising-stats.qzv
qiime feature-table tabulate-seqs --i-data representative-sequences.qza --o-visualization representative-sequences.qzv
八.绘制系统发育树
qiime phylogeny align-to-tree-mafft-fasttree --i-sequences representative-sequences.qza --o-alignment aligned-rep-seqs.qza --o-masked-alignment masked-aligned-rep-seqs.qza --o-tree unrooted-tree.qza --o-rooted-tree rooted-tree.qza
九. 计算核心多样性。depth参数按照table.qzv中最小值输入。若最小值差距太大,则删除异常值重新选择最小值
qiime diversity core-metrics-phylogenetic --i-phylogeny rooted-tree.qza --i-table table.qza --p-sampling-depth 27164 --m-metadata-file new.txt --output-dir core-metrics-result
十.Alpha 稀释曲线
qiime diversity alpha-rarefaction --i-table table.qza --i-phylogeny rooted-tree.qza --p-max-depth 40993 --m-metadata-file new.txt --o-visualization alpha-rarefaction50000.qzv
十一.物种组成分析
qiime feature-classifier classify-sklearn --i-classifier /share/lustre/fuyu/xxy/qiime2/database/gg_2022_10_backbone_full_length.nb.qza --i-reads representative-sequences.qza --o-classification taxonomy.qza
十二.用交互式条形图查看样本的分类组成
qiime taxa barplot --i-table table.qza --i-taxonomy taxonomy.qza --m-metadata-file new.txt --o-visualization taxa-bar-plots.qzv




















 4642
4642

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








