思路来源&致谢 :https://www.kaggle.com/jesucristo/1-house-prices-solution-top-1?scriptVersionId=12846740
git 地址: https://github.com/jamjar102/kaggle_HousePrice new.py文件跑出来的submission score大概0.11 达到top5%
具体思路可以看kaggle的这篇教程和我代码上的一些注释,以下大概写几个之前没有用过的技术点。
记性不好,写下来自己看防止以后忘记。
一、数据分析
1.标签分布:
sns.distplot(y, kde=False, fit=stats.johnsonsu)
sns.distplot(y, kde=False, fit=stats.norm)
sns.distplot(y, kde=False, fit=stats.lognorm)其中y为标签序列,采用不同的fit函数可以用不同的分布去进行拟合。
2.stats.shapiro()
test_normality = lambda x: stats.shapiro(x.fillna(0))[1] < 0.01 #统计学知识
输出结果中第一个为统计量,第二个为P值(统计量越接近1越表明数据和正态分布拟合的好,P值大于指定的显著性水平,接受原假设,认为样本来自服从正态分布的总体)
以下来自scipy官网:
The Shapiro-Wilk test tests the null hypothesis that the data was drawn from a normal distribution.
该检测的无效假设是该分布符合正态分布。对于N>5000的样本可能会造成p值不准确
以下致谢这篇优快云文章:https://blog.youkuaiyun.com/weixin_42059534/article/details/101703027
'''输出结果中第一个为统计量,第二个为P值(统计量越接近1越表明数据和正态分布拟合的好,
P值大于指定的显著性水平,接受原假设,认为样本来自服从正态分布的总体)''' 相当于P值越小越拒绝原假设(拒绝无效假设)
print(stats.shapiro(a))
kstest的原假设是两个分布相同,所以p值大于阈值可以接收
'''输出结果中第一个为统计量,第二个为P值(注:统计量越接近0就越表明数据和标准正态分布拟合的越好,
如果P值大于显著性水平,通常是0.05,接受原假设,则判断样本的总体服从正态分布)'''
print(stats.kstest(a, 'norm'))
'''输出结果中第一个为统计量,第二个为P值(注:p值大于显著性水平0.05,认为样本数据符合正态分布)'''
print(stats.normaltest(a))
为什么p值越小约拒绝原假设:https://www.zhihu.com/question/35891708
个人理解:
相当于认为此分布为正态分布,正态分布有对应的各种情况出现的概率,然后去进行抽取实验或者什么实验去验证,但是抽出来的结果是十分极端的(在原假设下概率出现的十分低),概率低,p值低,所以拒绝原假设,即不认为当前数据的分布为正态分布。
3. 对离散型变量进行encode
def encode(frame, feature):
ordering = pd.DataFrame()
ordering['val'] = frame[feature].unique() #数组返回该特征里面属性的唯一值(去重)
ordering.index = ordering.val
ordering['spmean'] = frame[[feature, 'SalePrice']].groupby(feature).mean()['SalePrice']
ordering = ordering.sort_values('spmean')
ordering['ordering'] = range(1, ordering.shape[0] + 1)
ordering = ordering['ordering'].to_dict()
for cat, o in ordering.items():
frame.loc[frame[feature] == cat, feature + '_E'] = o
#print(frame)
qual_encoded = []
for q in qualitative: #离散型特征名
encode(train, q)
qual_encoded.append(q + '_E')
print(qual_encoded) #处理后的数值特征名 + _E其中 frame.loc[frame[feature] == cat, feature + '_E'] = o
参考官网教程:https://pandas.pydata.org/pandas-docs/stable/reference/api/pandas.DataFrame.loc.html


4.查看每列feature对标签的斯皮尔曼相关系数
def spearman(frame, features):
spr = pd.DataFrame()
spr['feature'] = features
spr['spearman'] = [frame[f].corr(frame['SalePrice'], 'spearman') for f in features]
#这块调用的是https://pandas.pydata.org/pandas-docs/stable/reference/api/pandas.Series.corr.html
#series中的corr方法,第一个参数接收一个series参数
spr = spr.sort_values('spearman') #排序
#plt.figure(figsize=(6, 0.25 * len(features)))
#sns.barplot(data=spr, y='feature', x='spearman', orient='h')
#plt.show()
features = quantitative + qual_encoded
spearman(train, features)其中:
spr['spearman'] = [frame[f].corr(frame['SalePrice'], 'spearman') for f in features] #这块调用的是https://pandas.pydata.org/pandas-docs/stable/reference/api/pandas.Series.corr.html #series中的corr方法,第一个参数接收一个series参数

5.查看特征相关系数,发现之前对特征进行encode之后可以直线增加相关性
# 查看相关稀疏矩阵
corr = train[qual_encoded + ['SalePrice']].corr()
sns.heatmap(corr)
#plt.show()
corr = train[quantitative + ['SalePrice']].corr()
sns.heatmap(corr)
#plt.show()
corr = pd.DataFrame(np.zeros([len(quantitative) + 1, len(qual_encoded) + 1]), index=quantitative + ['SalePrice'],
columns=qual_encoded + ['SalePrice'])
for q1 in quantitative + ['SalePrice']:
for q2 in qual_encoded + ['SalePrice']:
corr.loc[q1, q2] = train[q1].corr(train[q2])
sns.heatmap(corr)直接使用以下两行代码即可查看dataframe的相关性
corr = train[qual_encoded + ['SalePrice']].corr()
sns.heatmap(corr)
6. TSNE()特征降维
致谢:https://www.cnblogs.com/bonelee/p/7849867.html
假设数据是均匀采样于一个高维欧氏空间中的低维流形,流形学习就是从高维采样数据中恢复低维流形结构,即找到高维空间中的低维流形,并求出相应的嵌入映射,以实现维数约简或者数据可视化。它是从观测到的现象中去寻找事物的本质,找到产生数据的内在规律。
简单地理解,流形学习方法可以用来对高维数据降维,如果将维度降到2维或3维,我们就能将原始数据可视化,从而对数据的分布有直观的了解,发现一些可能存在的规律
model = TSNE(n_components=2, random_state=0, perplexity=50)
_components,int, default=2,means:Dimension of the embedded space.
对原始特征进行降维,备用。对原始特征进行标准化后进行PCA,PCA后进行聚类,按类别查看降维后(此处是2维)后的相关性
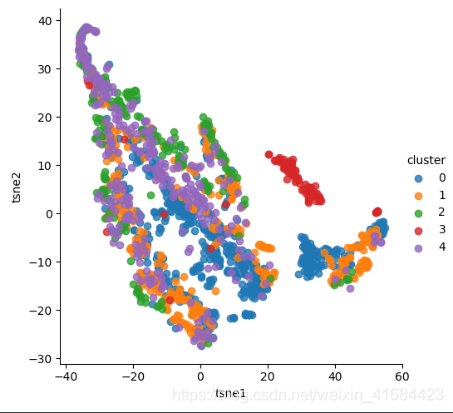
超级todo:进行了这些特征分析后对算法有什么帮助与提升呢?
二.数据处理
1.利用log1p和exp1p对数据进行平滑和恢复
train["SalePrice"] = np.log1p(train["SalePrice"]) #数据平滑处理
2.数据清洗后需要重新排序,X和y都要排序
y = train['SalePrice'].reset_index(drop=True)
3.dataframe.groupby
features['MSZoning'] = features.groupby('MSSubClass')['MSZoning'].transform(lambda x: x.fillna(x.mode()[0]))
致谢:https://www.cnblogs.com/bjwu/p/8970818.html
4.dataframe.update()
5.去掉0太多的特征列
#0太多的去掉
overfit = []
for i in X.columns:
counts = X[i].value_counts()
zeros = counts.iloc[0]
if zeros / len(X) * 100 > 99.94:
overfit.append(i)
overfit = list(overfit)
X = X.drop(overfit, axis=1)
X_sub = X_sub.drop(overfit, axis=1)
三、Model
大体:参见代码
1.RobustScaler()

致谢:
https://blog.youkuaiyun.com/u013749051/article/details/105870716
2.几个用到的回归&之间的关系:
线性回归的目的是要得到输出向量Y和输入特征X之间的线性关系,求出线性回归系数θ,也就是Y=Xθ, 其中Y的维度为m x 1,X的维度为 m x n,而θ的维度为 n x 1, m代表样本个数, n代表样本特征的维度
RidgeCV(岭回归):只要数据线性相关,用LinearRegression拟合的不是很好,需要正则化,可以考虑使用RidgeCV回归, 如何输入特征的维度很高,而且是稀疏线性关系的话, RidgeCV就不太合适,考虑使用Lasso回归类家族
LassoCV:Lasso回归可以使得一些特征的系数变小,甚至还使一些绝对值较小的系数直接变为0,从而增强模型的泛化能力
使用场景:对于高纬的特征数据,尤其是线性关系是稀疏的,就采用Lasso回归,或者是要在一堆特征里面找出主要的特征,那么 Lasso回归更是首选了
致谢:https://www.jianshu.com/p/88cb43166bae
3.regressor.StackingCVRegressor
StackingCVRegressor扩展了使用Stacking预测的标准Stacking算法(实现为StackingRegressor),预测的结果作为2级分类器的输入数据。
在标准 stacking程序中,拟合一级回归器的时候,我们如果使用第二级回归器的输入的相同训练集,这很可能会导致过度拟合。 然而,StackingCVRegressor使用了‘非折叠预测’的概念:数据集被分成k个折叠,并且在k个连续的循环中,使用k-1折叠来拟合第一级回归器(即k折交叉验证的stackingRegressor)。
在每一轮中(一共K轮),一级回归器然后被应用于在每次迭代中还未用过的模型拟合的剩余1个子集。
然后将得到的预测叠加起来并作为输入数据提供给二级回归器。在StackingCVRegressor的训练完之后,一级回归器拟合整个数据集以获得最佳预测。
致谢:https://blog.youkuaiyun.com/zhuiqiuuuu/article/details/84866502

























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








