原论文地址:
U-Net: Convolutional Networks for Biomedical Image Segmentation
Pytorch 实现: https:// github.com/milesial/Pyt orch-UNet
一、U-Net 概述
U-Net 作为一个图像语义分割网络,提出时主要用于对医学图像进行处理。深度学习用于医学影像处理的一个挑战在于,提供的样本往往比较少,而 U-Net 则在这个限制下依然有很好的表现:
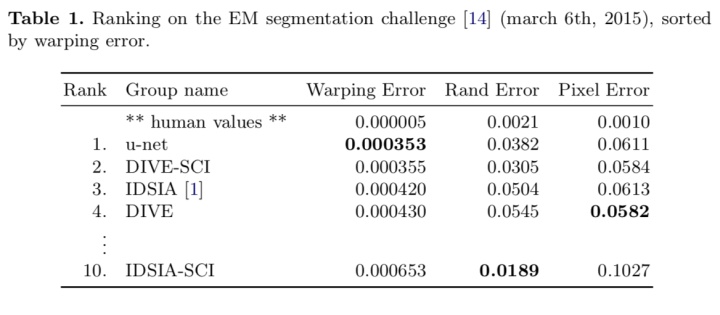
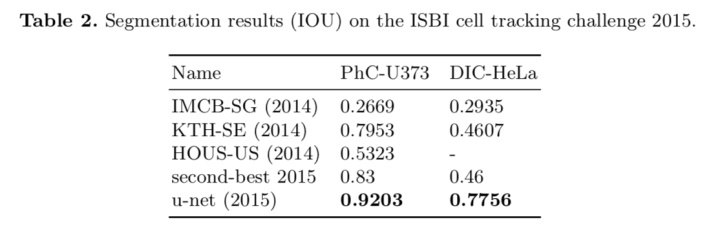
U-Net 是怎么做到的呢?接下来介绍一下 U-Net 的结构:
二、U-Net 模型结构介绍
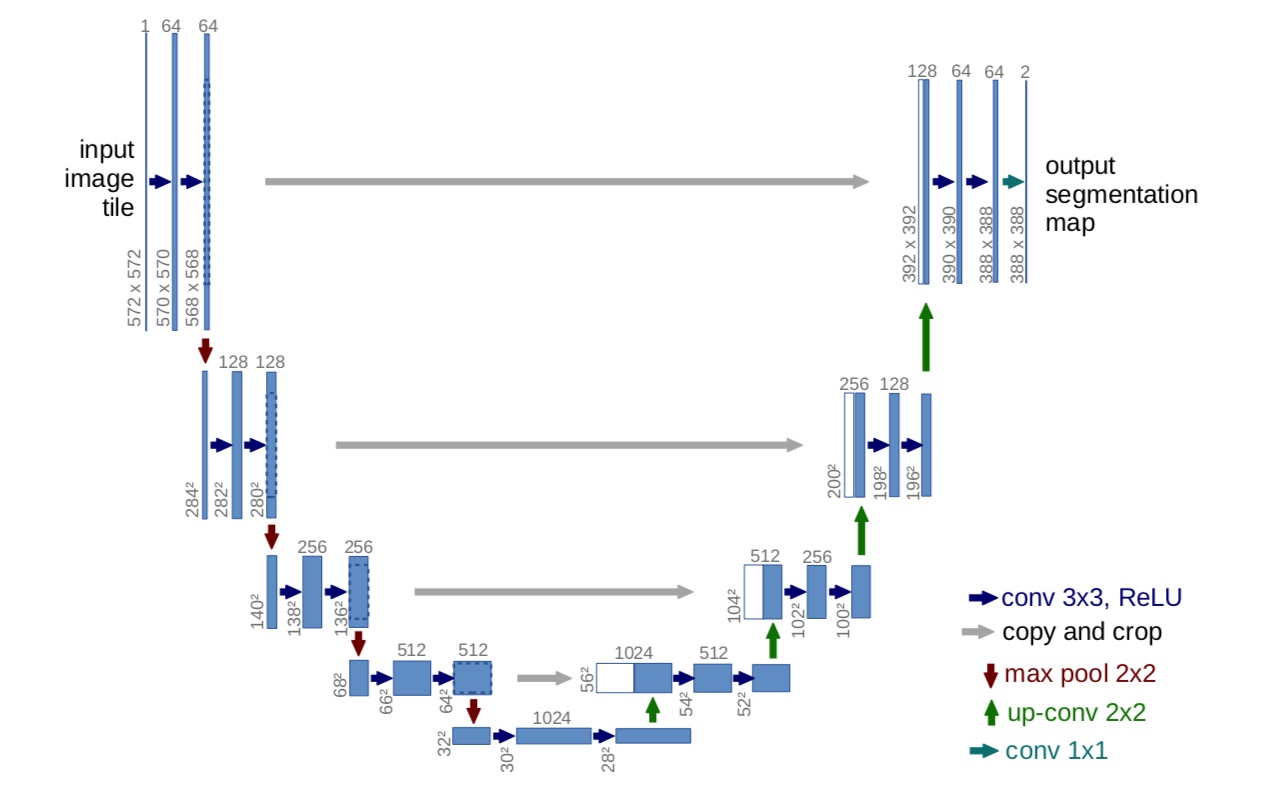
看下图便知 U-Net 模型名字的来由:
不好意思,放错图了:

U-Net 整个流程为 U 型,左边的为下采样过程,右边为上采样过程,中间的灰色箭头是将特征图进行跳层联结,其原理和 DenseNet 相同,即 concatenate ,torch.cat([x1,x2])。可以将浅层的定位信息和高层的像素分类判定信息进行融合,从而得到更佳的结果。
除此之外,U-Net 有几点值得注意的地方:
- 卷积层 Padding = 0,所以每次做卷积,特征图的大小都会 -2。
- 特征提取的卷积层都为
大小。
- 下采样使用 max-pooling
- 上采样使用步长为 2 的反卷积
- 最后的分类使用
的卷积层
为啥卷积层的 Padding = 0 呢?主要原因在于,医学影像的分辨率一般比较大,模型是无法一次性在GPU上计算完成的,会爆显存。
咋办呢?分块呗。
小编在也曾遇到类似的问题,最终的解决思路也和 U-Net 论文类似:
Uno Whoiam:心中无码,自然高清 || 联合去马赛克与超分辨率研究论文Pytorch复现
这是一个基于深度残差网络的超分辨率模型,可以把数码相机拍摄的 bayer 图像超分辨率化为正常的 RGB 图像。要知道,这类图片基本是千万像素级别,放到 GPU 里分分钟保显存,咋整?
切成一块块,用模型跑完后拼起来呗!
然而,拼起来后:

拼接缝隙过于明显啊。这可咋办?
很简单,切大块,但只取中间的部分进行拼接,边边角角的部分的话,则采用镜像 Padding 扩大一圈。
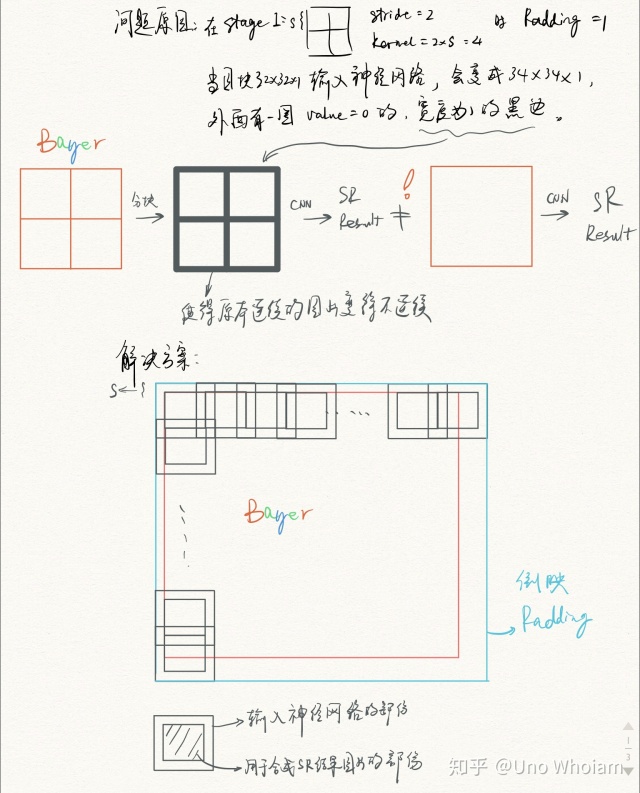
而 U-Net 的做法也是如此,这样就可以保证图片没有拼接痕迹:
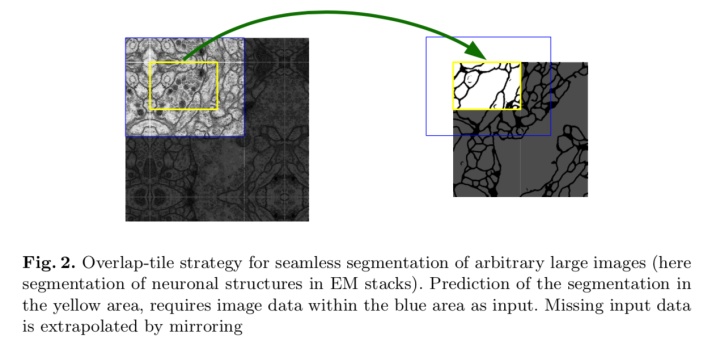
更进一步的,如上文所提到的,卷积层 Padding = 0,其实也是为这个目标服务的。
Pytorch 实现(和原版稍有不同,卷积有Padding):
import torch
import torch.nn as nn
import torch.nn.functional as F
# https://github.com/milesial/Pytorch-UNet
# full assembly of the sub-parts to form the complete net
class UNet(nn.Module):
def __init__(self, n_channels, n_classes):
super(UNet, self).__init__()
self.inc = inconv(n_channels, 64)
self.down1 = down(64, 128)
self.down2 = down(128, 256)
self.down3 = down(256, 512)
self.down4 = down(512, 512)
self.up1 = up(1024, 256)
self.up2 = up(512, 128)
self.up3 = up(256, 64)
self.up4 = up(128, 64)
self.outc = outconv(64, n_classes)
def forward(self, x):
x1 = self.inc(x)
x2 = self.down1(x1)
x3 = self.down2(x2)
x4 = self.down3(x3)
x5 = self.down4(x4)
x = self.up1(x5, x4)
x = self.up2(x, x3)
x = self.up3(x, x2)
x = self.up4(x, x1)
x = self.outc(x)
return F.sigmoid(x)
# sub-parts of the U-Net model
class double_conv(nn.Module):
'''(conv => BN => ReLU) * 2'''
def __init__(self, in_ch, out_ch):
super(double_conv, self).__init__()
self.conv = nn.Sequential(
nn.Conv2d(in_ch, out_ch, 3, padding=1),
nn.BatchNorm2d(out_ch),
nn.ReLU(inplace=True),
nn.Conv2d(out_ch, out_ch, 3, padding=1),
nn.BatchNorm2d(out_ch),
nn.ReLU(inplace=True)
)
def forward(self, x):
x = self.conv(x)
return x
class inconv(nn.Module):
def __init__(self, in_ch, out_ch):
super(inconv, self).__init__()
self.conv = double_conv(in_ch, out_ch)
def forward(self, x):
x = self.conv(x)
return x
class down(nn.Module):
def __init__(self, in_ch, out_ch):
super(down, self).__init__()
self.mpconv = nn.Sequential(
nn.MaxPool2d(2),
double_conv(in_ch, out_ch)
)
def forward(self, x):
x = self.mpconv(x)
return x
class up(nn.Module):
def __init__(self, in_ch, out_ch, bilinear=True):
super(up, self).__init__()
# would be a nice idea if the upsampling could be learned too,
# but my machine do not have enough memory to handle all those weights
if bilinear:
self.up = nn.Upsample(scale_factor=2, mode='bilinear', align_corners=True)
else:
# torch.nn.ConvTranspose1d(in_channels, out_channels, kernel_size, stride=1,
# padding=0, output_padding=0, groups=1, bias=True, dilation=1)
self.up = nn.ConvTranspose2d(in_ch // 2, in_ch // 2, 2, stride=2)
self.conv = double_conv(in_ch, out_ch)
def forward(self, x1, x2):
x1 = self.up(x1)
# input is CHW
diffY = x2.size()[2] - x1.size()[2]
diffX = x2.size()[3] - x1.size()[3]
x1 = F.pad(x1, (diffX // 2, diffX - diffX // 2,
diffY // 2, diffY - diffY // 2))
# for padding issues, see
# https://github.com/HaiyongJiang/U-Net-Pytorch-Unstructured-Buggy/commit/0e854509c2cea854e247a9c615f175f76fbb2e3a
# https://github.com/xiaopeng-liao/Pytorch-UNet/commit/8ebac70e633bac59fc22bb5195e513d5832fb3bd
x = torch.cat([x2, x1], dim=1)
x = self.conv(x)
return x
class outconv(nn.Module):
def __init__(self, in_ch, out_ch):
super(outconv, self).__init__()
self.conv = nn.Conv2d(in_ch, out_ch, 1)
def forward(self, x):
x = self.conv(x)
return x
三、U-Net 的训练
U-Net 的训练和其它模型没啥大的区别,它的目标是识别出细胞,而细胞之间的边界则是 U-Net 重点关注的的对象,用 softmax + crossentropy 分类。值得注意的是,对细胞之间的边界的像素,U-Net 会对其特别关照,损失函数设计如下:
即加了权重的交叉熵,权重

另外值得一提的是,在数据增强上,使用了弹性形变,这和细胞的特性一致:
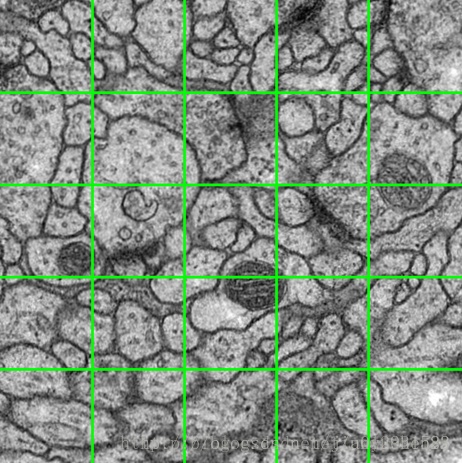

U-Net 在GPU上只需训练十小时,可以说比较快的了。
PS:
广告时间啦~
理工狗不想被人文素养拖后腿?不妨关注微信公众号:








 本文介绍了U-Net,一种在有限样本情况下仍能实现高精度医学影像语义分割的深度学习模型。U-Net模型因其U型结构得名,通过结合浅层的定位信息和深层的像素分类信息。文章详细阐述了模型结构,包括卷积层、下采样和上采样的方式,并讨论了如何应对大分辨率图像的内存问题。此外,还提及了训练过程中的损失函数设计和数据增强策略。
本文介绍了U-Net,一种在有限样本情况下仍能实现高精度医学影像语义分割的深度学习模型。U-Net模型因其U型结构得名,通过结合浅层的定位信息和深层的像素分类信息。文章详细阐述了模型结构,包括卷积层、下采样和上采样的方式,并讨论了如何应对大分辨率图像的内存问题。此外,还提及了训练过程中的损失函数设计和数据增强策略。
















 893
893

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








