目录
LiTS - Liver Tumor Segmentation Challenge数据集概况
LiTS - Liver Tumor Segmentation Challenge数据集概况
参考:
[1] LiTS 数据集介绍 - 知乎 (zhihu.com)
[2]The Liver Tumor Segmentation Benchmark (LiTS)
[3] linhandev/dataset: 医学影像数据集列表 『An Index for Medical Imaging Datasets』 (github.com)
LiTS (The Liver Tumor Segmentation Benchmark) 是专注于肝脏及其肿瘤分割的 CT 数据集。该数据集收集了 7 个不同医学中心的数据,包含 131 例训练集和 70 例测试集,其中测试数据标签不公开。LiTS训练集中包含3DIRCADB中的所有数据,所以不要合并这两个数据集。
基于该数据集,已在 ISBI 2017,MICCAI 2017 和 MICCAI 2018 都成功举办了相关竞赛,并被 MSD (Medical Segmentation Decathlon) 收作 Task03 子任务。

- 数据集基本信息
| 维度 | 模态 | 任务类型 | 解剖结构 | 类别数 | 数据量 | 文件格式 |
| 3D | CT | 分割 | 肝脏/肝脏肿瘤 | 2 | 131训练集(含标注) 70测试集(不含标注) | .nii |
总切片个数: 58638 (基于训练集 131 张统计)
- 图像尺寸统计
| spacing/mm | size | |
| min | (0.5566, 0.5566, 0.7000) | (512, 512, 74) |
| median | (0.7676, 0.7676, 1) | (512, 512, 432) |
| max | (1, 1, 5) | (512, 512, 987) |
- 标注信息统计
| 肝脏 | 肝脏肿瘤 | |
| 出现次数 | 131 | 118 |
| 出现占比 | 100% | 90.08% |
| 最大体积/ | 3194.77 | 987.66 |
| 最小体积/ | 541.79 | 0.04 |
| 中位数体积/ | 1586.49 | 16.11 |
数据集下载
官网:http://www.lits-challenge.com
会跳转到以下界面:

注册账号并登录,然后在Praticipate-Get Data中会提供下载链接(需要科学上网)。
训练集下载(mirror1 & mirror2):https://drive.google.com/drive/folders/0B0vscETPGI1-Q1h1WFdEM2FHSUE?resourcekey=0-XIVV_7YUjB9TPTQ3NfM17A&usp=sharing
https://campowncloud.in.tum.de/public.php?service=files&t=70efd04540107043914f67f7fe1950b0

我是使用Google Drive链接下载的,容易下载到一半失败(尤其是测试集)。而且在Edge中一次性下测试集会被自动分成若干个压缩包。所以还是要盯着点,很容易遗漏。
训练集包含两个.zip文件,Batch1包含28例样本,Batch2包含103例样本,一共131例(均包含标注)(所以官网上写130还错了,233)

测试集包含70例样本,没有标注。(没有提供压缩包,只有散的文件。因此务必往下滑加载全部文件!!不然下完会发现不全)

如何查看数据集?
参考:
怎样打开并查看.nii和DICOM格式的医学图像_nii文件用什么打开-优快云博客
- ITK-SNAP软件
下载地址:ITK-SNAP Version 3.x Downloads (itksnap.org)
我下载的是下面的Windows版本。(被3D Slice坑怕了,再也不下最新版了,谁懂
会让你填写个人信息,这个应该无所谓,正常填写提交即可。
接着跳转到如下页面,提供了citation。直接点击download下的网址,会跳转到下载网址。
下载完成后直接根据指示安装即可,需要改路径就改一下安装路径,其他没什么特别的。安装完成后打开软件,File->Open Main Image,找到你需要浏览的图像。
以打开测试集样本为例。
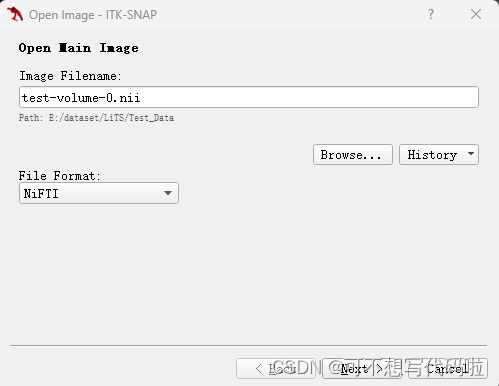
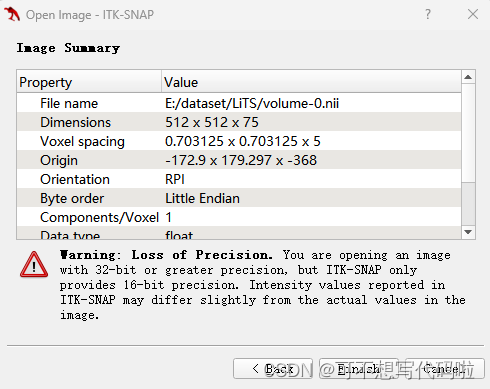
选择文件后,点击Next,就会显示图像的属性和x、y、z轴视图。鼠标点击任一视图的位置并拖动,可以显示相应位置的平面视图。每个视图的右下角都会显示切片位置。
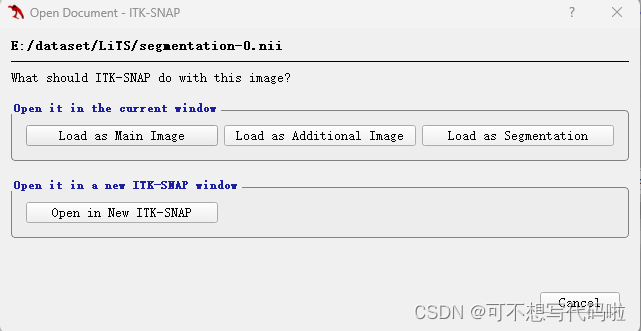
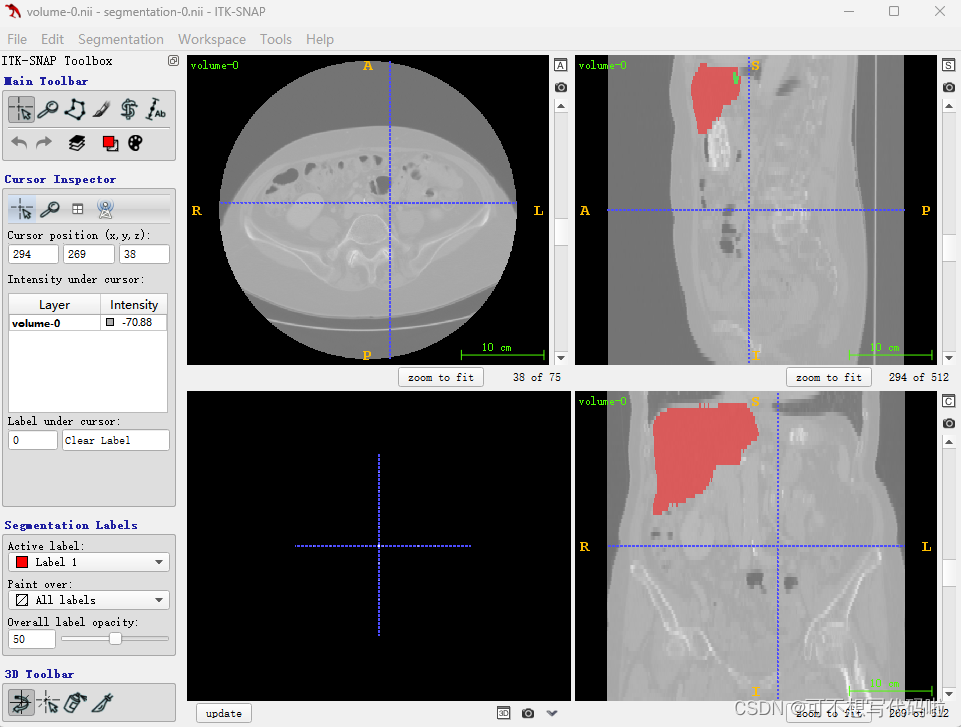
还可以将分割结果直接拖入软件,选择Load as Segmentation,就可以同时显示原始数据和分割结果。(这里用的是训练集,但是报错了,说是图像分辨率问题...咱也不懂)
- Fiji / ImageJ
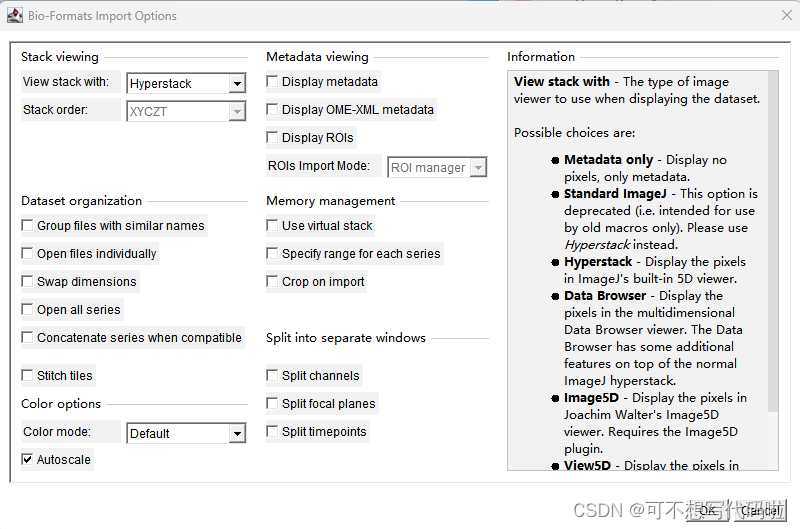
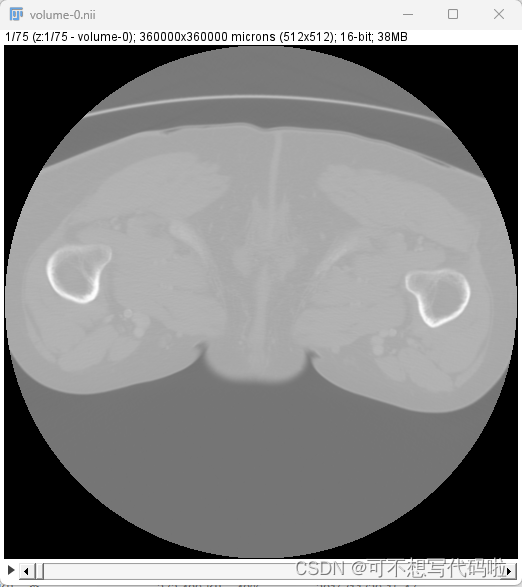
把要查看的图像直接拖进ImageJ也可以查看。不过就只有一个z轴视图可以看,我觉得如果是快速浏览做二维分割也够用了。文件拖进来后有如下页面,不用管直接点OK。
然后就能看到这样的二维视图。拖动最下方的条可以手动调整查看位置,或者使用左下角的播放按钮自动顺序播放。
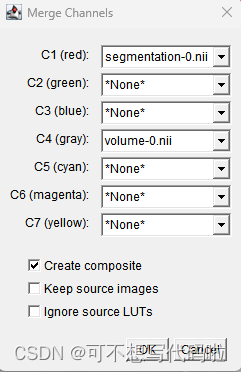
也可以一并显示分割结果。分别打开原数据和分割结果,然后选择Image->Color->Merge Channels,做如下设置,其中分割结果随便选择一个合适的颜色都行,我选的是红色。如果希望还能分别查看原图和分割结果,可以勾选Keep source images。
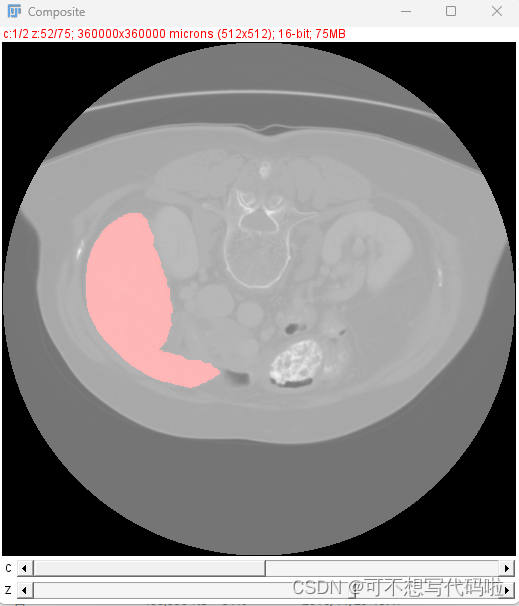
然后就能看到如下结果。
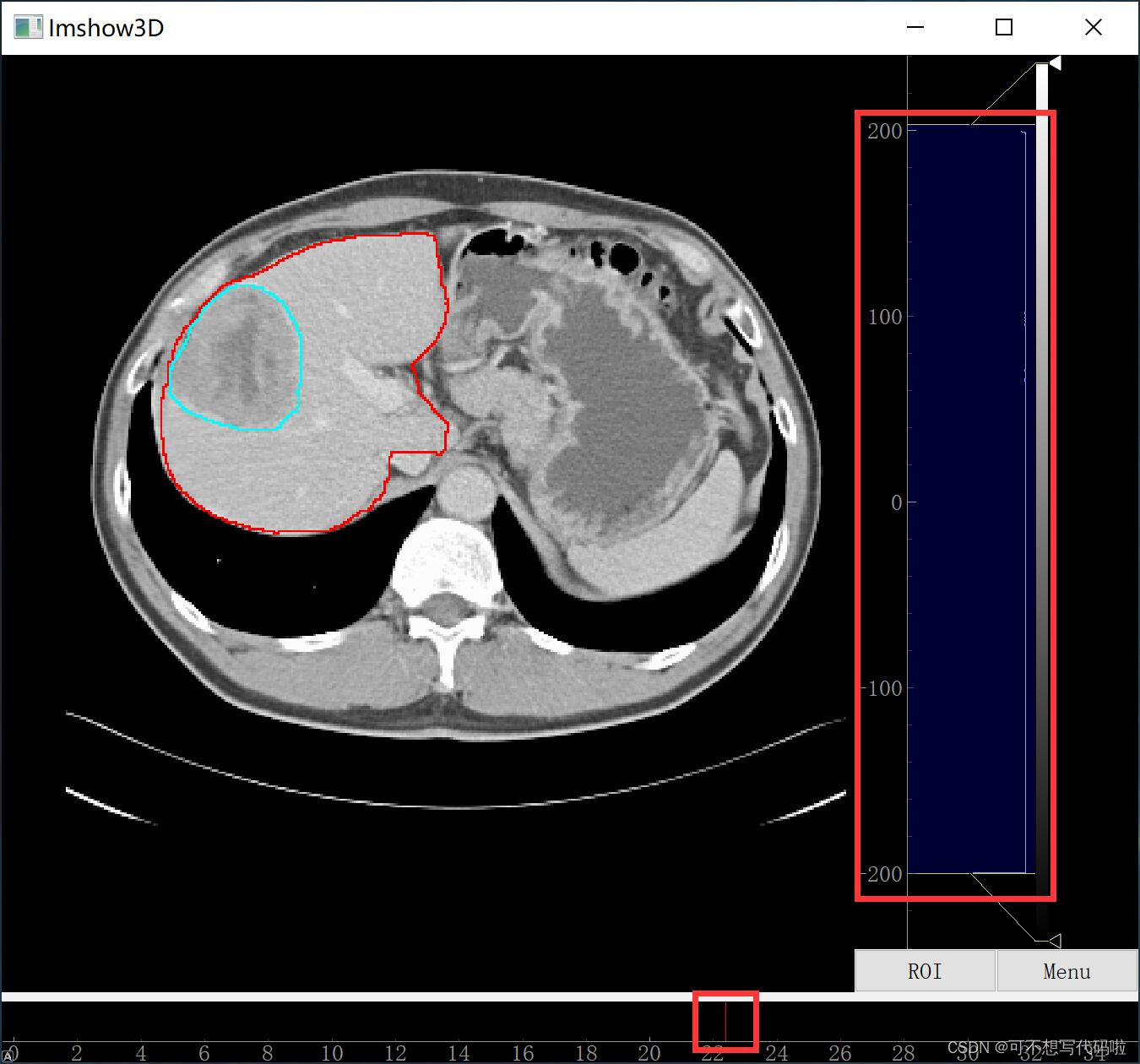
- python代码
我没有深入尝试,感兴趣的可以试试看。详情参考博客中作者给出的代码地址,见:zuzhiang/show-nii (github.com)
原文:我给出了两个代码,一个是可以同时显示器官、器官分割结果、肿瘤分割结果的,另一个是只显示器官的。当然,大家可以在第一个的基础上做改动。与 ITK-SNAP 不同,在显示的时候只显示分割结果的边界,这样更有利于观察分割的是否合理。并且还可以通过调节上图中右边红框中的滑块来调节图像灰度值的范围,通过调节上图中下方红框中滑块来查看不同横截面的图像。
数据集预处理、使用与转格式
因为提供的都是.nii格式的文件,而我自己的数据集一般倾向于.tif .jpg .png格式。所以在使用之前,我想做完预处理,然后转成我偏好的格式。
- 官方的文件路径结构
LiTS
│
├── Traing Batch 1
│ ├── volume-0.nii
│ └── segmentation-0.nii
│ └── ...
│ ├── volume-27.nii
│ └── segmentation-27.nii
│
├── Traing Batch 2
│ ├── volume-28.nii
│ └── segmentation-28.nii
│ └── ...
│ ├── volume-130.nii
│ └── segmentation-130.nii
|—— Test Data
| |—— test-volume-0.nii
| |—— test-volume-1.nii
| |—— ...
| |—— test-volume-69.nii- 直接读取.nii文件(仅转载,未尝试)
参考:医学图像处理(三):nii和dcm格式读取与保存 - 梅雨明夏 - 博客园 (cnblogs.com)
nibabel
读取:
import nibabel as nib
img = nib.load(path)
print(img.shape) # (z,y,x)
img_array = np.asarray(img.get_fdata())
print(img_array.shape) # (x,y,z)保存:
img = nib.Nifti1Image(img_array, np.eye(4))
nib.save(img, os.path.join('output', 'img.nii.gz')关于get_data()和get_fdata()的区别:一个是返回原始数据,一个是返回浮点数
见神经影像学中的python的一些基本函数的用法_get-fdata-优快云博客
SimpleITK
读取:
import SimpleITK as sitk
img = sitk.ReadImage(ct_path, sitk.sitkInt16) # 可自行改变存储类型
print(img.GetSize()) # (x,y,z)
img_array =sitk.GetArrayFromImage(img)
print(img_array.shape) # (z,y,x)保存:
out = sitk.GetImageFromArray(img_array)
sitk.WriteImage(out,'simpleitk_save.nii.gz')将 numpy array 保存为 nii 格式图像之后,有时候会发现使用 itk-snap 却打不开,这是为什么呢?
因为 itk-snap 只接受 int16 类型的数据,所以你需要将 numpy 数组先强制转换成 int16 类型。
array = array.astype(np.int16)
out = sitk.GetImageFromArray(array)
sitk.WriteImage(out,'save.nii.gz')NIfTI格式医学图像不同方向之间旋转 - 知乎 (zhihu.com)
- 批量预处理与转格式
参考:[1] 将lits2017数据集nii格式转换为png格式以及简单的预处理操作(具体操作代码有注释,代码能运行)_lits数据集-优快云博客
[2] pytorch实战-Unet3d(LiTS)_lits数据集在unet上分割-优快云博客
整个数据集实在是太大了,有40多个G。所以打算对原始图像做截取处理以减少数据量。在标注中,肝脏被标记为1,肿瘤标记为2。
涉及的处理内容:
- CT值调窗
- 截取肝脏和肝肿瘤信号所在slice的大致范围
- 调节对比度
- 重新设置标签(如果不做肿瘤分割的话)
- 转为.png .tif .jpg或其他格式
处理代码主体来自[1],有一点小改动。
import os
import nibabel as nib
import numpy as np
import cv2
import re
from tqdm import tqdm
# 原始数据文件夹路径
data_folder = 'E:/dataset/LiTS/Training Batch 1/'
# 转换后的图像文件夹路径和标签文件夹路径
output_image_folder = 'E:/dataset/LiTS/Train/images/'
output_label_folder = 'E:/dataset/LiTS/Train/masks/'
# 创建保存转换后图像和标签的文件夹
os.makedirs(output_image_folder, exist_ok=True)
os.makedirs(output_label_folder, exist_ok=True)
# 排序函数,将文件名按数字排序
def extract_number(filename):
num = re.findall(r'\d+', filename)
return (int(num[0])) if num else None
# 调窗函数,将像素值映射到指定范围内
def windowing(img, window_center, window_width):
img_min = window_center - (window_width / 2.0)
img_max = window_center + (window_width / 2.0)
img = np.clip(img, img_min, img_max)
img = (img - img_min) / (img_max - img_min)
return img
# 直方图均衡化
def histogram_equalization(img):
hist, bins = np.histogram(img.flatten(), 256, [0, 1])
cdf = hist.cumsum()
cdf_normalized = cdf * hist.max() / cdf.max()
img_equalized = np.interp(img.flatten(), bins[:-1], cdf_normalized)
return img_equalized.reshape(img.shape)
# 获取文件夹中所有的nii文件
image_files = [f for f in os.listdir(os.path.join(data_folder)) if f.endswith('.nii') and 'volume' in f]
label_files = [f for f in os.listdir(os.path.join(data_folder)) if f.endswith('.nii') and 'segmentation' in f]
# 确保文件列表是按样本编号排序的
image_files.sort(key=extract_number)
label_files.sort(key=extract_number)
for image_file, label_file in tqdm(zip(image_files, label_files), total=len(image_files), desc='Processing files...'):
# 从文件名中提取编号
image_index = int(re.search(r'volume-(\d+)', image_file).group(1))
label_index = int(re.search(r'segmentation-(\d+)', label_file).group(1))
# 确认编号相同
assert image_index == label_index, "Image and label files do not match in index!"
# 读取原始图像和标签
image_path = os.path.join(data_folder, image_file)
label_path = os.path.join(data_folder, label_file)
image_data = nib.load(image_path).get_fdata()
label_data = nib.load(label_path).get_fdata().astype(int)
# 获取肝脏出现的切片索引
slices_with_liver = np.where(np.sum(label_data, axis=(0, 1)) > 0)[0]
# 将切片索引前后扩张n,并保证范围不小于0,也不大于总切片数量
n = 1
total_slices = label_data.shape[2]
slices_with_liver_expanded = np.unique([i for slice_index in slices_with_liver for i in range(max(0, slice_index - n), min(total_slices, slice_index + n + 1))])
for i, slice_index in enumerate(slices_with_liver_expanded):
# 将图像数据调窗,窗宽为200,窗位为100
windowed_image = windowing(image_data[:, :, slice_index], 100, 200)
# 直方图均衡化
equalized_image = histogram_equalization(windowed_image)
# 归一化
normalized_image = (equalized_image - np.min(equalized_image)) / (
np.max(equalized_image) - np.min(equalized_image))
# 将标签中的肝脏和肿瘤灰度值设为1,背景灰度值设为0
liver_mask = np.where(label_data[:, :, slice_index] > 0, 1, 0)
# 构建新的文件名
new_image_file = f'{image_index + 1:03d}_{i + 1:03d}.png'
new_label_file = f'{label_index + 1:03d}_{i + 1:03d}.png'
# 将图像保存为彩色png格式,肝脏区域使用浅绿色表示
output_image_path = os.path.join(output_image_folder, new_image_file)
output_image = cv2.cvtColor((normalized_image * 255).astype(np.uint8), cv2.COLOR_GRAY2RGB)
# output_image[liver_mask == 1] = [152, 251, 152] # 使用浅绿色表示肝脏区域
cv2.imwrite(output_image_path, output_image)
# 将标签保存为灰度png格式
output_label_path = os.path.join(output_label_folder, new_label_file)
cv2.imwrite(output_label_path, liver_mask * 255)
print("转换完成!")其中在路径字符串部分差点踩雷。参考:python指定路径斜杠与反斜杠的问题_pycharm复制路径斜杠反过来-优快云博客
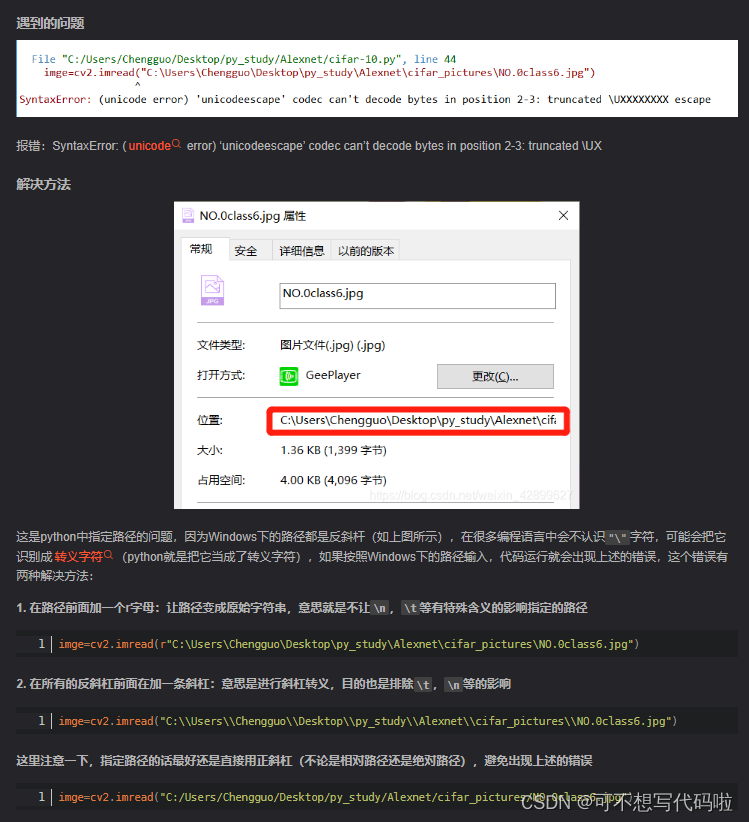
我是在windows跑的代码,配置Pycharm环境可参见:Windows配置Anaconda+Pycharm-优快云博客
我直接在Pycharm中安装nibabel和cv2会报错,显示无法安装。这时在终端的conda环境中下载即可。
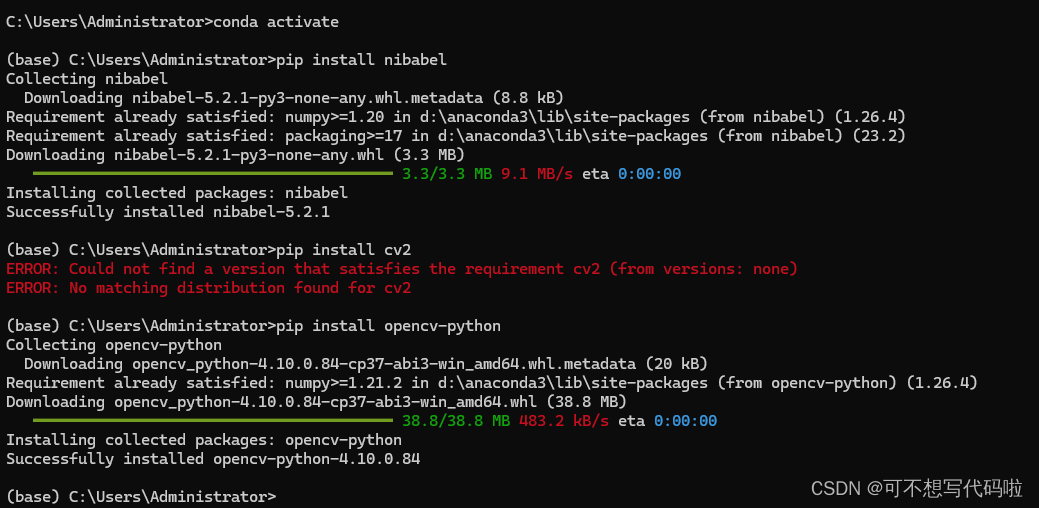
直接跑代码,跑完部分结果如下:
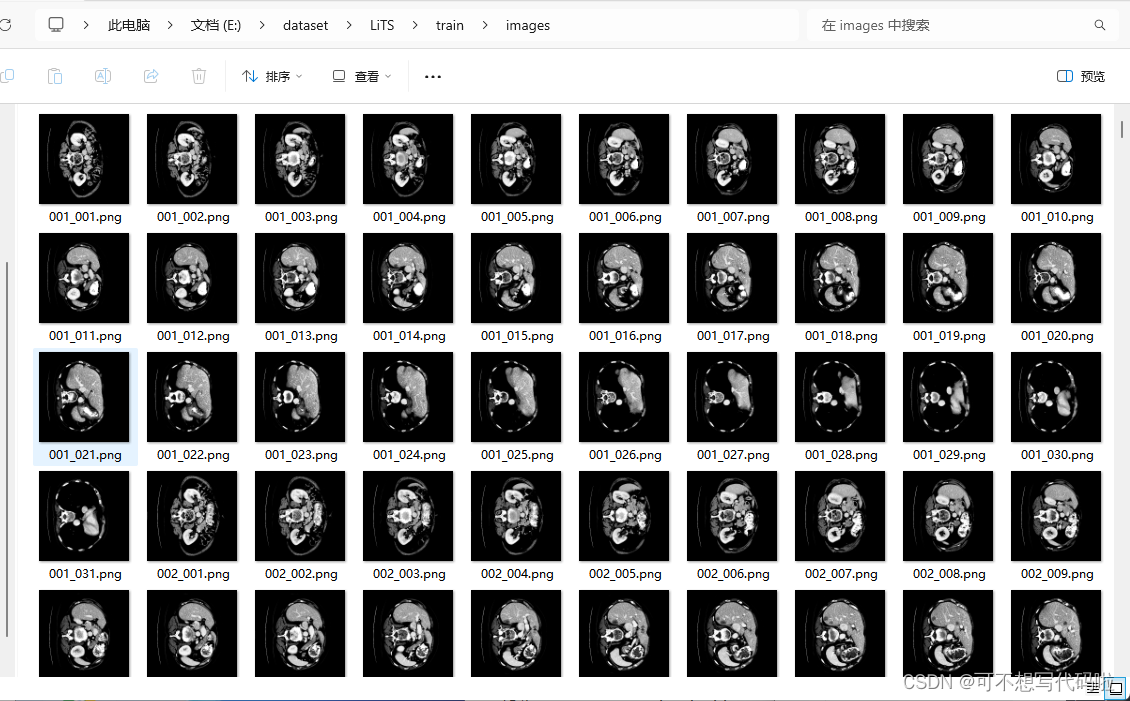
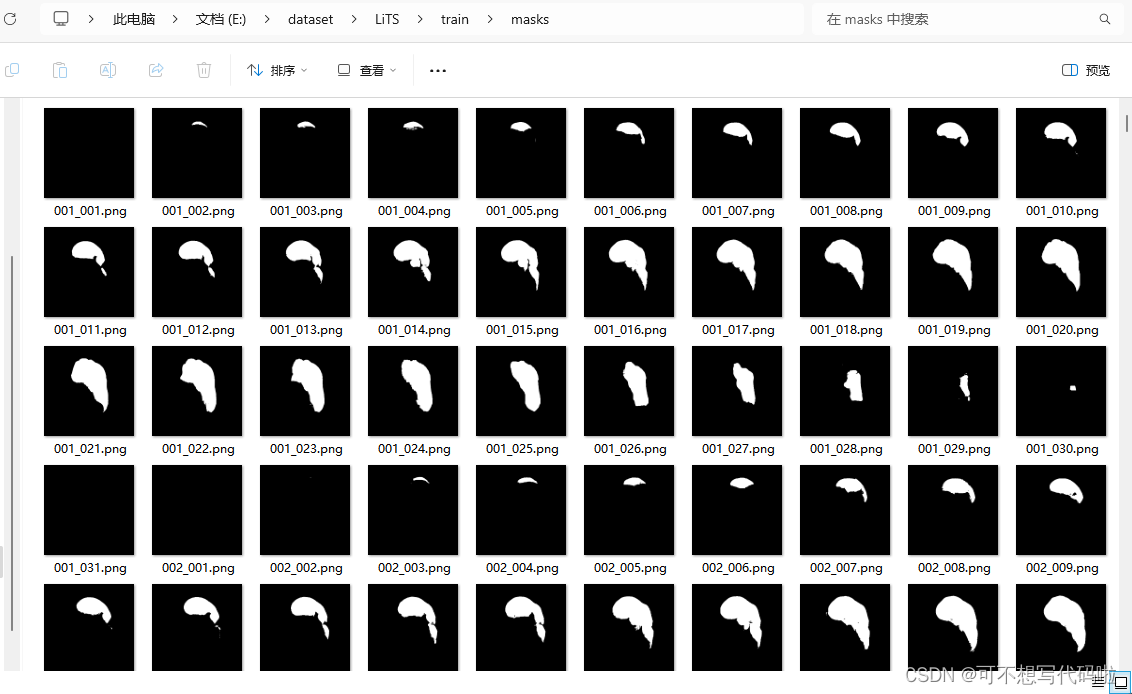
新的训练集文件格式(*中的文字代指该部分的实际含义):
*LiTS Trainset folder name*
│
├── *your images folder name*
│ ├── 001_*start slice number*.png
│ |── 001_*start slice number + 1*.png
│ |── ...
│ ├── 001_*end slice number*.png
│ |── ...
| |—— 002_*start slice number*.png
| |—— ...
| |—— 131_*end slice number*.png
├── *your masks folder name*
│ ├── 001_*start slice number*.png
│ |── 001_*start slice number + 1*.png
│ |── ...
│ ├── 001_*end slice number*.png
│ |── ...
| |—— 002_*start slice number*.png
| |—— ...
| |—— 131_*end slice number*.png测试集也是同理,修改一下上面的代码即可。
import os
import nibabel as nib
import numpy as np
import cv2
import re
from tqdm import tqdm
# 原始数据文件夹路径
data_folder = 'E:/dataset/LiTS/Test_Data/'
# 转换后的图像文件夹路径
output_image_folder = 'E:/dataset/LiTS/test/images/'
# 创建保存转换后图像的文件夹
os.makedirs(output_image_folder, exist_ok=True)
# 排序函数,将文件名按数字排序
def extract_number(filename):
num = re.findall(r'\d+', filename)
return (int(num[0])) if num else None
# 调窗函数,将像素值映射到指定范围内
def windowing(img, window_center, window_width):
img_min = window_center - (window_width / 2.0)
img_max = window_center + (window_width / 2.0)
img = np.clip(img, img_min, img_max)
img = (img - img_min) / (img_max - img_min)
return img
# 直方图均衡化
def histogram_equalization(img):
hist, bins = np.histogram(img.flatten(), 256, [0, 1])
cdf = hist.cumsum()
cdf_normalized = cdf * hist.max() / cdf.max()
img_equalized = np.interp(img.flatten(), bins[:-1], cdf_normalized)
return img_equalized.reshape(img.shape)
# 获取文件夹中所有的nii文件
image_files = [f for f in os.listdir(os.path.join(data_folder)) if f.endswith('.nii') and 'volume' in f]
# 确保文件列表是按样本编号排序的
image_files.sort(key=extract_number)
for image_file in tqdm(image_files, total=len(image_files), desc='Processing files...'):
# 从文件名中提取编号
image_index = int(re.search(r'test-volume-(\d+)', image_file).group(1))
# 读取原始图像和标签
image_path = os.path.join(data_folder, image_file)
image_data = nib.load(image_path).get_fdata()
# 获取切片总数索引
total_slices = image_data.shape[2]
all_slices_indices = np.arange(total_slices)
for i, slice_index in enumerate(all_slices_indices):
# 将图像数据调窗,窗宽为200,窗位为100
windowed_image = windowing(image_data[:, :, slice_index], 100, 200)
# 直方图均衡化
equalized_image = histogram_equalization(windowed_image)
# 归一化
normalized_image = (equalized_image - np.min(equalized_image)) / (
np.max(equalized_image) - np.min(equalized_image))
# 构建新的文件名
new_image_file = f'{image_index + 1:03d}_{i + 1:03d}.png'
# 将图像保存为彩色png格式,肝脏区域使用浅绿色表示
output_image_path = os.path.join(output_image_folder, new_image_file)
output_image = cv2.cvtColor((normalized_image * 255).astype(np.uint8), cv2.COLOR_GRAY2RGB)
cv2.imwrite(output_image_path, output_image)
print("转换完成!")主要是要记得去掉zip()函数,因为这里不需要并行迭代;以及修改一下search函数中的文件名。
参考:Python zip()用法,看这一篇就够了-优快云博客





















 2万+
2万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








