参考:
https://zhuanlan.zhihu.com/p/72490817?from_voters_page=true
一、准备数据集
wget http://zzz.bwh.harvard.edu/plink/dist/example.zip
unzip example.zip
cd example/检查ped文件和map文件的存在
plink --file wgas1生成二进制的ped文件(fam bed bai)
plink --file wgas1 --make-bed --out wgas1
二、逻辑回归
plink --bfile wgas1 --logistic
head plink.assoc.logistic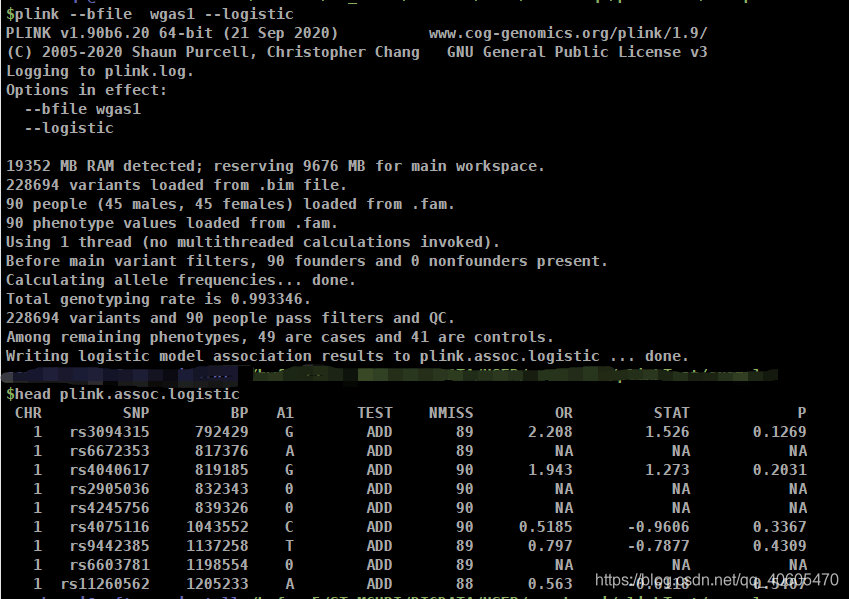




 这篇博客介绍了如何利用PLINK工具进行基因关联研究。首先,通过wget下载并解压数据集,然后使用plink检查ped和map文件,接着将文本格式的ped文件转换为二进制bed文件。最后,运用plink的logistic选项执行逻辑回归分析,并查看关联结果。这是一个关于生物信息学和基因组学研究的基础教程。
这篇博客介绍了如何利用PLINK工具进行基因关联研究。首先,通过wget下载并解压数据集,然后使用plink检查ped和map文件,接着将文本格式的ped文件转换为二进制bed文件。最后,运用plink的logistic选项执行逻辑回归分析,并查看关联结果。这是一个关于生物信息学和基因组学研究的基础教程。
















 891
891

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








