1、python astype(‘category’), 编码和标签对应,categories 和 code 映射为字典
import pandas as pd
# 创建数据集
df = pd.DataFrame.from_dict(
{
'col1': ['A', 'B', 'C', 'B', 'A', 'D'],
'col2': ['aa', 'bb', 'bb', 'aa', 'dd', 'dd'],
'col3': ['yes', 'yes','no','no','yes','yes']
}
,orient='columns')
# df
# col1 col2 col3
# 0 A aa yes
# 1 B bb yes
# 2 C bb no
# 3 B aa no
# 4 A dd yes
# 5 D dd yes
# 转换为分类数据
df = df.astype('category') # 可以指定特定的列转为分类数据 df['col1'] = df['col1'].astype('category')
# 将标签数据转换为编码
df_code = pd.DataFrame({col: df[col].cat.codes for col in df}, index=df.index)
# df_code
# col1 col2 col3
# 0 0 0 1
# 1 1 1 1
# 2 2 1 0
# 3 1 0 0
# 4 0 2 1
# 5 3 2 1
# 将编码和标签一一对应,转为字典,方便查询
df_code_dict = {col: {code: category for code, category in enumerate(df[col].cat.categories)} for col in df}
# df_code_dict
# {'col1': {0: 'A', 1: 'B', 2: 'C', 3: 'D'},
# 'col2': {0: 'aa', 1: 'bb', 2: 'dd'},
# 'col3': {0: 'no', 1: 'yes'}}
2.1、csr_matrix索引方式1
这种方式百度介绍的较多
indptr = np.array([0, 2, 3, 6])
indices = np.array([0, 2, 2, 0, 1, 2])
data = np.array([1, 2, 3, 4, 5, 6])
csr_matrix((data, indices, indptr), shape=(3, 3)).toarray()
输出:
array([[1, 0, 2],
[0, 0, 3],
[4, 5, 6]])
csr中的r表示row,即为行优先。
indptr中的元素两个两个的看,前两个为0和2,表示第一行有2-0=2个元素,再在indices中选前两个元素,得到在列上的位置为0和2,填充data的元素为1和2。接在是indptr中的2和3,所以第二行有3-2=1个元素,要填充的列位置是indices 中的2,元素从data中找为3. 在接下来是indptr中的3和6,表示第三行有6-3=3个元素,要填充的位置是indices 中的0、1、2,元素从data中找为4,5,6.
2.1、csr_matrix索引方式2
根据矩阵值进行索引
df = pd.DataFrame.from_dict(
{
'col1': [1, 0, 2, 3, 2, 1],
'col2': [1, 0, 1, 2, 3, 4],
'col3': [14, 15, 10, 20, 30, 40]
}
,orient='columns')
df
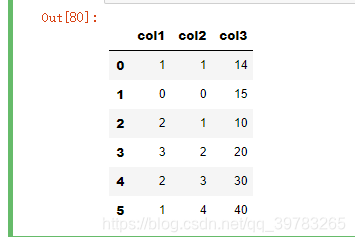
sparse_item_user = sparse.csr_matrix((df['col3'], (df['col1'], df['col2'])))
sparse_item_user.toarray()





 本文详细介绍使用Python的Pandas库进行数据编码和标签映射的方法,以及如何利用Scipy的CSR矩阵进行高效的数据存储与索引。通过具体实例展示了Pandas在数据处理中的灵活性,及CSR矩阵在稀疏数据存储方面的优势。
本文详细介绍使用Python的Pandas库进行数据编码和标签映射的方法,以及如何利用Scipy的CSR矩阵进行高效的数据存储与索引。通过具体实例展示了Pandas在数据处理中的灵活性,及CSR矩阵在稀疏数据存储方面的优势。
















 611
611

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








