这是ECCV2018的一篇关于脑部肿瘤分割的论文
文中提出目前脑部肿瘤分割的两个主要困难
1、class imbalance
主要表现在两个方面,第一是不是肿瘤的区域可能是肿瘤区域大小的上千倍,第二是一些肿瘤组织相对于其他的要大
2、inter-class interference
主要来源于不同肿瘤组织之间的相似特征,导致对于每一类的辨别造成困难
这篇文章提出了FSENet处理这些问题
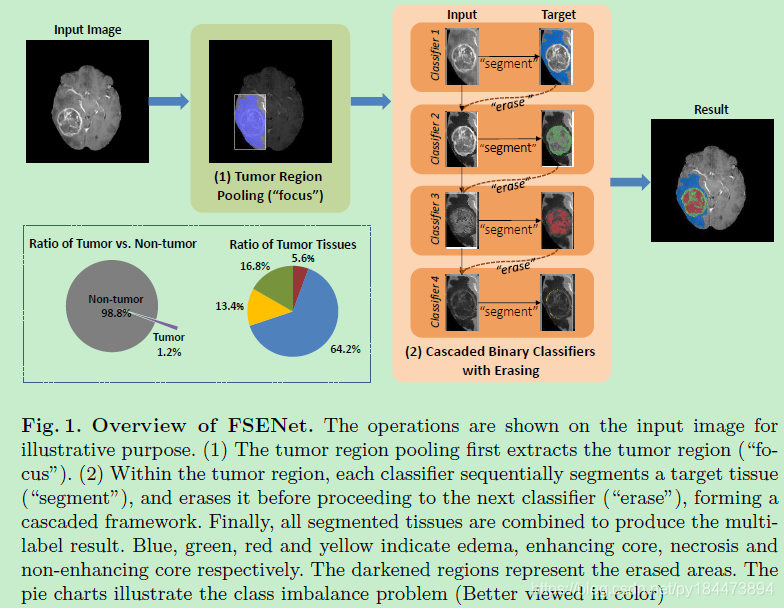
分为三步focus、segment、erase
对于整个大脑来说,检测出含有肿瘤的区域较为简单,因此先进行focus得到含有肿瘤的区域
接着对于含有肿瘤区域的肿瘤多组织分割分为两步,“outer-to-inner”,第一步是segment,也就是先分割出一个肿瘤组织,然后再进行erase,也就是把上一步分割出来的组织进行擦除,之后再进行下一个肿瘤组织的分割,依次反复,最终得到各个肿瘤组织的单独分割结果,再融合到一起成为最终分割图
FSENet
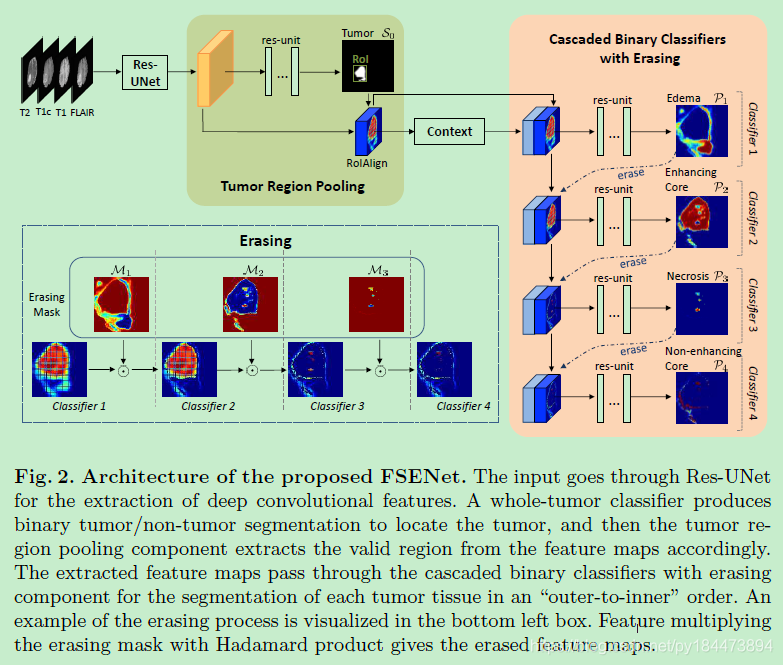
FSENet分为两个部分tumor region pooling and cascaded binary classifiers with erasing
网络的输入使用了四种模态(contrast-enhanced T1-weighted (T1c) image,T1-weighted (T1) image, T2-weighted (T2) image and FLuid-Attenuated Inversion Recovery (FLAIR) image)的MRI进行concate
Res-UNet
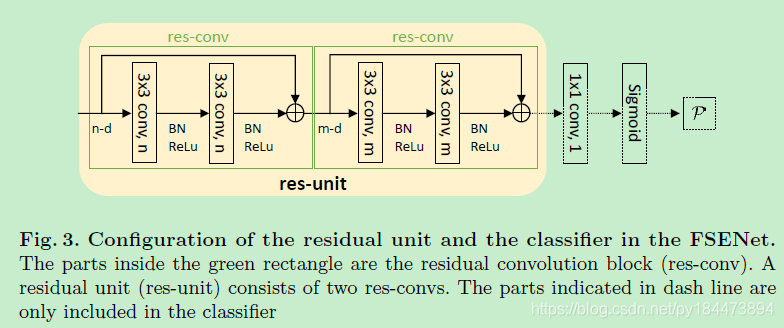
使用res-conv模块替代U-net中的conv模块
Res-UNet作为backbone进行深层卷积特征的提取
首先使用网络进行肿瘤区域的分割,然后使用RoIAlign从原本特征图上提取到肿瘤区域的特征,接着把这些特征用于肿瘤组织的分割
这么做的好处有两点:1、非肿瘤区域含有一些no-measurement area(black region),能够节省计算资源
2、非肿瘤区域不会阻碍接下来的多label分割
Cascaded Binary Classifiers with Erasing
inter-class future similarity和class imbalance在不同肿瘤组织中很常见,因此很难使用one-stage方法进行同时分割,因此本文提出了将multi-label问题转化为多个binary问题,one-stage到multi-stage任务。
简单地转为binary问题并不会带来很大的提升,这是因为对于相似或者微小组织的分割,依旧收到其他组织的影响,为了解决这个问题,先使用outer-to-inner从外到内进行分割,然后使用erase操作将已经分割后的组织进行消除
erase操作是使用element-wise对RoI feature map进行mask的擦除:


erase操作并不是简单的将predict map进行一个thresholding
Loss and Final Result
对于每个肿瘤组织的分割结果,使用dice loss
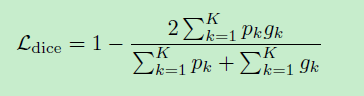










 FSENet是一种针对脑部肿瘤分割难题的深度学习解决方案,通过Focus、Segment和Erase三步骤,有效应对类不平衡和类别间干扰问题。采用Res-UNet作为骨干网络,结合RoIAlign和级联二元分类器,实现多组织肿瘤的精准分割。
FSENet是一种针对脑部肿瘤分割难题的深度学习解决方案,通过Focus、Segment和Erase三步骤,有效应对类不平衡和类别间干扰问题。采用Res-UNet作为骨干网络,结合RoIAlign和级联二元分类器,实现多组织肿瘤的精准分割。

















 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








