1、github下载安装包
OpenNE有两个版本,pytorch版本安装一直报错,放弃;
然后安装了tensorflow版本,成功。
2、本地安装
2、1 先创建虚拟环境:conda create --name openneenv python=3.6
2、2 进入虚拟环境,安装相关的依赖包(注意指定版本):
numpy == 1.16.4
networkx == 2.0
scipy == 1.2.1
tensorflow == 1.12.2
gensim == 3.0.1
scikit-learn == 0.19.0
2、3 在虚拟环境中执行以下两步(即安装openne库)
cd src
python setup.py install
执行之后 pip list可以看见openne库
2、4 安装好之后,就可以在终端运行了
注意一定要在主目录OpenNE-master下运行
尝试运行一个较小的数据集:
python -m openne --method node2vec --label-file data/cora/cora_labels.txt --input data/cora/cora_edgelist.txt --graph-format edgelist --feature data/cora/cora.features --epochs 200 --output vec_all.txt --clf-ratio 0.1
终端运行截图如下:
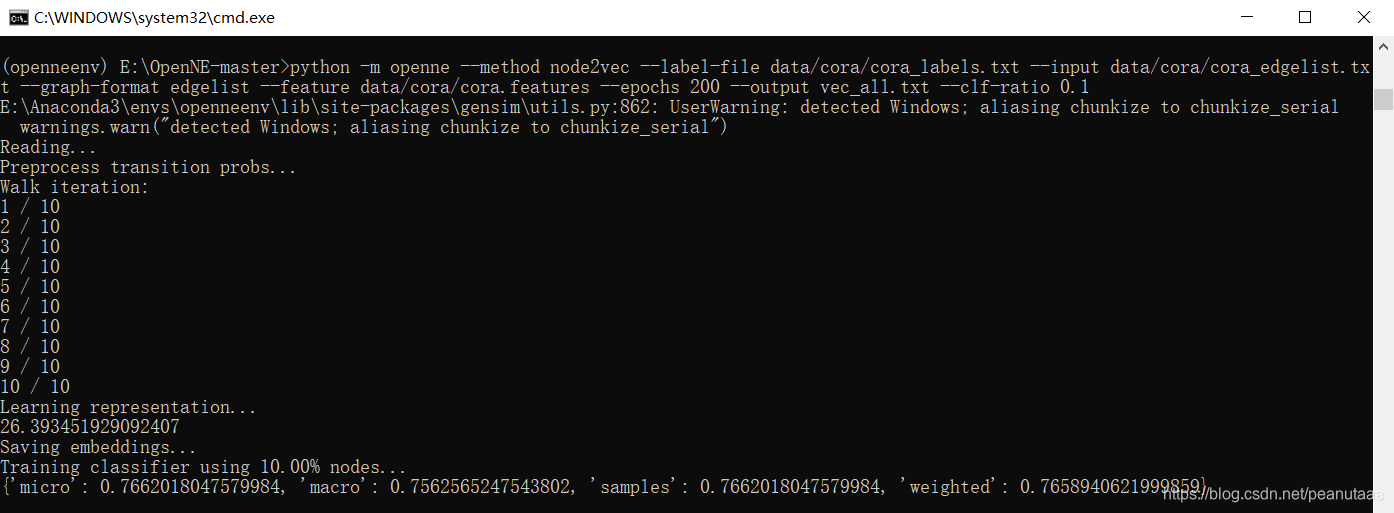
2、5
进行到这一步,代码终于跑通了!但只是代码跑通了一个而已,具体怎么实现还需要仔细的调试运行。
然后就是根据自己的理解、自己的算法让代码按照自己的想法进行,能够跑自己的数据集。
再者,目前只能在终端通过命令跑,如何调试使其能够在pycharm上跑。。。待我前去研究0-0
注
关于deepwalk在终端的执行,类似操作。下载链接
终端执行deepwalk
(condavenv1) E:\deepwalk\deepwalk>python main.py --input E:\deepwalk\example_graphs\karate.adjlist --output E:\karate.embeddings
输出:
Number of nodes: 34
Number of walks: 340
Data size (walks*length):13600
Walking…
Training…
openne在pycharm中运行
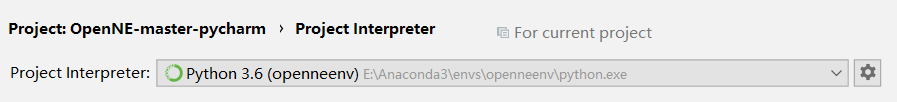
1、设置pycharm中的解释器为创建的虚拟环境的解释器(这样就可以使用虚拟环境中安装的依赖包)
2、修改原文件中的导包方式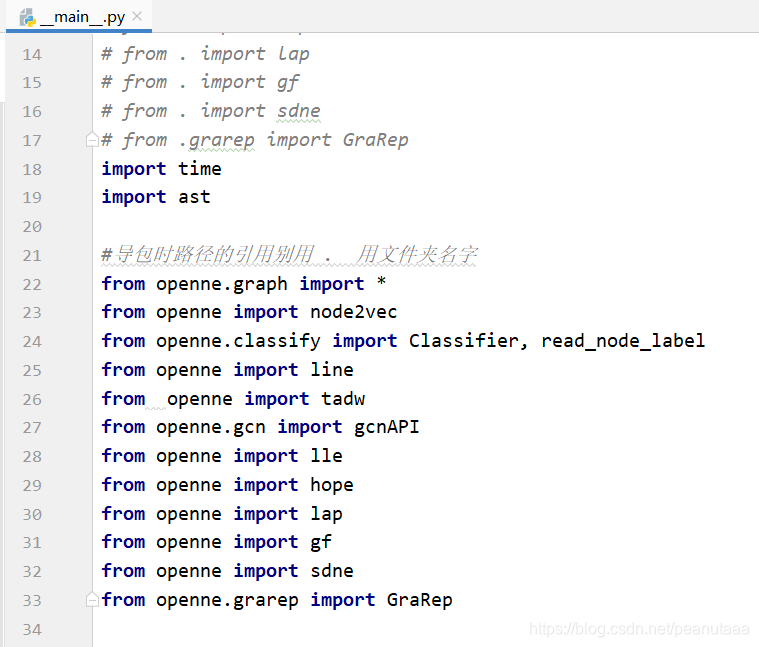
3、由于从github中下载的代码是作为包直接在cmd运行的,所以想要在pycharm中运行还需要修改以下内容
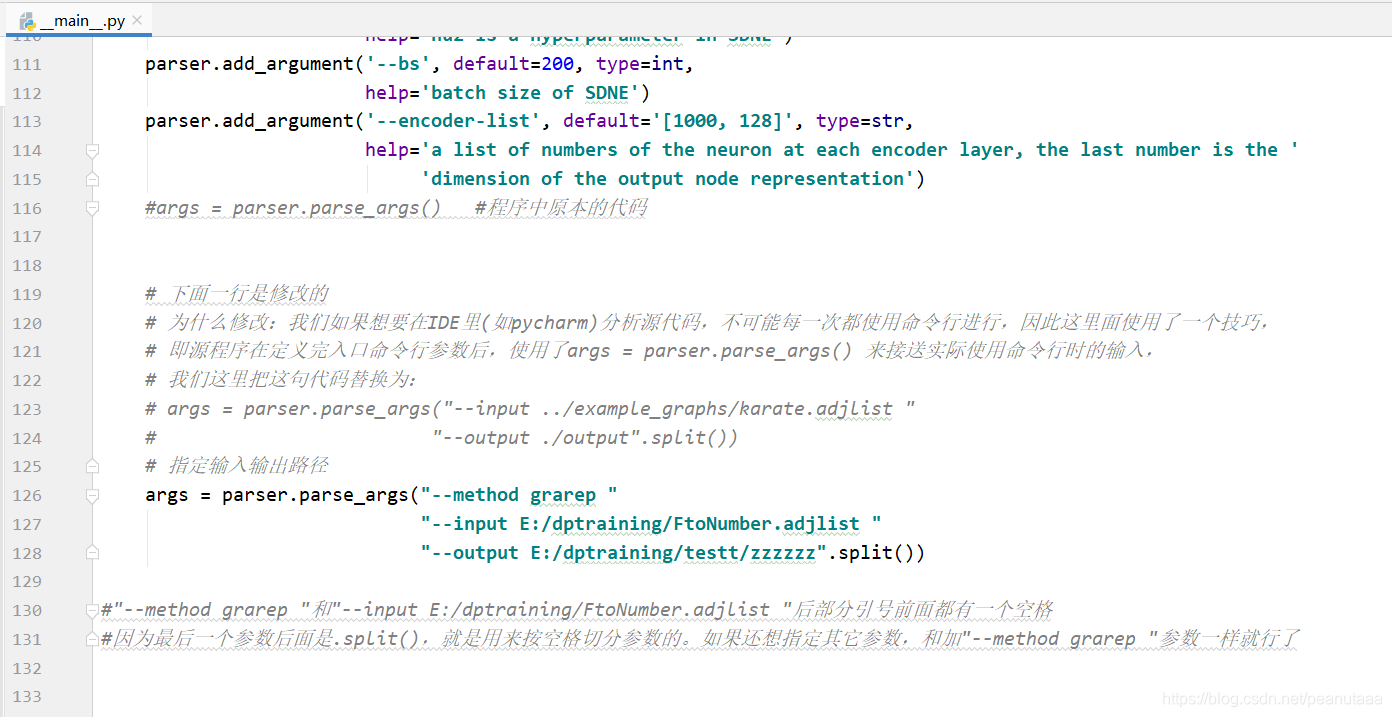
这样就可以在pycharm中运行了







 本文介绍了如何在Python中通过conda环境安装OpenNE的tensorflow版本,包括创建虚拟环境、配置依赖包、源码安装并成功运行node2vec示例。后续探讨了如何在PyCharm中调试和应用到实际项目中,以及对DeepWalk的执行过程。
本文介绍了如何在Python中通过conda环境安装OpenNE的tensorflow版本,包括创建虚拟环境、配置依赖包、源码安装并成功运行node2vec示例。后续探讨了如何在PyCharm中调试和应用到实际项目中,以及对DeepWalk的执行过程。
















 3034
3034

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








