细胞通讯分析是一种研究不同细胞类型之间如何通过信号分子(如配体和受体)进行相互交流和调控的分析方法。它在揭示细胞间相互作用的机制,理解组织和器官如何协调运作方面具有重要意义。
细胞通讯分析的主要内容如下:
-
配体-受体相互作用: 每个细胞类型都会表达特定的配体和受体。细胞通讯分析通过研究这些配体和受体之间的相互作用,来了解细胞是如何“交流”的。例如,一个细胞释放的配体可以与另一种细胞的受体结合,从而触发一系列细胞内的信号传导事件。
-
信号通路活性: 通过分析哪些信号通路在不同细胞类型之间被激活,研究者可以了解哪些信号通路在特定生物学过程中起作用,如细胞分化、免疫反应或疾病状态等。
-
细胞间网络构建: 细胞通讯分析通常会构建一个细胞间的网络图,显示不同细胞类型之间的相互作用方式。这个网络可以帮助识别主要的信号发送者和接收者,以及哪些细胞在组织中起主导作用。
-
定量分析: 通过计算细胞通讯的强度(如配体-受体结合的概率),可以定量分析不同细胞类型之间的通讯强度。这可以揭示在特定条件下,哪种细胞类型在通讯中占主导地位。
-
功能富集分析: 细胞通讯分析还可以与功能富集分析结合使用,探讨哪些生物学功能或通路受到特定细胞通讯的调控。
常用工具:
1、CellChat:这是一个常用的 R 包,可以识别和量化不同细胞类型之间的通讯,并提供丰富的可视化工具。
2、CellPhoneDB:另一个广泛使用的工具,专注于基于配体-受体相互作用的细胞通讯分析。
本次主要来学习一下cellchat这个工具。
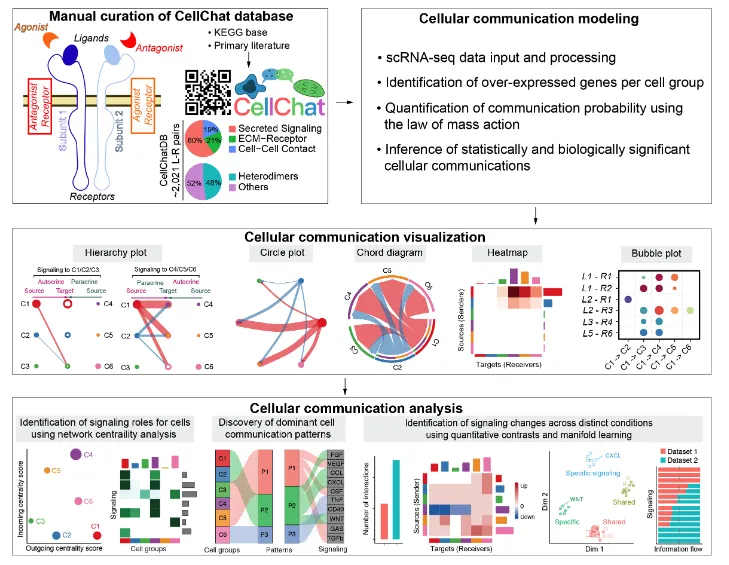

CellChatDB v2数据库中包含了丰富的先验信息:
1、数据库内容来源于已知文献的人和小鼠的配-受体相互作用信息。
2、这些信息包含了大约3300个经过验证的分子互作信息(40%是自分泌/旁分泌,17%是细胞外基质-受体,13%是细胞-细胞间接触,30%是非蛋白质信号)
3、相比于v1版本, CellChatDB v2增加了1000多种的蛋白质/非蛋白质的互作信息,比如代谢和突触信号传导。
4、对于数据没有直接作用的关键分子,该工具还能通过分析这些分子的关键介质/酶的情况来预测配-受体表达情况进而预测潜在通讯(这个是对~30%的非蛋白信号互作信息来源的解释)。
5、这个版本还增加了更多的附加注释信息:比如UniProtKB关键字(包括生物过程、分子功能、功能类别、疾病等)、亚细胞位置以及与神经递质的相关性。
6、人和小鼠的配受体库是不一样的,需要自行设定:CellChatDB.human/CellChatDB.mouse。

关于数据输入和准备的流程里开发者提供了不同数据的处理方式,包括conut matrix,seruat对象,SingleCellExperiment对象,Anndata对象等。
详细准备流程可以查看github官网,链接附在参考资料中,笔者这里使用的是Seurat对象。
步骤流程
1.导入
rm(list?=?ls())
library(CellChat)
library(Seurat)
library(qs)
library(patchwork)
library(BiocParallel)
register(MulticoreParam(workers?=?4,?progressbar?=?TRUE))
load('scRNA.Rdata')?
#?check一下
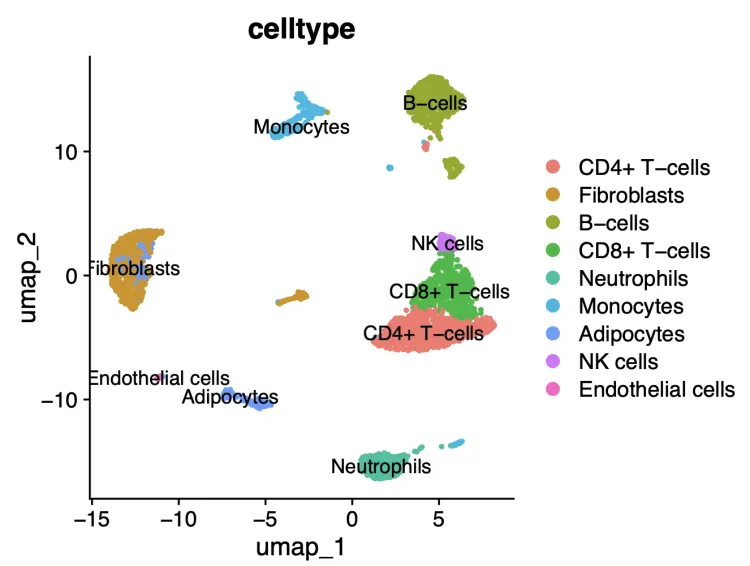
DimPlot(scRNA,pt.size?=?0.8,group.by?=?"celltype",label?=?T)
2.数据预处理
CellChat需要两个用户输入:一个是细胞的基因表达数据,另一个是用户分配的细胞标签。
对于基因表达数据矩阵,行为基因,列为细胞。
CellChat需要数据归一化数据。如果是count数据,里面内置了功能可以进行处理转换(但一般不用count,使用归一化数据)。
建议使用前先捋顺自己的数据。
data.input?<-?GetAssayData(scRNA,?layer?=?'data')
meta?<-?scRNA@meta.data[,c("orig.ident","celltype")]
colnames(meta)?<-??c("group","celltypes")
table(meta$celltypes)
meta$celltypes?<-?gsub("?cells|-cells",?"",?meta$celltypes)
table(meta$celltypes)
?#?Adipocytes???????????B??????CD4+?T??????CD8+?T?Endothelial?Fibroblasts?
?#????????244?????????667????????1007?????????547??????????28?????????762?
?#??Monocytes?Neutrophils??????????NK?
?#????????311?????????378??????????87?
#?建议提前对celltype进行排序~?
identical(rownames(meta),colnames(data.input))
celltype_order?<-?c("CD4+?T","CD8+?T","B","Adipocytes","Endothelial",
????????????????????"Fibroblasts","Monocytes","Neutrophils","NK")
meta$celltypes?<-?factor(meta$celltypes?,levels?=?celltype_order)
#?根据?meta$celltypes?的顺序进行排序
ordered_indices?<-?order(meta$celltypes)
#?对?meta?和?data.input?进行排序
meta?<-?meta[ordered_indices,?]
data.input?<-?data.input[,?ordered_indices]
identical(rownames(meta),colnames(data.input))
3.创建Cellchat对象
#?构建cellchat
cellchat?<-?createCellChat(object?=?data.input,?
???????????????????????????meta?=?meta,?
???????????????????????????group.by?=?"celltypes")
levels(cellchat@idents)
#?[1]?"CD4+?T"??????"CD8+?T"??????"B"???????????"Adipocytes"??"Endothelial"?"Fibroblasts"
#?[7]?"Monocytes"???"Neutrophils"?"NK"?
4.设置配体-受体相互作用数据库
CellChatDB?<-?CellChatDB.human?#?use?CellChatDB.mouse?if?running?on?mouse?data
showDatabaseCategory(CellChatDB)
dplyr::glimpse(CellChatDB$interaction)
#?使用CellChatDB的中特定的数据库进行细胞-细胞通信分析
#?示例中使用了Secreted?Signaling
#?CellChatDB.use?<-?subsetDB(CellChatDB,?search?=?"Secreted?Signaling",?key?=?"annotation")?
#?Only?uses?the?Secreted?Signaling?from?CellChatDB?v1
#??CellChatDB.use?<-?subsetDB(CellChatDB,?search?=?list(c("Secreted?Signaling"),?c("CellChatDB?v1")),?key?=?c("annotation",?"version"))
#?使用所有CellChatDB数据进行细胞-细胞通信分析。
CellChatDB.use?<-?CellChatDB?
#?在构建的cellchat中设定需要使用的数据库
cellchat@DB?<-?CellChatDB.use
5.预处理细胞-细胞通讯分析的表达数据
cellchat?<-?subsetData(cellchat)?
#?future::plan("multisession",?workers?=?1)?#?do?parallel
cellchat?<-?identifyOverExpressedGenes(cellchat)
cellchat?<-?identifyOverExpressedInteractions(cellchat)
#?默认情况下,cellchat使用object@data.signaling进行网络推断
#?此外提供了projectData函数,将基因投射到PPI,开发者说PPI并不会或导致很少的伪通讯
cellchat?<-?projectData(cellchat,?PPI.human)

如何推断细胞间的通讯信息:
该工具可识别纳入分析数据的细胞群中存在








 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章

















 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








