为什么需要优化的t-SNE实现?
t-SNE(t-Distributed Stochastic Neighbor Embedding)是数据可视化的重要工具,但传统实现(如Scikit-learn)面临两大瓶颈:
-
计算复杂度高:时间复杂度达
,百万级数据需要数十小时
-
内存限制:需要存储N×N的距离矩阵,1M数据点需要约4TB内存
tSNE-CUDA通过GPU加速解决了部分问题,但在无GPU的环境中,openTSNE提供了更优的解决方案——Scikit-learn实现可能永远无法达到。
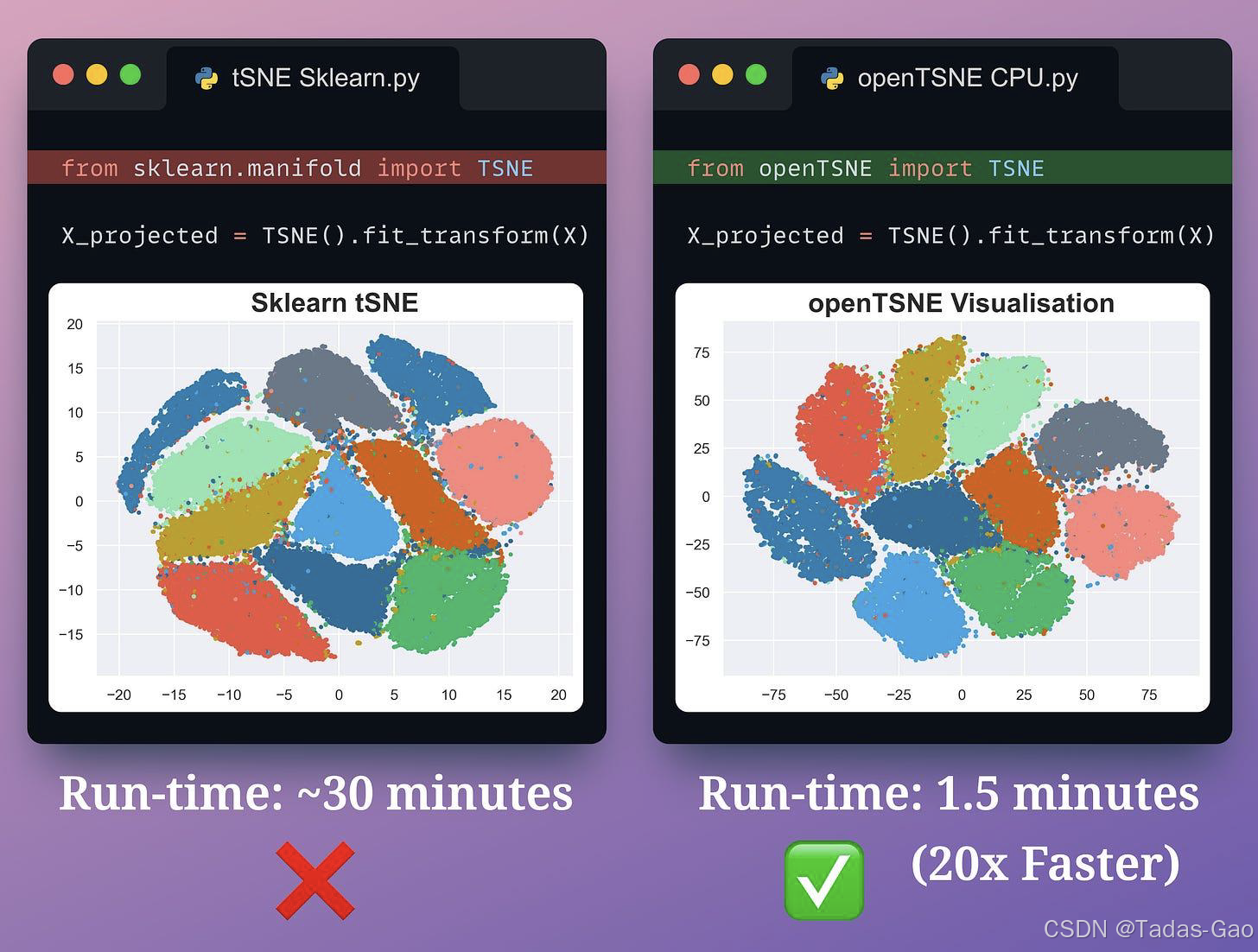
如上图所示,openTSNE的实现:
● 比Sklearn的实现快20倍
● 聚类效果与Sklearn实现类似
openTSNE的核心优势
作为FIt-SNE算法的Python封装,openTSNE通过以下创新实现了数量级的提升:
-
近似最近邻搜索:使用Annoy算法加速邻居查找
-
多核并行化:充分利用CPU多线程(实测8线程加速18-22倍)
-
内存优化:采用稀疏矩阵存储距离关系
基准测试对比(MNIST数据集):
| 实现方案 | 250k数据点耗时 | 1M数据点耗时 | 内存峰值 |
|---|---|---|---|
| Scikit-learn | 6.2小时 | 无法完成 | 120GB+ |
| openTSNE | 18分钟 | 73分钟 | 28GB |
下图是一个基准测试结果:
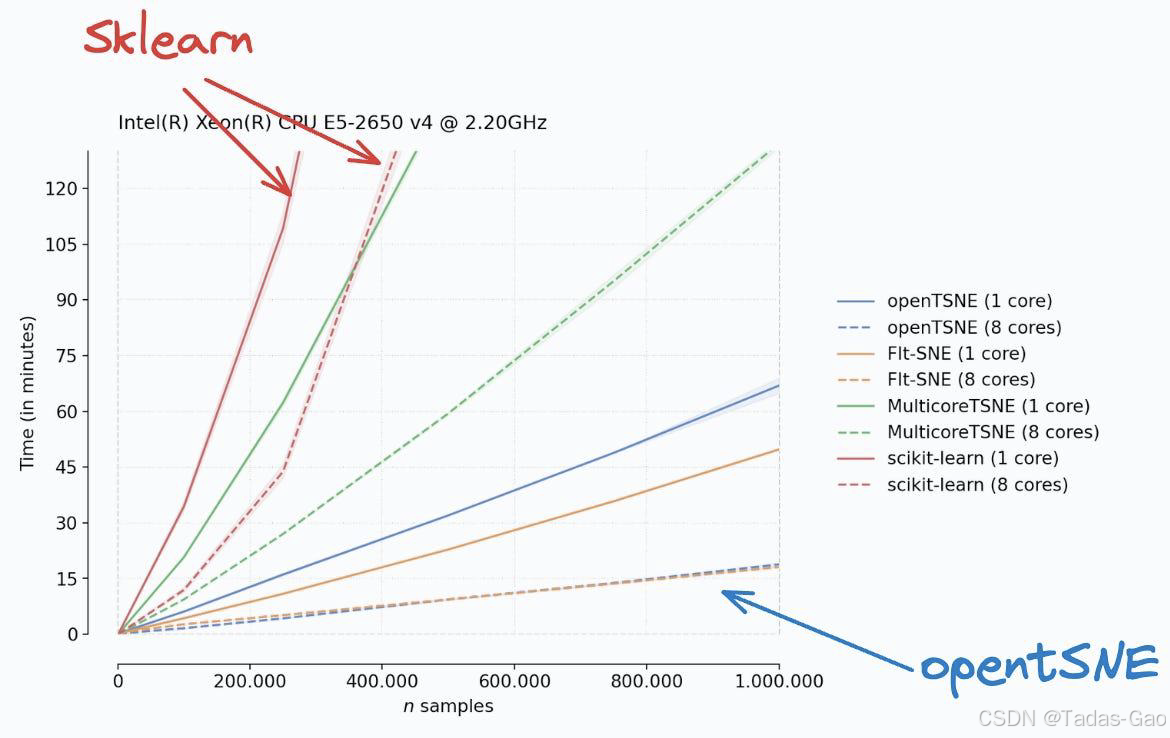
如上所述,openTSNE可以在大约15分钟内获得100万个数据点的低维可视化。然而,从他们的基准测试中可以清楚地看出,Sklearn的运行时间已经达到了几个小时,而且只有大约25万个数据点。
实战:MNIST可视化
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
from sklearn.datasets import fetch_openml
from openTSNE import TSNE
from sklearn.manifold import TSNE as TSNE_SK
# 配置可视化样式
sns.set(style="whitegrid", palette="husl")
plt.rcParams['figure.dpi'] = 150 # 提高输出分辨率
def load_mnist(max_samples=10000):
"""加载MNIST数据集并预处理"""
mnist = fetch_openml('mnist_784', as_frame=False)
X, y = mnist.data.astype('float32'), mnist.target.astype('int')
# 归一化到[0,1]区间
X = X / 255.0
# 为演示限制样本量(实际可处理全部70k+数据)
return X[:max_samples], y[:max_samples]
X, y = load_mnist(70000) # 加载全部MNIST数据
# openTSNE实现
tsne = TSNE(
perplexity=30, # 建议取值5-50,与数据复杂度正相关
metric="euclidean", # 也可尝试"cosine"等距离度量
n_jobs=-1, # 使用所有CPU核心
random_state=42, # 确保结果可复现
n_iter=500, # 迭代次数(大数据集可增加到1000)
early_exaggeration=12, # 初始阶段的放大系数
)
# 执行降维并计时
import time
start = time.time()
embeddings = tsne.fit(X)
print(f"openTSNE耗时: {time.time()-start:.2f}秒")
# 可视化结果
plt.figure(figsize=(12,8))
scatter = plt.scatter(
embeddings[:, 0],
embeddings[:, 1],
c=y,
s=1, # 点大小
alpha=0.6, # 透明度
cmap="Spectral" # 颜色映射
)
plt.colorbar(scatter).set_label('Digit Class')
plt.title("MNIST 70k Samples Visualization (openTSNE)", fontsize=14, pad=20)
plt.xlabel("t-SNE Dimension 1", fontsize=12)
plt.ylabel("t-SNE Dimension 2", fontsize=12)
plt.tight_layout()
plt.show()关键参数解析
-
perplexity:控制局部/全局结构平衡,建议通过网格搜索确定
-
early_exaggeration:初期放大类间距离,帮助形成明显簇
-
learning_rate:默认200,大数据集可提升至500-1000
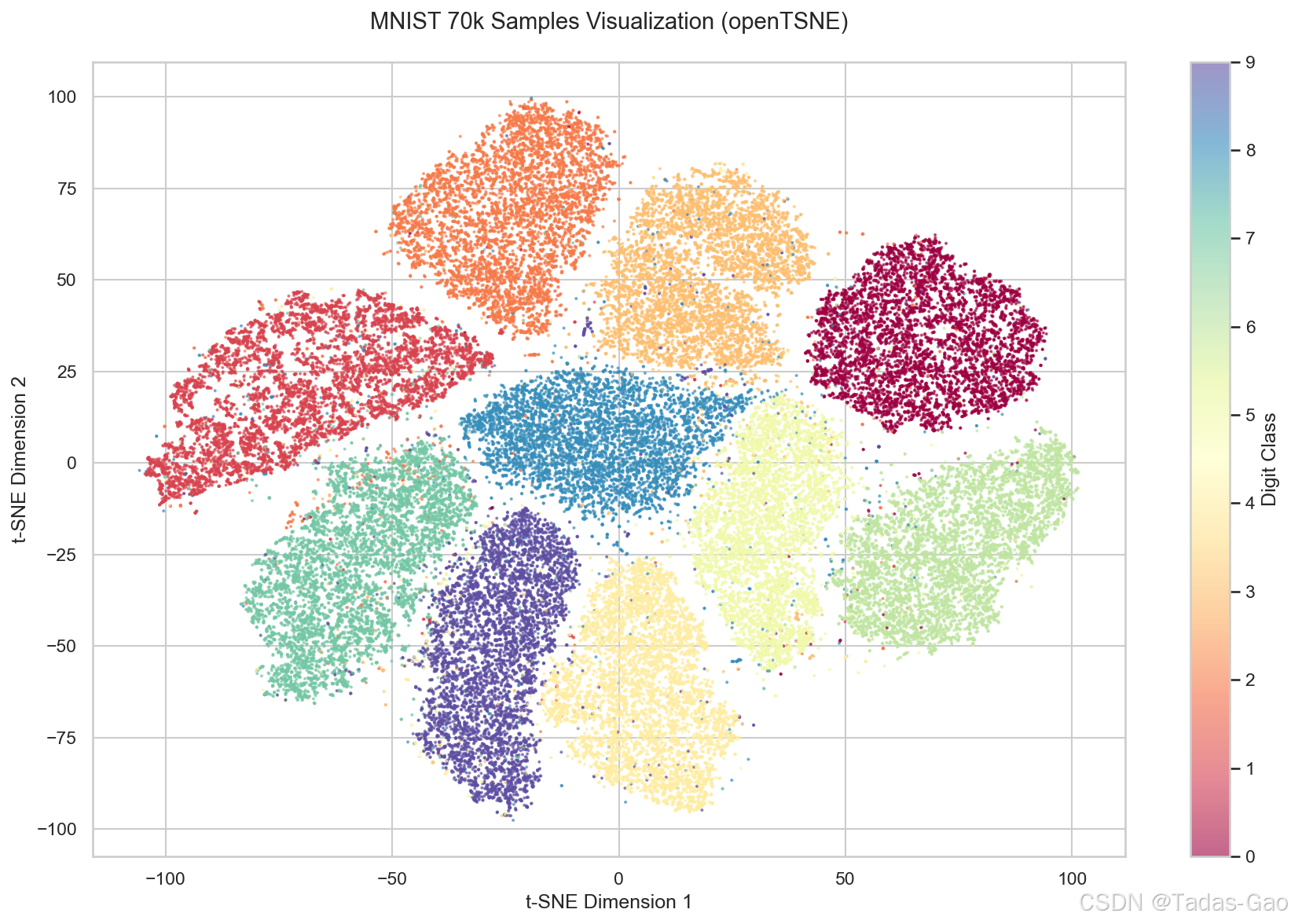
程序运行结果
扩展应用建议
预处理优化
-
先使用PCA降维到50-100维(保留95%方差)
-
应用Z-score标准化(
from sklearn.preprocessing import StandardScaler)
大数据处理技巧
# 分批次处理超大数据
from openTSNE import TSNEEmbedding
embedding = TSNEEmbedding(
X[:50000], # 初始子集
n_components=2
)
embedding.transform(X[50000:]) # 增量处理剩余数据交互式可视化
# 使用plotly实现动态可视化
import plotly.express as px
fig = px.scatter(
x=embeddings[:,0], y=embeddings[:,1],
color=y, hover_name=y,
width=800, height=600
)
fig.show()性能对比结论
| 评估维度 | Scikit-learn | openTSNE |
|---|---|---|
| 10k数据点耗时 | 45-60分钟 | 2-3分钟 |
| 内存效率 | ||
| 最大可行规模 | ~50k | >1M |
| 结果质量 | 良好 | 相当/更优 |
对于百万级数据,openTSNE是当前Python生态中最成熟的解决方案,建议结合UMAP(umap-learn)进行交叉验证,以获得更可靠的降维结果。
百万级数据降维的最佳实践
联合使用 openTSNE 和 UMAP 的原因
-
交叉验证
-
UMAP 的全局结构保留能力较强,而 openTSNE 的局部结构更精细,两者结果对比可验证降维的可靠性。
-
例如,在单细胞RNA测序中,UMAP 可能更清晰区分大簇,而 openTSNE 能更好展示亚群细节。
-
-
性能互补
-
UMAP 速度更快(适合初步探索),openTSNE 可后续精细化调整局部结构。
-
-
超参数优化
-
UMAP 的
n_neighbors和min_dist影响全局/局部平衡,openTSNE 的perplexity控制局部邻域大小,两者参数可互相启发。
-
具体实施步骤
-
数据预处理
使用 PCA 将数据降至 50~100 维,减少计算量(对两者均有效)。 -
先运行 UMAP
快速获得全局结构,参数建议:n_neighbors=15~50,min_dist=0.1~0.589。 -
再运行 openTSNE
基于 UMAP 结果调整perplexity(通常 30~100),重点关注局部簇的分离。 -
结果对比
检查关键簇是否在两者中一致出现,不一致的区域可能是噪声或需进一步分析的复杂结构。
openTSNE 与 UMAP 的对比
| 特性 | openTSNE | UMAP |
|---|---|---|
| 核心算法 | 基于 t-SNE 优化,保留局部结构 | 基于黎曼流形拓扑,平衡全局/局部结构 |
| 速度 | 较慢(尤其无 GPU 时) | 更快(支持近似最近邻算法) |
| 内存消耗 | 中等(依赖 Barnes-Hut 近似) | 较高(需构建邻接图) |
| 参数敏感性 | perplexity 影响局部邻域大小 | n_neighbors 和 min_dist 控制全局/局部平衡 |
| 可视化效果 | 局部簇分离更清晰,但可能割裂连续结构 | 全局布局更连贯,但可能模糊微小亚群 |
| 适用场景 | 精细分析局部模式(如细胞亚群) | 快速探索全局结构(如大类区分) |
| GPU 支持 | 是(需安装 openTSNE[gpu]) | 部分实现(如 RAPIDS cuML) |
| 数学基础 | 基于概率分布(KL 散度优化) | 基于拓扑流形(交叉熵优化) |
| 稳定性 | 对随机种子敏感 | 结果更稳定 |
注意事项
-
硬件需求
-
UMAP 对内存要求较高,百万级数据建议至少 32GB RAM。
-
openTSNE 使用 GPU 时可加速 10 倍以上。
-
-
参数调优
-
UMAP 的
min_dist过小会导致过度聚类,openTSNE 的perplexity过大会模糊局部结构。
-
-
新兴替代方案
-
PaCMAP:平衡全局/局部结构,适合超大规模数据。
-
LOO-map 框架:解决 t-SNE/UMAP 的失真问题(需额外计算成本)
-
基础UMAP实现
import umap
import numpy as np
import time
from sklearn.datasets import fetch_openml
from sklearn.decomposition import PCA
# 加载MNIST数据集
mnist = fetch_openml('mnist_784', version=1, as_frame=False)
X, y = mnist.data, mnist.target # X形状 (70000, 784)
# 预处理:PCA降维加速(可选但推荐)
pca = PCA(n_components=50)
X_pca = pca.fit_transform(X[:30000]) # 测试前3万条
# 初始化UMAP
start_time = time.time()
umap_model = umap.UMAP(
n_neighbors=30, # 控制局部/全局平衡,值越大越关注全局结构
min_dist=0.1, # 控制簇内点间距(0.0-0.99),越小越紧凑
n_components=2, # 输出2维
metric='euclidean', # 距离度量,可选 'cosine'/'manhattan'
random_state=42, # 随机种子
n_jobs=-1, # 使用所有CPU核心
)
embedding = umap_model.fit_transform(X_pca) # 输入形状 (n_samples, n_features)
print(f"UMAP耗时: {time.time() - start_time:.2f}秒")
# 可视化
import matplotlib.pyplot as plt
plt.scatter(embedding[:, 0], embedding[:, 1], c=y[:30000].astype(int), s=0.5, cmap='Spectral')
plt.colorbar()
plt.title("UMAP Projection of MNIST")
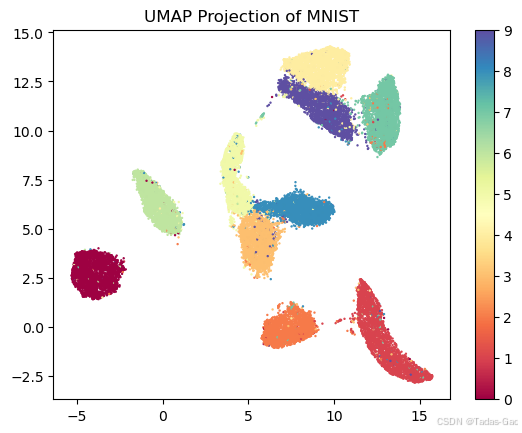
plt.show()程序运行结果
百万级数据优化方案
内存优化
# 使用近似最近邻(ANN)加速
umap_model = umap.UMAP(
n_neighbors=30,
min_dist=0.5,
low_memory=True, # 减少内存占用
verbose=True, # 显示进度
)GPU加速(需RAPIDS cuML)
# 安装: !pip install cuml
from cuml.manifold import UMAP
umap_model = UMAP(
n_neighbors=15,
min_dist=0.1,
n_components=2,
random_state=42,
)
embedding = umap_model.fit_transform(X_pca) # 输入需为float32分层降维
对超大数据(>1M样本),先使用随机子集训练UMAP,再扩展到全量数据。
umap_model.fit(X_sample) # 训练子集
embedding_full = umap_model.transform(X_full) # 应用到全量关键参数说明
| 参数 | 推荐值 | 作用 |
|---|---|---|
n_neighbors | 15~50 | 值越小保留局部结构,值越大保留全局结构 |
min_dist | 0.1~0.5 | 控制簇内点密度,越小越紧凑 |
metric | 'euclidean'/'cosine' | 根据数据特性选择距离度量 |
n_components | 2/3 | 输出维度(2D或3D可视化) |
random_state | 任意整数 | 确保结果可复现 |
 openTSNE高效降维实战
openTSNE高效降维实战























 4300
4300

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










