🧬 推荐开源项目:Bio-transformers —— 蛋白质语言模型的强大工具
项目介绍
bio-transformers 是一个基于 ESM/Protbert 模型的 Python 封装库,这些模型是经过数百万蛋白质训练的 Transformers 蛋白质语言模型,用于预测嵌入向量。通过简单易用的接口,该项目充分利用了这些最先进的模型,提供了丰富的实用功能。
该包提供了一个统一的接口来使用所有这些模型——我们称之为 后端。例如,您将能够计算天然氨基酸的概率、嵌入向量,或者使用 Ray 在多 GPU 上轻松微调您的模型。
📕 更多详细信息请参考官方文档。
原始模型仓库:
项目技术分析
什么是 Transformer?
Transformer 是一种 AI 语言模型,在语音识别和自动化聊天机器人领域产生了巨大影响,现在已被指出是理解和设计蛋白质的革命性技术。它们在论文 Attention is all you need 中被引入。这篇博客文章可以为您提供关于 Transformer 架构及其背后数学的深入理解。
当用大量数据在特定语言(如英语)上进行训练时,模型会学习单词和语法,并在提示时生成完整的句子。例如:
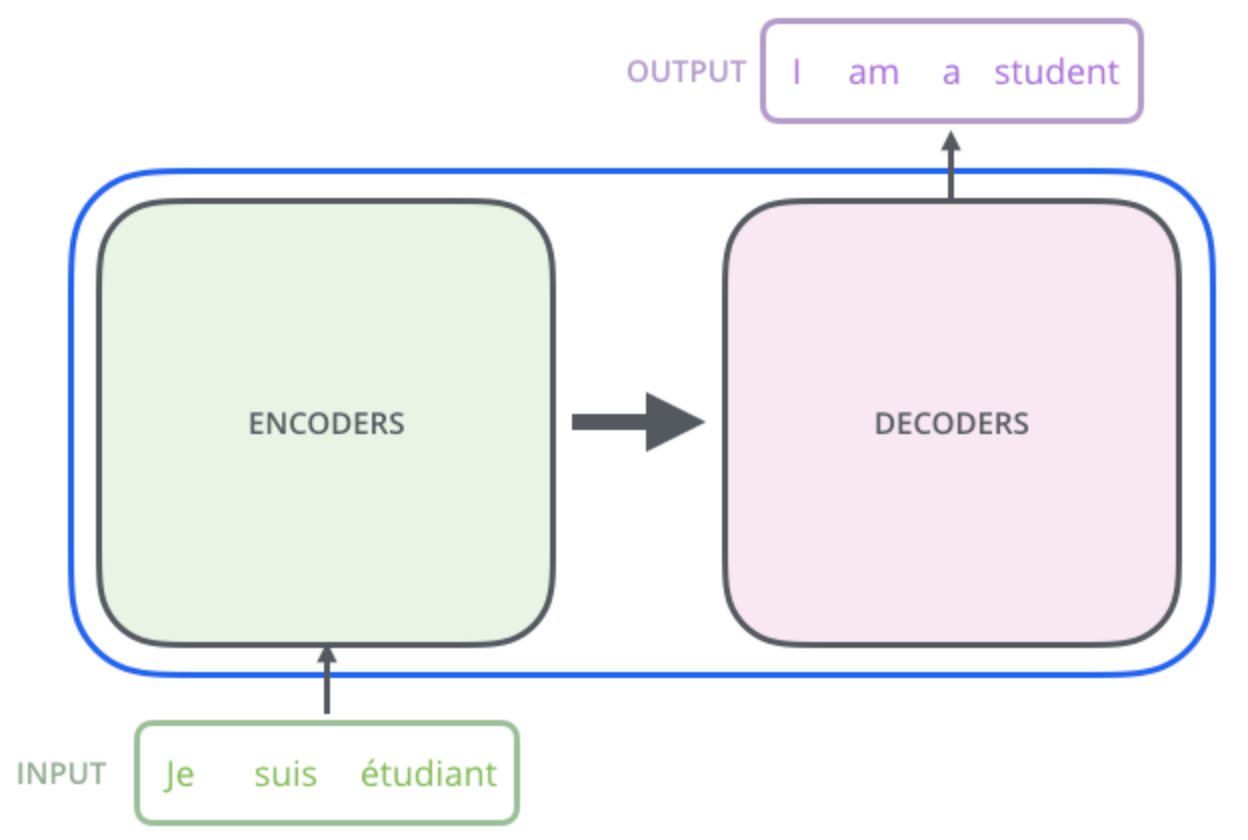
为什么用 Transformer 处理蛋白质?
蛋白质是所有生物体中执行关键功能的分子,由一个或多个氨基酸链组成。只有 20 种不同的氨基酸,它们的不同组合在人类中形成了数千种功能性蛋白质。如果我们把氨基酸看作构成蛋白质的“单词”,那么蛋白质就是“句子”,我们可以使用 Transformer 来理解蛋白质的语言。当用迄今为止在多个物种中识别的数十亿蛋白质序列进行训练时,Transformer 能够理解哪些氨基酸序列从语言角度是有意义的,并提出新的组合。
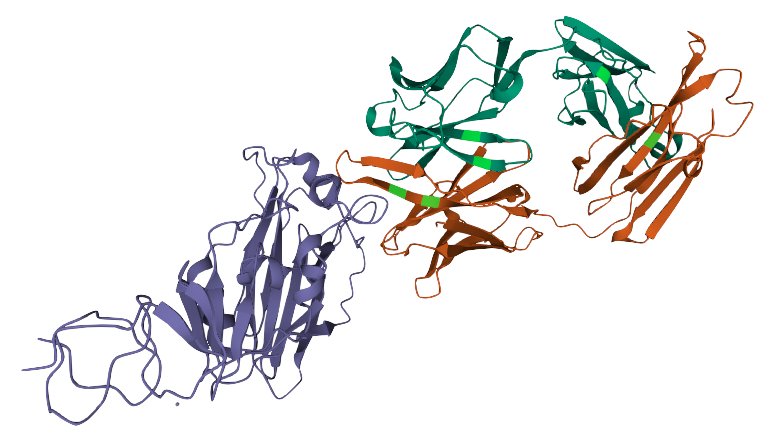
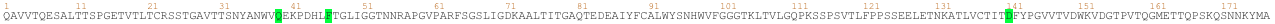
查询在蛋白质语言上训练的 Transformer 可以提供关于该蛋白质的丰富信息。如上图所示,Transformer 可以告诉您哪些氨基酸可能是关键,并需要存在于感兴趣的蛋白质中。这些信息在尝试理解对蛋白质功能或稳定性可能至关重要的氨基酸区域时特别有意义。
项目及技术应用场景
- 蛋白质嵌入向量计算:通过将氨基酸序列转换为低维向量,便于进行可视化、数学运算和分析。
- 氨基酸概率预测:评估特定氨基酸在给定上下文中的出现概率,帮助理解蛋白质结构和功能。
- 蛋白质稳定性评估:通过计算伪对数似然度,估计突变蛋白质的“自然性”和可产生性。
- 模型微调:在特定数据集上微调预训练模型,提升模型在特定任务上的表现。
项目特点
- 统一接口:支持多种后端模型(如 ESM、Protbert),简化使用流程。
- 多 GPU 支持:利用
Ray实现 multi-GPU 加速,提升计算效率。 - 易于安装和使用:提供详细的文档和示例代码,快速上手。
- 开源免费:遵循 Apache 2.0 许可证,完全开源,免费使用。
安装与使用
使用 conda 环境
conda create --name bio-transformers python=3.7 -y && conda activate bio-transformers
- 安装包:
pip install bio-transformers
快速开始
from biotransformers import BioTransformers
BioTransformers.list_backend()
>>
使用以下后端:
* esm1_t34_670M_UR100
* esm1_t6_43M_UR50S
* esm1b_t33_650M_UR50S
* esm_msa1_t12_100M_UR50S
* protbert
* protbert_bfd
计算嵌入向量
sequences = [
"MKTVRQERLKSIVRILERSKEPVSGAQLAEELSVSRQVIVQDIAYLRSLGYNIVATPRGYVLAGG",
"KALTARQQEVFDLIRDHISQTGMPPTRAEIAQRLGFRSPNAAEEHLKALARKGVIEIVSGASRGIRLLQEE",
]
bio_trans = BioTransformers(backend="protbert")
embeddings = bio_trans.compute_embeddings(sequences, pool_mode=('cls','mean'), batch_size=2)
cls_emb = embeddings['cls']
mean_emb = embeddings['mean']
多 GPU 支持
import ray
ray.init()
bio_trans = BioTransformers(backend="protbert", num_gpus=2)
embeddings = bio_trans.compute_embeddings(sequences, pool_mode=('cls','mean'), batch_size=2)
计算伪对数似然度
sequences = [
"MKTVRQERLKSIVRILERSKEPVSGAQLAEELSVSRQVIVQDIAYLRSLGYNIVATPRGYVLAGG",
"KALTARQQEVFDLIRDHISQTGMPPTRAEIAQRLGFRSPNAAEEHLKALARKGVIEIVSGASRGIRLLQEE",
]
bio_trans = BioTransformers(backend="protbert", num_gpus=1)
loglikelihood = bio_trans.compute_loglikelihood(sequences)
微调预训练模型
import biodatasets
import numpy as np
from biotransformers import BioTransformers
import ray
data = biodatasets.load_dataset("swissProt")
X, y = data.to_npy_arrays(input_names=["sequence"])
X = X[0]
# 训练短序列
length = np.array(list(map(len, X))) < 200
train_seq = X[length][:15000]
ray.init()
bio_trans = BioTransformers("esm1_t6_43M_UR50S", num_gpus=1)
bio_trans.finetune(train_seq)
总之,bio-transformers 是一个功能强大且易于使用的工具,适用于蛋白质语言模型的研究和应用。无论您是生物信息学研究人员还是机器学习工程师,这个项目都能为您的工作提供强大的支持。立即尝试,开启您的蛋白质研究新旅程!🚀
创作声明:本文部分内容由AI辅助生成(AIGC),仅供参考







 项目地址: https://gitcode.com/gh_mirrors/bi/bio-transformers
项目地址: https://gitcode.com/gh_mirrors/bi/bio-transformers 



















 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










