Tangram:单细胞空间映射的强大工具
项目介绍
Tangram 是一个基于 PyTorch 和 scanpy 的 Python 包,专门用于将单细胞(或单核)基因表达数据映射到空间基因表达数据上。该工具适用于从相同解剖区域或组织类型收集的数据,理想情况下是生物学重复样本,并且需要共享一组基因。Tangram 通过在共享基因上拟合基因表达,将单细胞数据在空间上对齐。为了更好地了解 Tangram 的工作原理和使用方法,建议查看我们的教程和文档。
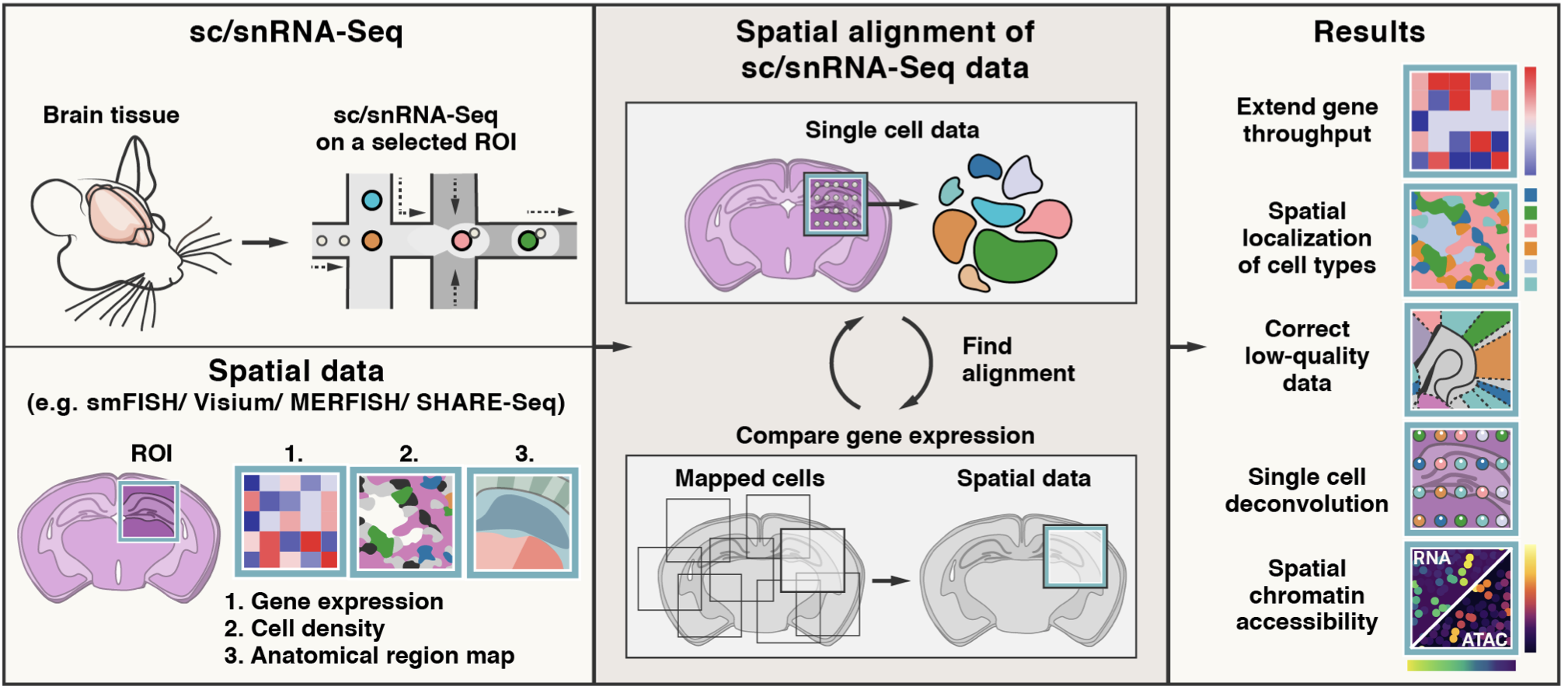
项目技术分析
技术架构
Tangram 的核心技术架构基于 PyTorch 和 scanpy,利用深度学习技术来实现单细胞数据与空间数据的映射。其主要步骤包括:
- 数据预处理:使用
tg.pp_adatas函数对单细胞数据和空间数据进行预处理,找出两者之间的共享基因。 - 映射计算:通过
tg.map_cells_to_space函数计算单细胞数据在空间上的映射矩阵。 - 基因表达投影:使用
tg.project_genes函数将单细胞的基因表达投影到空间数据上。
算法原理
Tangram 的核心算法通过最大化单细胞数据与空间数据之间的余弦相似度来计算映射矩阵。具体来说,Tangram 通过以下公式来优化映射矩阵:
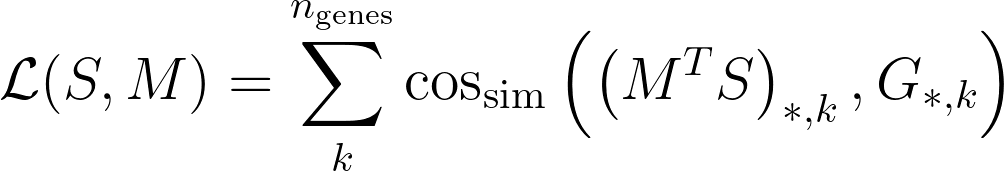
其中,cos_sim 表示余弦相似度。该公式的意义在于,映射后的单细胞基因表达应尽可能接近空间数据中的基因表达。
项目及技术应用场景
应用场景
Tangram 在以下场景中具有广泛的应用潜力:
- 空间转录组学研究:通过将单细胞数据映射到空间数据上,研究人员可以更好地理解基因表达在组织中的空间分布。
- 细胞类型注释:Tangram 可以将单细胞数据中的细胞类型信息投影到空间数据上,从而实现空间分辨率的细胞类型注释。
- 基因表达校正:通过将高质量的单细胞数据投影到空间数据上,可以校正空间数据中的基因表达缺失或低质量数据。
技术优势
Tangram 的技术优势主要体现在以下几个方面:
- 高效性:基于 PyTorch 的深度学习框架,Tangram 能够高效地处理大规模的单细胞和空间数据。
- 灵活性:Tangram 支持在细胞级别和集群级别进行映射,适应不同的数据处理需求。
- 易用性:通过简单的 API 和详细的文档,用户可以快速上手并应用 Tangram 进行数据分析。
项目特点
主要特点
- 深度学习驱动:Tangram 利用深度学习技术,能够更准确地映射单细胞数据到空间数据上。
- 多层次映射:支持细胞级别和集群级别的映射,满足不同精度和计算资源的需求。
- 开源社区支持:作为一个开源项目,Tangram 得到了广泛的开源社区支持,用户可以轻松获取帮助和资源。
未来发展
Tangram 作为一个新兴的单细胞空间映射工具,未来有望在以下方面进一步发展:
- 算法优化:通过引入更多的先验知识,进一步优化映射算法的准确性和效率。
- 多模态数据融合:支持更多类型的数据输入,如多模态数据(基因表达、蛋白质表达等)的融合分析。
- 用户友好性:通过开发更多的可视化工具和交互式界面,提升用户体验。
结语
Tangram 是一个功能强大且易于使用的单细胞空间映射工具,适用于各种空间转录组学研究。无论你是初学者还是资深研究人员,Tangram 都能为你提供高效、准确的数据分析支持。快来尝试 Tangram,开启你的单细胞空间映射之旅吧!
创作声明:本文部分内容由AI辅助生成(AIGC),仅供参考







 项目地址: https://gitcode.com/gh_mirrors/tang/Tangram
项目地址: https://gitcode.com/gh_mirrors/tang/Tangram 


















 809
809

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










