胰腺分割的3D分割与提取框架
摘要
定位与分割(LAS)框架是用于从腹部CT中分割胰腺的有效方法。由粗到精是最广泛使用的LAS框架,结合多种网络架构在胰腺分割任务中取得了优异的结果。然而,胰腺区域定位的不准确性会降低LAS方法的性能。为解决这些问题,我们提出了分割与提取(SAP)框架,该框架在训练过程中利用人工标注直接计算胰腺的感兴趣区域(ROI),并训练神经网络在该ROI内进行胰腺分割。在测试过程中,我们首先使用训练好的分割网络对整个CT扫描进行胰腺分割,然后采用区域增长法从噪声中提取最终的分割结果。我们在NIH数据集上结合ResNet与SAP框架进行了实验,获得了86.96的DSC分数,证明了我们的SAP框架在胰腺分割任务上的表现优于常规的LAS框架。
关键词 - 分割,卷积神经网络,胰腺,分割与拾取
I. 引言
通过计算机断层扫描(CT)进行胰腺分割是计算机辅助诊断中的重要前提,因为准确的胰腺分割有助于临床医生精确定义胰腺病变。为了帮助医生在复杂的医学影像中快速定位胰腺并有效评估相关疾病,准确且高效的胰腺分割方法具有重要的临床应用价值。
当前大多数胰腺分割任务的解决方案依赖于神经网络。在所有基于深度学习神经网络(CNNs)的胰腺分割方法中,多数研究集中在改进网络架构。Roth et al. [1]使用整体多视图网络对胰腺进行粗略定位。在分割过程中采用整体多视图深度网络(HNNs),并结合多尺度方法。NBO et al. [2]提出使用U-Net 2阶段在三维CT体积上定位胰腺区域。Sidike et al.[3]在U-Net [4],中引入了注意力机制,有效提高了胰腺分割的准确性。另一方面,一些工作关注多个网络协同工作的方案。通常这些方法将分割过程分为两个阶段:第一个阶段是定位感兴趣区域(ROI);第二个阶段是在该感兴趣区域内完成胰腺分割。我们将这种两阶段方法称为定位与分割(LAS)框架。对于胰腺的粗略定位,已有多种方法被应用。最常用的LAS框架基于由粗到精(C2F),涉及两个网络:其中一个网络用于在整个CT体积上分割胰腺,并根据分割结果定位感兴趣区域(粗分割)。第二个网络用于在感兴趣区域中分割胰腺,以有效提升第二个网络的性能。Zhou et al. [5]结合三维深度网络与C2F框架进行胰腺分割,取得了最先进的结果。Yue et al. [6]在C2F框架中融合注意力机制U-Net,在NIH数据集上达到了85.9%的DSC。
LAS框架存在两个问题。第一,由于感兴趣区域的定义为最小立方体,与胰腺的实际形状不符,因此最小立方体ROI可能包含大量与胰腺无关的体素,由这些体素训练的模型可能导致过多与胰腺无关的响应,容易导致假阳性。第二,ROI不准确。立方体ROI的定位方法主要依赖于边界框预测,而边界框预测常常存在误差。此外,胰腺是腹腔中最难分割的器官之一,这进一步增加了ROI定位的难度。立方体ROI的形状与定位方法使得LAS框架不够准确且不可靠。
为了克服LAS框架的缺陷,本文提出了一个用于胰腺分割的分割与提取(SAP)框架。我们基于每个体素的人工标注,构建了精确轮廓的ROI。轮廓ROI中体素与胰腺的重叠度接近100%。这使得轮廓ROI在空间上的体素与胰腺实际分布的重叠比立方体ROI更高更精确。在训练阶段,我们使用真实标签计算ROI,从而保证了精确的ROI用于训练。在测试过程中,真实标签不可用,但我们使用独立网络来提取分割结果。这就是分割与提取过程。
最后,我们在Zhou等[5],的基础上提出了长程残差网络(LS-ResNet),并在NIH胰腺分割数据集上验证了我们的方法。该方法的Dice相似系数(DSC)为86.96%,优于基于LAS框架的方法。
II. 方法
A. 分割与提取框架
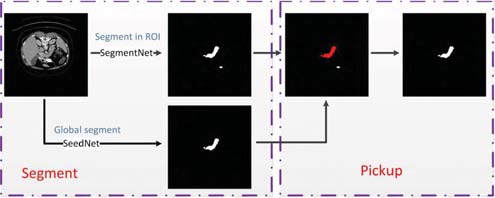
我们提出的SAP框架包含两个阶段。第一阶段是分割胰腺,第二阶段是从粗糙的分割结果中提取更精确的分割结果。分割与提取框架流程见图1。为了实现该框架,我们设计了两个独立的胰腺分割网络。其中一个是用于精确分割胰腺的网络,我们称之为SegNet。SegNet在训练过程中仅使用胰腺内部的真实标签,从而避免背景体素干扰对胰腺边缘体素的分类,减少了因背景不平衡问题带来的影响。然而,这会导致SegNet在整幅CT扫描上分割胰腺时可能出现较多噪声。因此,我们使用SeedNet从整幅的CT扫描中分割出相对粗糙但召回率较高的结果。利用SeedNet的分割结果,我们通过区域生长方法从SegNet的分割结果中提取更精确的胰腺分割结果。
SAP框架不会引入非胰腺体素,从而使SegNet的训练过程不受背景不平衡问题的影响。SeedNet不需要精确分割胰腺,背景不平衡问题对其影响较小。我们使用标准的交叉熵损失函数来训练所有网络,如下所示:
$$ \mathcal{L} = -\sum_{i} Z_i \log(Y_i) $$
其中,$Z_i$ 是体素 $i$ 的真实标签,$Y_i$ 是预测值,$\theta$ 是待优化的网络参数集合。
SeedNet 我们将SeedNet建模为 $ P_{seed} = \text{Net} {seed}(Y|\theta) $,其中Y是从整个CT扫描中采样的体素。$\theta$ 表示网络参数,$P {seed}$ 表示次级预测。该阶段的目标是从输入图像中高效重建出粗糙的次级分割$P_{seed}$,并提取出大致完整的胰腺区域,从而获得完整的胰腺体素。该完整胰腺体素被视为种子区域。
SegNet 我们将SegNet建模为 $ P_{seg} = \text{Net}_{seg}(Y, \text{ROI}_d|\theta) $,SegNet在训练阶段与测试阶段有不同的采样策略。在训练阶段,体素从轮廓ROI内进行采样:
$$ \mathcal{L} {seg} = -\sum {Y \in \text{ROI} d} Z_Y \log(P {seg}(Y)) $$
其中Y是CT扫描中的一个体素,距离函数$D(Y)$用于计算体素Y到胰腺的距离,若Y是胰腺内的体素,则$D(Y) = 0$。SegNet的目标是在胰腺区域内更精确地分割胰腺。SegNet的损失函数在ROI$_d$中计算:
$$ \mathcal{L} {seg} = -\sum {Y \in \text{ROI} d} Z_Y \log(P {seg}(Y)) $$
B. 网络架构
SegNet与SeedNet具有不同的架构,见图2,网络的编码器部分是收缩路径,用于提取深层特征,并在多次下采样后降低特征分辨率。网络的解码器部分是扩张路径,旨在将深层特征逐步恢复到原始二维或三维,并在恢复过程中增加特征分辨率,最终在网络的最后一层输出与输入数据尺寸相匹配的分割结果。借鉴U-Net与ResNet [8],的设计,我们在网络架构中引入了长程与短程跳跃连接,我们称这种网络架构为长程残差网络(LS-ResNet)。
LS-ResNet的编码器由卷积模块与下采样层组成。每个卷积模块包含一个卷积核为3×3×3的三维卷积层,后接一个批归一化层和一个ReLU激活层。我们使用最大池化层作为下采样层。每个下采样层的卷积核大小为2×2×2,步长为2。我们在第一个与第二个下采样层之后引入了残差跳跃连接。第一个下采样层的输出通过该跳跃连接传递到第二个下采样层的输入上,依此类推,第二个下采样层的输出可传递到第三个下采样层的输入上。我们使用这些残差跳跃连接来融合多个输出,从而在深层特征中保留更多细节。
解码器部分由反卷积层与跳跃连接组成。反卷积层的卷积核大小为4×4×4,步长为2。我们在编码器与解码器之间引入了一个长距离跳跃连接。编码器输出的特征图与反卷积输出进行拼接。



III. 实验
A. 数据集与实验设置
我们的实验在NIH胰腺数据集上进行,该数据集包含82例增强腹部CT扫描。CT切片中的像素值被截取至[-100, 240] HU,并归一化到[0, 1]。在训练阶段的数据增强中,我们在所有训练样本上采用了几种常用且有效的增强方法,包括旋转(±90°、180°和270°)、沿三个轴(轴向、冠状面和矢状面)的翻转,并保持所有扫描的分辨率不变。我们对82例病例进行了五折交叉验证进行训练与测试。本文使用Dice相似系数(DSC)、Jaccard指数和精确率来评估分割性能。
所有实验均在NVIDIA GTX 1080Ti GPU上进行,深度神经网络基于PyTorch框架实现。体素块大小为64×64×64,训练与测试阶段的步长均为8×8×8。在训练过程中,我们分两阶段训练网络:首先使用增强数据进行训练,初始学习率为0.001,经过4个训练周期后降至0.0001,总训练周期=16,批量大小=16,轮廓ROI扩展距离=16,优化器为Adam。
在测试阶段,我们令输出块大小为16×16×16,并以步长8×8×8进行拼接,以获得最终预测结果。
B. 消融研究
我们在图4中展示了四个样本的分割结果。第一列展示了SegNet的结果,胰腺主体区域出现了较多噪声。第二列展示了SeedNet的结果,胰腺区域虽较完整但边界模糊。第三列是SAP的最终结果,去除了噪声且边界清晰,显示出更高的精度。
表I比较了SAP中三个阶段的结果。从第一阶段提取的胰腺分割结果具有最佳性能。
| 方法 | DSC(%) | Jaccard(%) | 精确率(%) |
|---|---|---|---|
| SegNet | 81.71 ± 5.27 | 68.37 ± 5.38 | 84.62 ± 4.96 |
| SeedNet | 84.12 ± 4.92 | 71.81 ± 4.92 | 85.92 ± 4.26 |
| Ours (SAP) | 86.96 ± 3.85 | 74.62 ± 4.66 | 88.21 ± 4.45 |
与LAS框架的比较:表II对比了基线分割结果与所提方法的性能。基线分割结果考虑了相同网络结构下的性能上限,使我们能准确评估网络框架本身的性能。表II中所有方法均采用了LAS框架,最后一行为我们的结果。我们发现,所提出的SAP框架取得了最优性能。在与基线相同的网络架构下,SAP框架相比C2F [2] 的DSC提升了2.1%。
| 方法 | 架构 | DSC(%) | Jaccard(%) |
|---|---|---|---|
| Zhou et al. [5] | 3D ResNet | 84.06 | 66.80 |
| NBO et al. [2] | 2D U-Net | 86.25 | 74.17 |
| Chu et al. [9] | 3D U-Net | 86.96 | 74.62 |
| Li et al. [10] | 2D U-Net | 84.59 | — |
| 基线 | 3D ResNet | 85.90 | 75.70 |
| 提出的方法 | 3D LS-ResNet | 86.96 | 74.62 |
注:U-Net = 多尺度U-Net
比较轮廓ROI与立方体ROI:我们在本部分比较使用轮廓ROI与立方体ROI进行胰腺分割的性能。大小为d的立方体ROI是大小为d的轮廓ROI的最小外接立方体。本实验中的ROI基于真实标签计算,因此这两个ROI的定位是准确的。分割性能的差异完全源于感兴趣区域的形状差异。我们在基线架构上进行了该实验。
图4显示了在不同扩展距离下轮廓ROI与立方体ROI的分割DSC。当d=12时,立方体ROI达到最佳分割性能,最高DSC为85.23%;而对于轮廓ROI,d=16时性能最佳,最高DSC为86.25%。因此,我们得出结论:在网络用于胰腺分割时,轮廓ROI的表现优于立方体ROI。
C. 与最先进方法的比较
表III比较了所提方法与六种最先进方法的分割性能。其中一些方法使用了多视图融合[5]、注意力机制[3]和多尺度方法[2]。我们提出的方法(LS-ResNet + SAP)相较于多数方法具有优势。Li等人[10]通过精心设计2D U-Net的多尺度卷积模块,取得了比我们方法更高的DSC,这表明我们的方法仍有提升空间,但我们也说明了框架改进是整体胰腺分割方法进步的一个方向,未来可在网络架构上进行更深入探索。
| 方法 | DSC(%) | Jaccard(%) | 精确率(%) |
|---|---|---|---|
| Zhou et al. [5] | 84.57 | 79.70 | — |
| Yue et al. [6] | 85.90 | 75.70 | 87.60 |
| NBO et al. [2] | 85.22 | 78.77 | 86.63 |
| Lipskoch et al. [11] | 84.59 | — | — |
| Chen et al. [9] | 85.43 | 74.62 | 88.21 |
| Li et al. [10] | 87.57 | — | — |
| 提出的方法 (Ours) | 86.96 | 74.62 | 88.21 |
IV. 结论与未来工作
本文提出了一种用于CT体积胰腺分割的SAP框架。该框架包含两个网络:SegNet用于在精确轮廓ROI内进行精细分割,SeedNet用于生成高召回率的初始分割结果,二者协同工作,最终获得比任一单独网络更精确的分割结果。通过实验,我们证明了所提出的SAP框架能够比广泛使用的LAS框架更准确地分割胰腺。同时我们在实验中也发现,某些先进网络架构即使在简单框架下也能表现出优异的性能,这使我们意识到合适的框架与优秀的网络结构结合才能实现最佳效果。
在未来工作中,我们将进一步探索更先进的网络架构改进,尤其是在注意力机制、动态卷积和自监督预训练方面的集成,以进一步提升胰腺分割的鲁棒性与泛化能力。





















 1万+
1万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








