之前我们有介绍过使用Xenium Explorer对一张Xenium芯片上多个样本数据进行拆分或者在组织上圈选ROI,基本上步骤就是在Xenium Explorer里手动圈好样本/ROI → Export Cell Stats CSV → pandas 清洗 → 再 merge进adata.obs,这样的话就可以在adata的metadata里添加一列信息用于区分样本/ROI。
Xenium空间转录组实战 | Xenium Explorer 多样本拼片拆分 & ROI 区域圈选
但是经常有小伙伴反馈导出圈选细胞信息的csv后,读取步骤多、列名对不齐、CSV 里 cell_id 格式偶尔还带前缀,容易报错。这里我们可以使用SpatialData包中的一个现成函数match_sdata_to_table(),直接就能把样本/ROI标签直接写到单细胞metadata里。
1. Xenium Explorer圈选区域基操
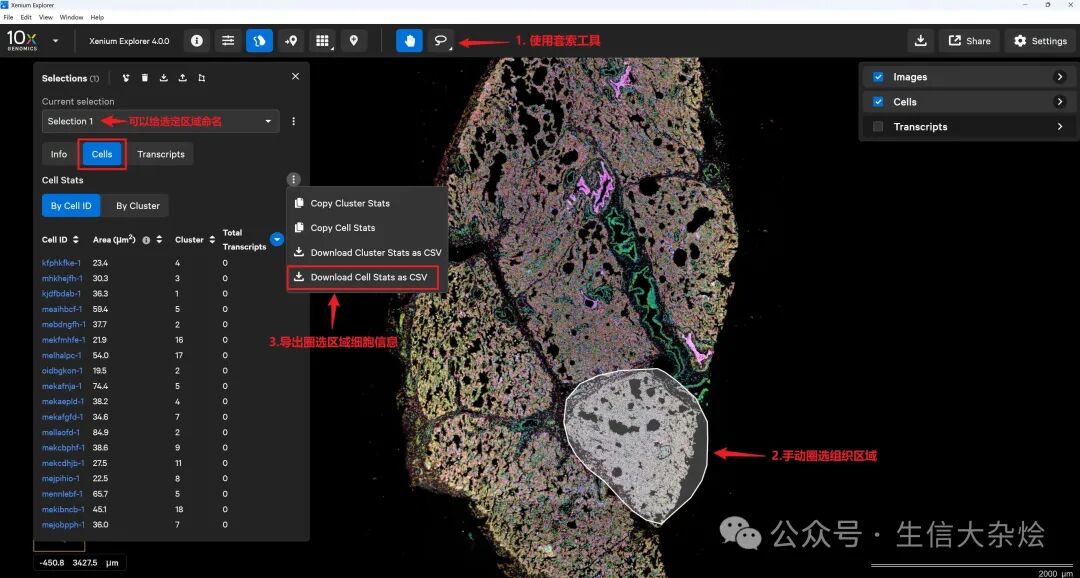
2. 也可以同时圈选多个区域
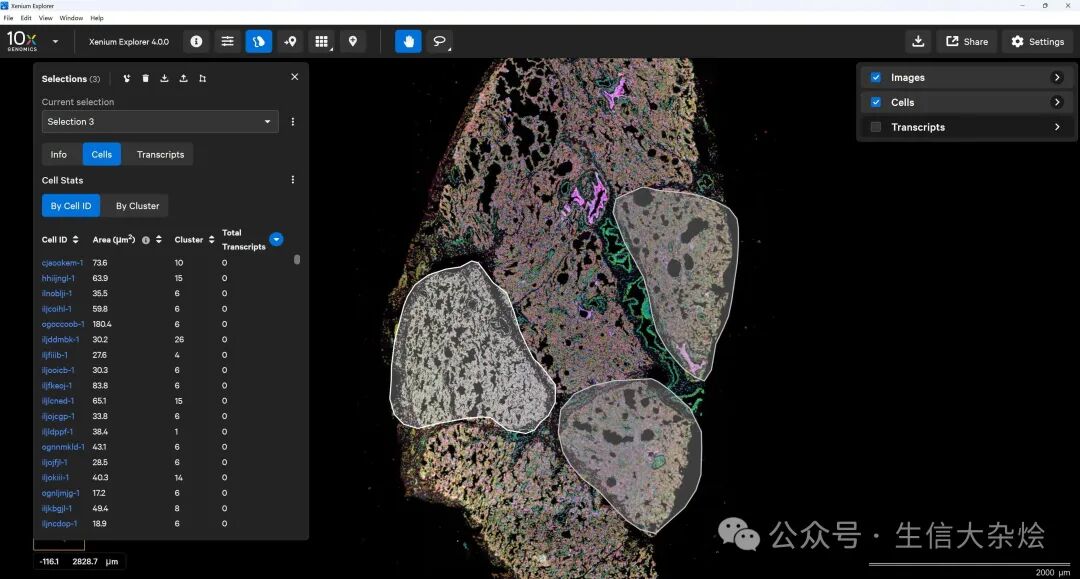
3. 导出圈选区域细胞信息
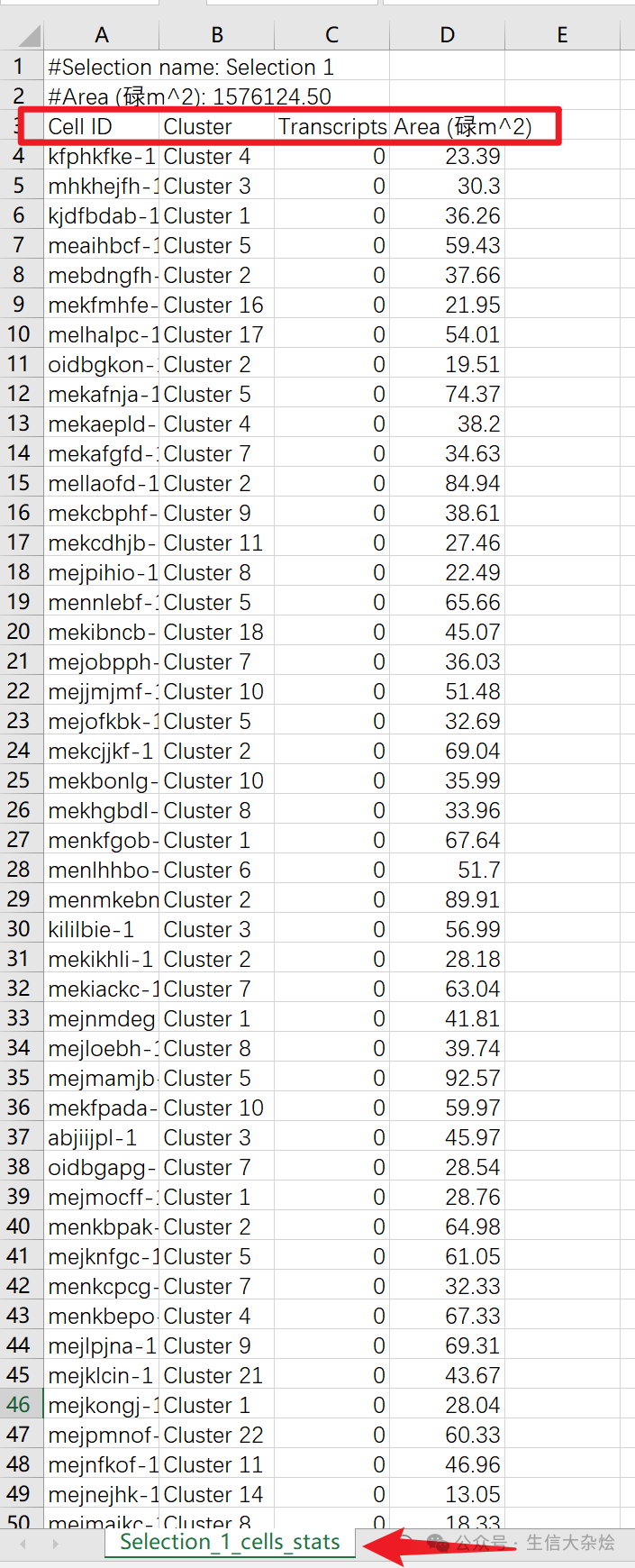
每个圈选区域能导出一个csv文件,内容如下,一共四列信息,包含圈选区域内的所有细胞信息:
4. Spatialdata直接处理圈选细胞csv文件
import pandas as pd
import scanpy as sc
import spatialdata as sd
from spatialdata_io import xenium
# 首先读取整张芯片数据
sdata = xenium(
path='output-XETG00077__0040306__11155-TF-1_ROI_A2__20240621__173354',
cells_boundaries=True,
nucleus_boundaries=True,
cells_as_circles=None,
cells_labels=True
)
整张芯片上所有细胞信息
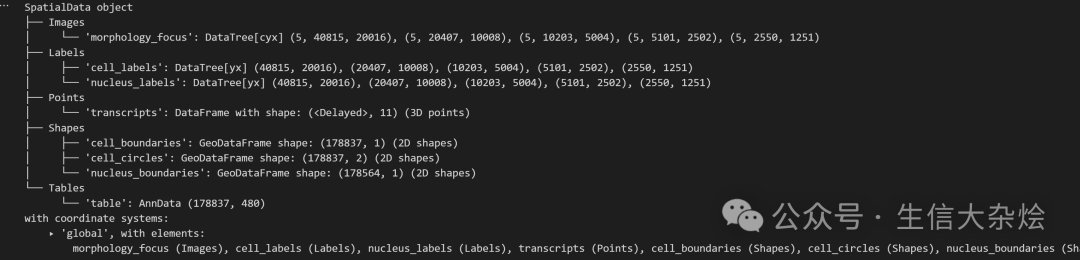
# 读取圈选的细胞信息(这里演示读取圈选的第一个区域细胞)
select_cells = pd.read_csv('Selection_1_cells_stats.csv', skiprows=2)
subset_sdata = sd.match_sdata_to_table(
sdata,
table=sdata["table"][sdata["table"].obs["cell_id"].isin(select_cells["Cell ID"])],
table_name="Sample1",
how="right",
)
adata_sample1 = subset_sdata.tables['Sample1']
Sample1样本的adata就得到了

🎯 常见问题
Q1. 圈完 ROI 忘记保存怎么办?
在 Explorer 里 Ctrl+S 或点击“Save Project”即可,Zones 会自动写进 .xenium 文件夹。
Q2. 为什么我的报错SpatialData包没有match_sdata_to_table函数?
SpatialData包需要升级到0.4.0版本,pip install spatialdata==0.4.0





















 1457
1457

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








