论文:https://arxiv.org/abs/2304.12620
看了一圈pull request,感觉网友在复现时遇到的问题很多,其中一个可能比较有帮助的pull request是这个:
验证集的iou值太低了 · Issue #67 · KidsWithTokens/Medical-SAM-Adapter · GitHub
虽然作者告诉了使用自己的数据集进行训练的方法,但我认为一上来就直接测自己的数据集会出比较多问题,还是先从复现它的2D图像开始。
配置环境
conda env create -f environment.yml(若不成功,则:① 根据environment.yml的内容先装python=3.10;② 装pytorh和torchvision,注意要根据自己的cuda版本进行调整https://download.pytorch.org/whl/torch_stable.htmltorch==1.12.1+cu102 torchvision==0.13.1+cu102 ;③ pip剩余的包:aiosignal==1.2.0 alembic==1.10.4 appdirs==1.4.4 astor==0.8.1 asttokens==2.2.1 backcall==0.2.0 beautifulsoup4==4.12.2 blinker==1.6.2 cachetools==4.2.2 certifi==2022.12.7 charset-normalizer==2.0.4 click==8.1.3 cmaes==0.9.1 colorama==0.4.6 colorlog==6.7.0 contextlib2==21.6.0 coverage==6.5.0 coveralls==3.3.1 cucim==23.4.1 cycler==0.11.0 databricks-cli==0.17.7 docker==6.1.1 docopt==0.6.2 einops==0.6.1 entrypoints==0.4 exceptiongroup==1.1.1 executing==1.2.0 filelock==3.12.0 fire==0.5.0 flask==2.3.2 fonttools==4.25.0 future==0.18.3 gdown==4.7.1 gitdb==4.0.10 gitpython==3.1.31 google-auth==2.6.0 google-auth-oauthlib==0.4.4 greenlet==2.0.2 gunicorn==20.1.0 h5py==3.8.0 huggingface-hub==0.14.1 iniconfig==2.0.0 ipython==8.13.1 itk==5.3.0 itk-core==5.3.0 itk-filtering==5.3.0 itk-io==5.3.0 itk-numerics==5.3.0 itk-registration==5.3.0 itk-segmentation==5.3.0 itsdangerous==2.1.2 jedi==0.18.2 jinja2==3.1.2 json-tricks==3.16.1 jsonschema==4.17.3 kornia==0.4.1 lmdb==1.4.1 lucent==0.1.0 mako==1.2.4 mlflow==2.3.1 nibabel==5.1.0 ninja==1.11.1 nni==2.10 nptyping==2.5.0 opencv-python==4.7.0.72 openslide-python==1.1.2 optuna==3.1.1 partd==1.2.0 pluggy==1.0.0 pooch==1.4.0 prettytable==3.7.0 prompt-toolkit==3.0.38 psutil==5.9.5 pyarrow==11.0.0 pyasn1==0.4.8 pyasn1-modules==0.2.8 pydicom==2.3.1 pygments==2.15.1 pynrrd==1.0.0 pyqt5-sip==12.11.0 pyrsistent==0.19.3 pytest==7.3.1 pytest-mock==3.10.0 pythonwebhdfs==0.2.3 pytorch-ignite==0.4.10 querystring-parser==1.2.4 regex==2023.5.5 requests-oauthlib==1.3.0 responses==0.23.1 rsa==4.7.2 safetensors==0.4.1 schema==0.7.5 simplejson==3.19.1 smmap==5.0.0 soupsieve==2.4.1 sqlalchemy==2.0.12 sqlparse==0.4.4 tabulate==0.9.0 tensorboardx==2.2 termcolor==2.3.0 threadpoolctl==2.2.0 tifffile==2021.7.2 timm==0.9.12 tokenizers==0.12.1 tomli==2.0.1 torch-lucent==0.1.8 traitlets==5.9.0 transformers==4.21.3 typeguard==3.0.2 types-pyyaml==6.0.12.9 wcwidth==0.2.6 websocket-client==1.5.1 websockets==11.0.3 werkzeug==2.3.4)
conda activate sam_adapt
下载SAM checkpoint(https://dl.fbaipublicfiles.com/segment_anything/sam_vit_b_01ec64.pth),导入到./checkpoint/sam中。这里提供的是vit_base,也可以下载vit_large和vit_huge进行尝试。
复现皮肤图像黑色素瘤2D数据集分割任务(Melanoma Segmentation from Skin Images)
第一步:下载数据集
在ISIC官网(ISIC Challenge)下载ISIC 2016 part 1(或者说Task 1)的Training data和Test data以及Training ground truth和Test ground truth。数据集中几乎都是单目标分割,在训练集中只有一张图是有两个区域(ISIC_0011166),在测试集中也仅有一两张图是两个区域,并且都是以其中一个占比非常大,另一个区域很小,因此此处是单目标分割下的单个区域分割而非多个区域分割任务:
 研究需要用到的是Training_ground_truth.csv和Test_ground_truth.csv,在Github仓库里直接提供,但是这个仓库中的并不是Part1,而是Part3B,但打开csv可以发现里面的内容还是Part1的,并且内容是data和segmentation ground truth的对应关系。所以,我们需要将两个csv命名中的Part3B替换为Part1,同时将两个GroundTruth文件夹也要上传,这个在Github readme里没写,但是是需要传的。
研究需要用到的是Training_ground_truth.csv和Test_ground_truth.csv,在Github仓库里直接提供,但是这个仓库中的并不是Part1,而是Part3B,但打开csv可以发现里面的内容还是Part1的,并且内容是data和segmentation ground truth的对应关系。所以,我们需要将两个csv命名中的Part3B替换为Part1,同时将两个GroundTruth文件夹也要上传,这个在Github readme里没写,但是是需要传的。
上述目录的排布为:
./data/isic/ISBI2016_ISIC_Part1_Test_Data/...
./data/isic/ISBI2016_ISIC_Part1_Training_Data/...
./data/isic/ISBI2016_ISIC_Part1_Test_GroundTruth/...
./data/isic/ISBI2016_ISIC_Part1_Training_GroundTruth/...
./data/isic/ISBI2016_ISIC_Part1_Test_GroundTruth.csv
./data/isic/ISBI2016_ISIC_Part1_Training_GroundTruth.csv
第二步:运行指令
python train.py -net sam -mod sam_adpt -exp_name *msa_test_isic* -sam_ckpt ./checkpoint/sam/sam_vit_b_01ec64.pth -image_size 1024 -b 32 -dataset isic -data_path *../data*
此处需要将exp_name和data_path后面的参数替换成 自己的。此外,cfg.py中对各种参数arguments进行汇总,其中vis的默认值为None。然而,train.py会调用function.py,第340行为if ind % args.vis == 0,如果不对vis进行设置,这步就会报错TypeError: unsupported operand type(s) for %: 'int' and 'NoneType'。因此设置vis,即迭代多少轮进行一次可视化,如果不需要可视化,可以将vis的值往大了设置。此外,对于训练轮数epoch则是在./conf/global_settings.py中呈现。如果需要设置更大的epoch可在这里面修改。我看到Github的pull request中说建议训练100轮以上,因此我将epoch改为120轮。综上,最终我的指令为:
CUDA_VISIBLE_DEVICES=0 python train.py -net sam -mod sam_adpt -exp_name msa_test_isic -sam_ckpt ./checkpoint/sam/sam_vit_b_01ec64.pth -image_size 1024 -b 2 -dataset isic -data_path ./data/isic -vis 1
运行报错:
TypeError: Descriptors cannot be created directly.
If this call came from a _pb2.py file, your generated code is out of date and must be regenerated with protoc >= 3.19.0.
If you cannot immediately regenerate your protos, some other possible workarounds are:
1. Downgrade the protobuf package to 3.20.x or lower.
2. Set PROTOCOL_BUFFERS_PYTHON_IMPLEMENTATION=python (but this will use pure-Python parsing and will be much slower).
解决办法:将protobuf包降版本,pip install protobuf==3.20.3
还需pip install monai==1.1.0 seaborn==0.12.2 scikit-image
此步为单GPU训练,不支持多GPU训练。如果报错CUDA out of memory的话,可以适当调小image_size和b/batch_size。在单张NVIDIA Tesla T4 16GB显卡上,image_size=512 b=8正常运行;在单张NVIDIA 3090 24GB上,image_size=1024 b=2不报错。
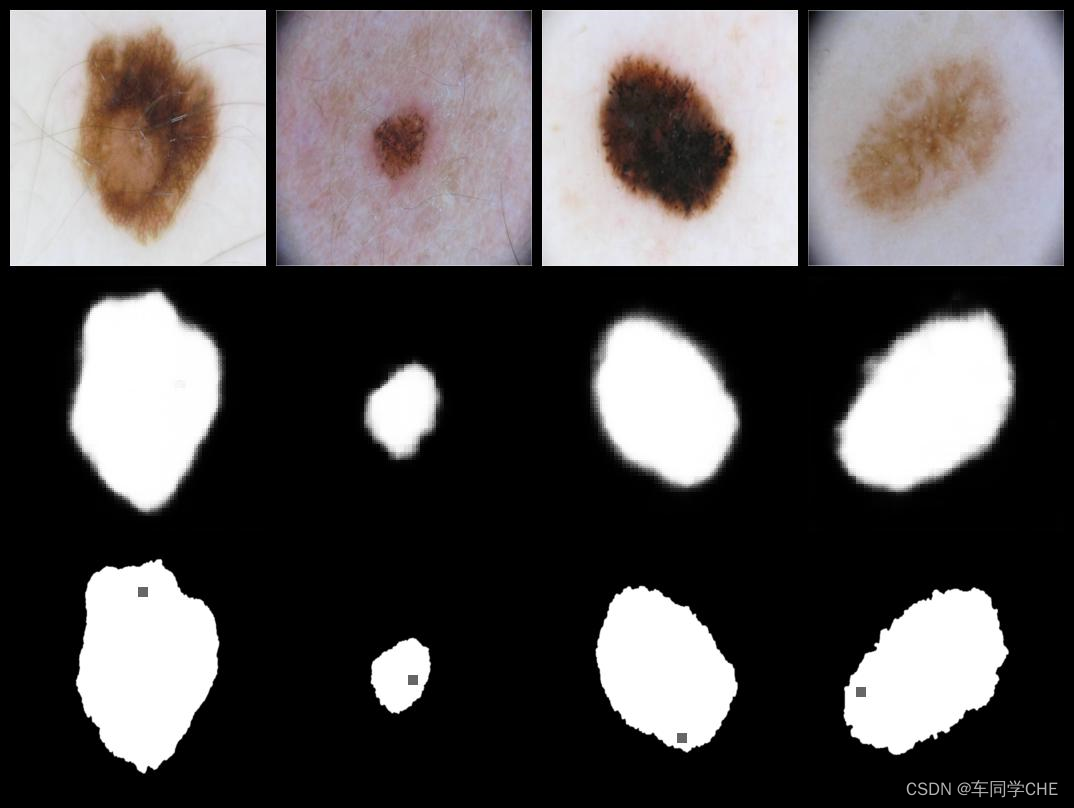
此步的结果生成在两个文件夹里:第一,训练过程曲线events.out.tfevents文件生成在./runs/sam/*文件夹里;第二,训练可视化结果在./logs/{exp_name}_{date}/Samples里(如下图,第一行是原图,第二行是MSA输出结果,第三行为Ground Truth,其中Ground Truth中的小灰点是随机生成的点prompt,该点是在positive区域中的,也就是要想让MSA输出结果,需要提供一个点prompt,分割多个目标区域则需要多个点),除了Samples以外还有存储最佳pth模型的Model文件夹以及Log文件夹(截至目前文件夹是空的)。
第3轮训练结果如下:
TestISIC_0000003+ISIC_0000012+ISIC_0000013+ISIC_0000014+ISIC_0000015+ISIC_0000020+ISIC_0000022+ISIC_0000023+epoch+3
复现眼底图像视盘2D数据集分割任务(REFUGE: Optic-disc Segmentation from Fundus Images)
第一步:下载数据集
下面简单介绍一下数据集,以及发现数据集中的csv文件如何对应的:
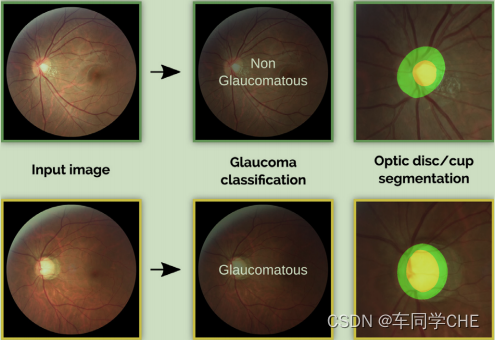
REFUGE2(Retinal Fundus Glaucoma Challenge Edition 2,第二届视网膜眼底青光眼竞赛),主要需要对视盘(Optic Disc, OD。大圆,下图绿色区域)和视杯(Optic Cup, OC。小圆,上图黄色区域)进行分割。视盘,全称视神经盘,也叫视神经乳头,为视网膜由黄斑向鼻侧约3mm处有一直径约1.5mm,境界清楚的淡红色圆盘状结构,如同一个盘子。眼底检查时,眼底所见视乳头有一个正常的生理性凹陷叫视杯(optic cup),如同一个杯子。杯子与盘子的直径之比就叫杯盘比,即C/D。视盘与视杯的示意图如下图所示:
数据集共计1200幅彩色眼底图像,其中120幅为青光眼患者,1080幅为非青光眼患者。图像大小height*width = 2056*2124。分辨率为96dpi。官方将数据集划分为训练集、验证集、测试集各400例,每个子集均包含20%的青光眼病例和80%的非青光眼病例。数据集图像由中国中山大学中山眼科中心提供。视盘和视杯的标注由该眼科中心的7名青光眼专家独立进行标注获得(对应cup和disc的7个png图像)。分割任务的参考标准由另一位高级青光眼专家将这7名专家标注整合为单个标注(对应cup和dics的1个bmp图像)。我认为,在实际进行模型训练时仅会用到原图和对应的disc/cup的bmp图像,而不会用到7个医生标注的png以及记录7个医生的视杯视盘标注细节的excel文件(如下图所示)。更多细节详见论文、官网(Program - Grand Challenge)和这个PDF(2nd Retinal Fundus Glaucoma Challenge 2020,第13页及之后详细描述了视杯视盘分割任务细节)

第二步:运行指令
CUDA_VISIBLE_DEVICES=0 python train.py -net sam -mod sam_adpt -exp_name REFUGE-MSAdapt -sam_ckpt ./checkpoint/sam/sam_vit_b_01ec64.pth -image_size 1024 -b 2 -dataset REFUGE -data_path ./data/REFUGE-MultiRater -vis 1
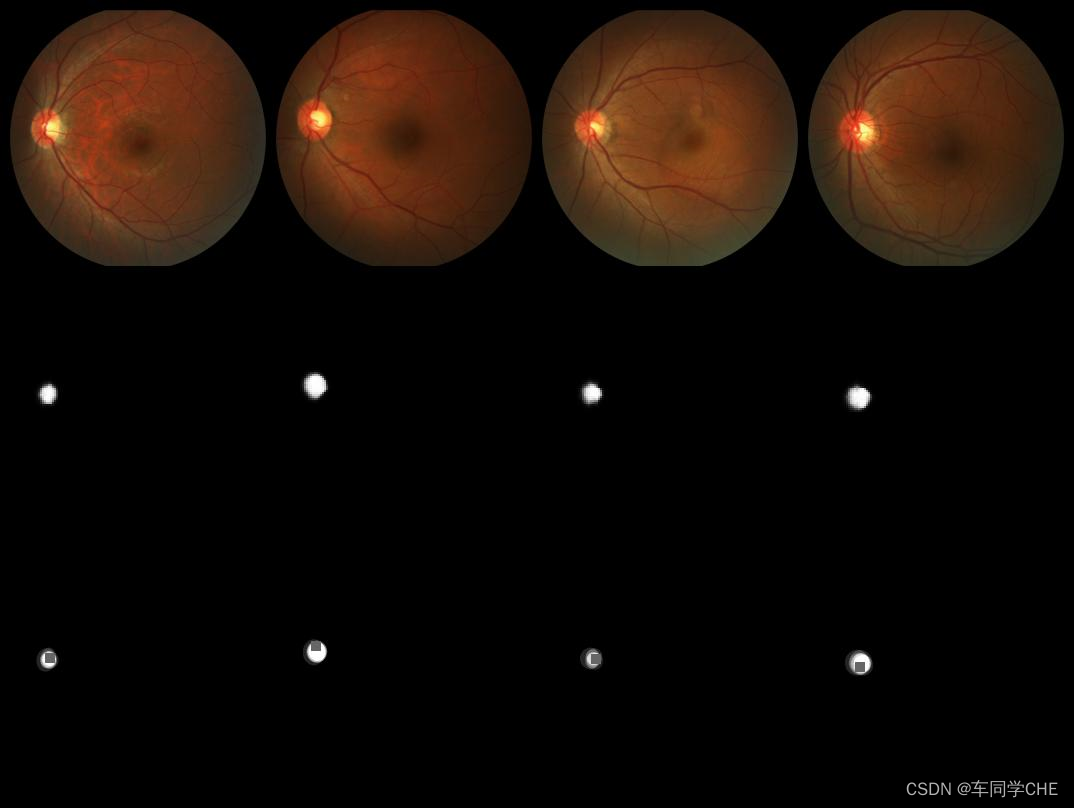
成功运行,第14轮训练结果如下:
Train1268+1072+1099+1085+1078+1093+0940+1151+epoch+14
复现腹部多器官3D数据集分割任务(Abdominal Multiple Organs Segmentation)
第一步:下载数据集
这里需要注意,在https://www.synapse.org/#!Synapse:syn3193805/wiki/217752下载BTCV数据集需要先在该页面进行注册,然后点击Wiki页面的How to Participate并点击Join,显示加入成功后刷新页面,即可在Files页面下载Abdomen/RawData.zip。如果没有注册,会显示这个页面:
如果成功注册并加入该比赛后,Files页面将变为下图:
温馨提醒:注意是下载约1.5GB的RawData.zip而不是将近40GB的Abdomen.zip!按照作者的说明分别解压、移至对应文件夹并重命名。此处建议将RawData.zip的Testing/img文件夹放在与imagesTr同级目录下,并命名为imagesTs,这是因为dataset_0.json中有涉及相关的路径。
经评论区提醒,目前作者对dataset_0.json的GoogleDrive分享链接设置了权限,需要申请。其全部内容如下:
{
"description": "btcv yucheng",
"labels": {
"0": "background",
"1": "spleen",
"2": "rkid",
"3": "lkid",
"4": "gall",
"5": "eso",
"6": "liver",
"7": "sto",
"8": "aorta",
"9": "IVC",
"10": "veins",
"11": "pancreas",
"12": "rad",
"13": "lad"
},
"licence": "yt",
"modality": {
"0": "CT"
},
"name": "btcv",
"numTest": 20,
"numTraining": 80,
"reference": "Vanderbilt University",
"release": "1.0 06/08/2015",
"tensorImageSize": "3D",
"test": [
"imagesTs/img0061.nii.gz",
"imagesTs/img0062.nii.gz",
"imagesTs/img0063.nii.gz",
"imagesTs/img0064.nii.gz",
"imagesTs/img0065.nii.gz",
"imagesTs/img0066.nii.gz",
"imagesTs/img0067.nii.gz",
"imagesTs/img0068.nii.gz",
"imagesTs/img0069.nii.gz",
"imagesTs/img0070.nii.gz",
"imagesTs/img0071.nii.gz",
"imagesTs/img0072.nii.gz",
"imagesTs/img0073.nii.gz",
"imagesTs/img0074.nii.gz",
"imagesTs/img0075.nii.gz",
"imagesTs/img0076.nii.gz",
"imagesTs/img0077.nii.gz",
"imagesTs/img0078.nii.gz",
"imagesTs/img0079.nii.gz",
"imagesTs/img0080.nii.gz"
],
"training": [
{
"image": "imagesTr/img0001.nii.gz",
"label": "labelsTr/label0001.nii.gz"
},
{
"image": "imagesTr/img0002.nii.gz",
"label": "labelsTr/label0002.nii.gz"
},
{
"image": "imagesTr/img0003.nii.gz",
"label": "labelsTr/label0003.nii.gz"
},
{
"image": "imagesTr/img0004.nii.gz",
"label": "labelsTr/label0004.nii.gz"
},
{
"image": "imagesTr/img0005.nii.gz",
"label": "labelsTr/label0005.nii.gz"
},
{
"image": "imagesTr/img0006.nii.gz",
"label": "labelsTr/label0006.nii.gz"
},
{
"image": "imagesTr/img0007.nii.gz",
"label": "labelsTr/label0007.nii.gz"
},
{
"image": "imagesTr/img0008.nii.gz",
"label": "labelsTr/label0008.nii.gz"
},
{
"image": "imagesTr/img0009.nii.gz",
"label": "labelsTr/label0009.nii.gz"
},
{
"image": "imagesTr/img0010.nii.gz",
"label": "labelsTr/label0010.nii.gz"
},
{
"image": "imagesTr/img0021.nii.gz",
"label": "labelsTr/label0021.nii.gz"
},
{
"image": "imagesTr/img0022.nii.gz",
"label": "labelsTr/label0022.nii.gz"
},
{
"image": "imagesTr/img0023.nii.gz",
"label": "labelsTr/label0023.nii.gz"
},
{
"image": "imagesTr/img0024.nii.gz",
"label": "labelsTr/label0024.nii.gz"
},
{
"image": "imagesTr/img0025.nii.gz",
"label": "labelsTr/label0025.nii.gz"
},
{
"image": "imagesTr/img0026.nii.gz",
"label": "labelsTr/label0026.nii.gz"
},
{
"image": "imagesTr/img0027.nii.gz",
"label": "labelsTr/label0027.nii.gz"
},
{
"image": "imagesTr/img0028.nii.gz",
"label": "labelsTr/label0028.nii.gz"
},
{
"image": "imagesTr/img0029.nii.gz",
"label": "labelsTr/label0029.nii.gz"
},
{
"image": "imagesTr/img0030.nii.gz",
"label": "labelsTr/label0030.nii.gz"
},
{
"image": "imagesTr/img0031.nii.gz",
"label": "labelsTr/label0031.nii.gz"
},
{
"image": "imagesTr/img0032.nii.gz",
"label": "labelsTr/label0032.nii.gz"
},
{
"image": "imagesTr/img0033.nii.gz",
"label": "labelsTr/label0033.nii.gz"
},
{
"image": "imagesTr/img0034.nii.gz",
"label": "labelsTr/label0034.nii.gz"
}
],
"validation": [
{
"image": "imagesTr/img0035.nii.gz",
"label": "labelsTr/label0035.nii.gz"
},
{
"image": "imagesTr/img0036.nii.gz",
"label": "labelsTr/label0036.nii.gz"
},
{
"image": "imagesTr/img0037.nii.gz",
"label": "labelsTr/label0037.nii.gz"
},
{
"image": "imagesTr/img0038.nii.gz",
"label": "labelsTr/label0038.nii.gz"
},
{
"image": "imagesTr/img0039.nii.gz",
"label": "labelsTr/label0039.nii.gz"
},
{
"image": "imagesTr/img0040.nii.gz",
"label": "labelsTr/label0040.nii.gz"
}
]
}
第二步:运行指令
CUDA_VISIBLE_DEVICES=0 python train.py -net sam -mod sam_adpt -exp_name msa-3d-sam-btcv -sam_ckpt ./checkpoint/sam/sam_vit_b_01ec64.pth -image_size 1024 -b 8 -dataset decathlon -thd True -chunk 96 -data_path ../data -num_sample 4 -vis 1
成功运行,相当一部分结果不太行,可能是我在调参时对于各个参数的意义的理解还不到位,不太会调。挑了一张能看的结果展示如下:
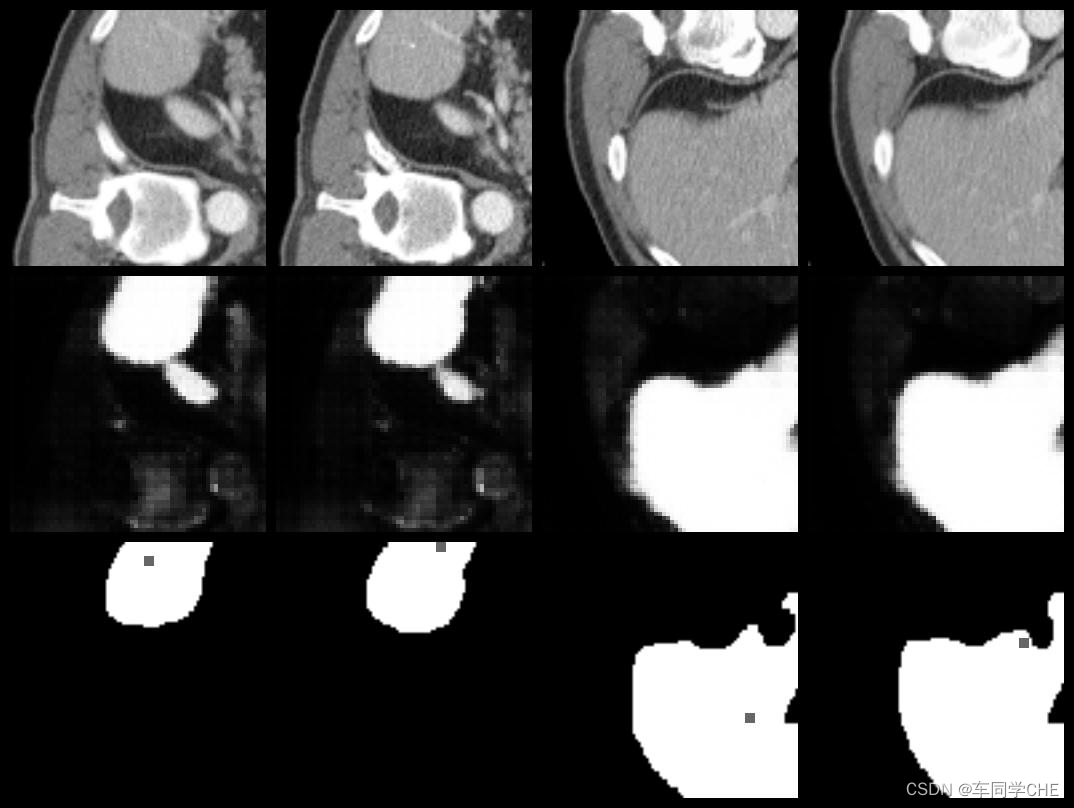
执行时遇到两个报错,下面对其进行说明:
第一,界面输出pixdim[0] (qfac) should be 1 (default) or -1; setting qfac to 1提示。解决办法:参考(https://blog.youkuaiyun.com/weixin_42699538/article/details/129250195),在train.py开头添加:import logging; logging.disable(logging.WARNING)。忽略掉该warning即可。
第二,在train时不报错,但在每若干轮train后的valid环节会报错(虽然会报error,但是代码没有终止,还是能继续执行直至结束):
Exception in thread Thread-165 (enqueue_values):
Traceback (most recent call last):
....
File ".../lib/python3.10/site-packages/monai/transforms/utility/array.py", line 231, in __call__
raise ValueError(msg)
ValueError: Metadata not available and channel_dim=None, EnsureChannelFirst is not in use.
The above exception was the direct cause of the following exception:
Traceback (most recent call last):
...
raise RuntimeError(f"applying transform {transform}") from e
RuntimeError: applying transform <monai.transforms.io.dictionary.LoadImaged object at 0x7f91141229b0>
The above exception was the direct cause of the following exception:
...
Traceback (most recent call last):
...
raise RuntimeError(f"applying transform {transform}") from e
RuntimeError: applying transform <monai.transforms.compose.Compose object at 0x7f9114121e40>
解决办法:未知。
使用自己的数据集进行测试
根据Github的readme的说明,对于运行自己的数据集,需要在./dataset.py中写一个数据集类,调用时返回一个字典,包括:
{
'image': A tensor saving images with size [C,H,W] for 2D image, size [C, H, W, D] for 3D data. D is the depth of 3D volume, C is the channel of a scan/frame, which is commonly 1 for CT, MRI, US data. If processing, say like a colorful surgical video, D could the number of time frames, and C will be 3 for a RGB frame.
'label': The target masks. Same size with the images except the resolutions (H and W).
'p_label': The prompt label to decide positive/negative prompt. To simplify, you can always set 1 if don't need the negative prompt function.
'pt': The prompt. Should be the same as that in SAM, e.g., a click prompt should be [x of click, y of click], one click for each scan/frame if using 3d data.
'image_meta_dict': Optional. if you want save/visulize the result, you should put the name of the image in it with the key ['filename_or_obj'].
'xxxx': ... (others as you want)
}
之所以需要写一个./dataset.py类,是因为在train.py的第35行from dataset import *,将dataset.py中的类导入到train.py里面,并在第95行开始的if语句里进行调用。查看dataset.py会发现,第一个项目用到了csv进行配对,而第二个项目没有用到csv(我也觉得第二个项目的csv乱作一团,并不匹配,csv表头的unnamed0.1.1是pandas的格式错误,并非真正的标题)。参考作者给出的dataset.py类可以比较容易的写自己的类。
补充说明
1、对于论文Figure 3的补充说明
对于作者在文献中的Figure 3,它并没有进行详细的说明,其中一点是可能被大家所忽略而造成理解上的偏差。先将图粘贴如下:
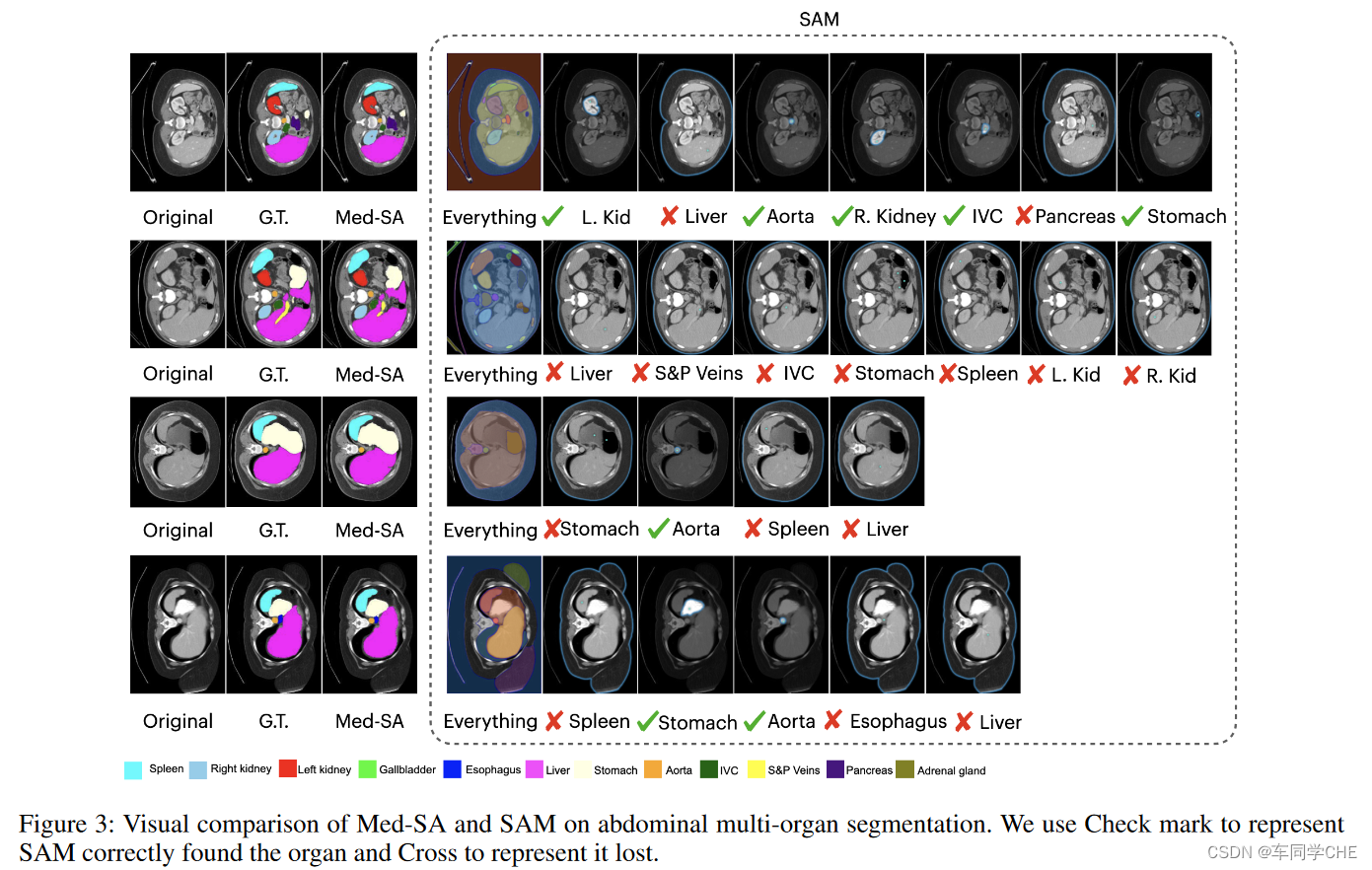
这个图是作者将所提的Med-SA和原先的SAM进行对比。一个奇怪的点是,例如第二行的例子,明明SAM的Everything里已经有正确的分割Spleen了,但是在右边的Spleen那里还打了个叉。这里可能被大家忽略的一点是,作者对于SAM的操作分为两块,一个是Everything的分割结果,另一个是使用单点标记的分割结果。将Figure 3第二行的例子中的SAM进行放大,如下图所示:
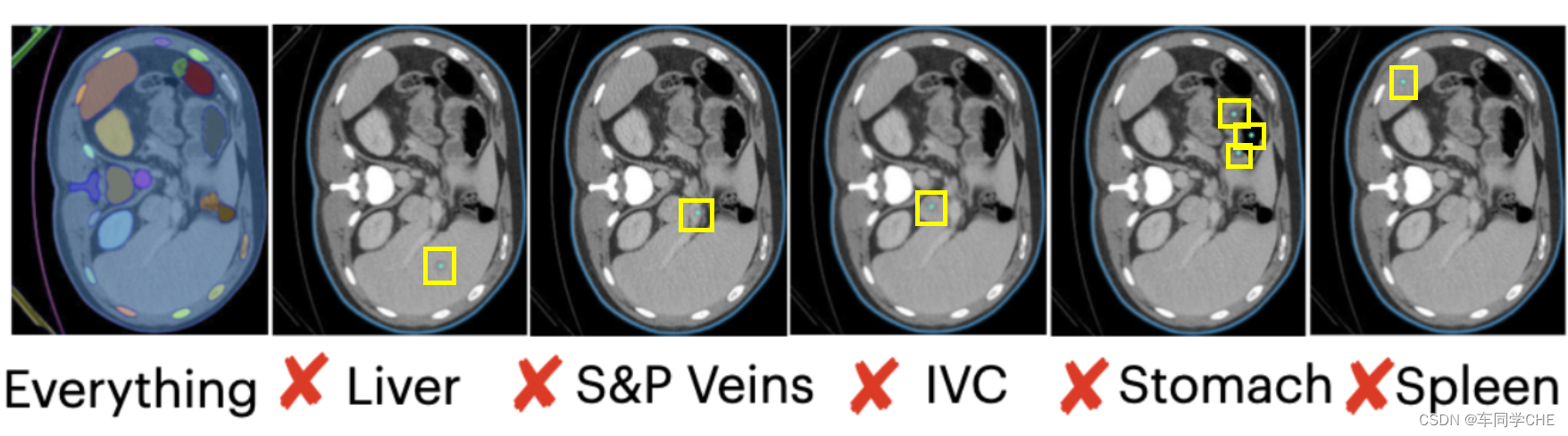
请注意,在上图中,作者在评判时,分别在对应位置点了一个浅蓝色的点并让SAM进行分割(我在此用黄色框进行突出说明),而SAM对于该点的prompt输入的输出结果都是最外侧的浅蓝色轮廓,所以作者认为都没有成功分割结果。虽然在上图的Everything中,Spleen区域被成功分割,但是对于单点prompt输入的Spleen分割没有能够输出Spleen的结果,所以没有成功分割。作者试图传递的想法应该是,并不指望SAM自动分割出所需区域,而是希望在给定一个点或者后文提到的框的prompt的情况下进行准确的分割。此外,对于SAM的Everything中的颜色与Figure 3的颜色或Ground Truth的颜色并不匹配,我猜想的原因是作者直接将SAM的Everything的分割结果进行了截图,这里的颜色是SAM自己随机赋予的,作者并没有将分割结果中的颜色与左边Ground Truth的颜色进行匹配。
2、对于一次点多个点能否识别多个目标的说明——不要这么干!
在SAM的官网有提供demo(Segment Anything | Meta AI)
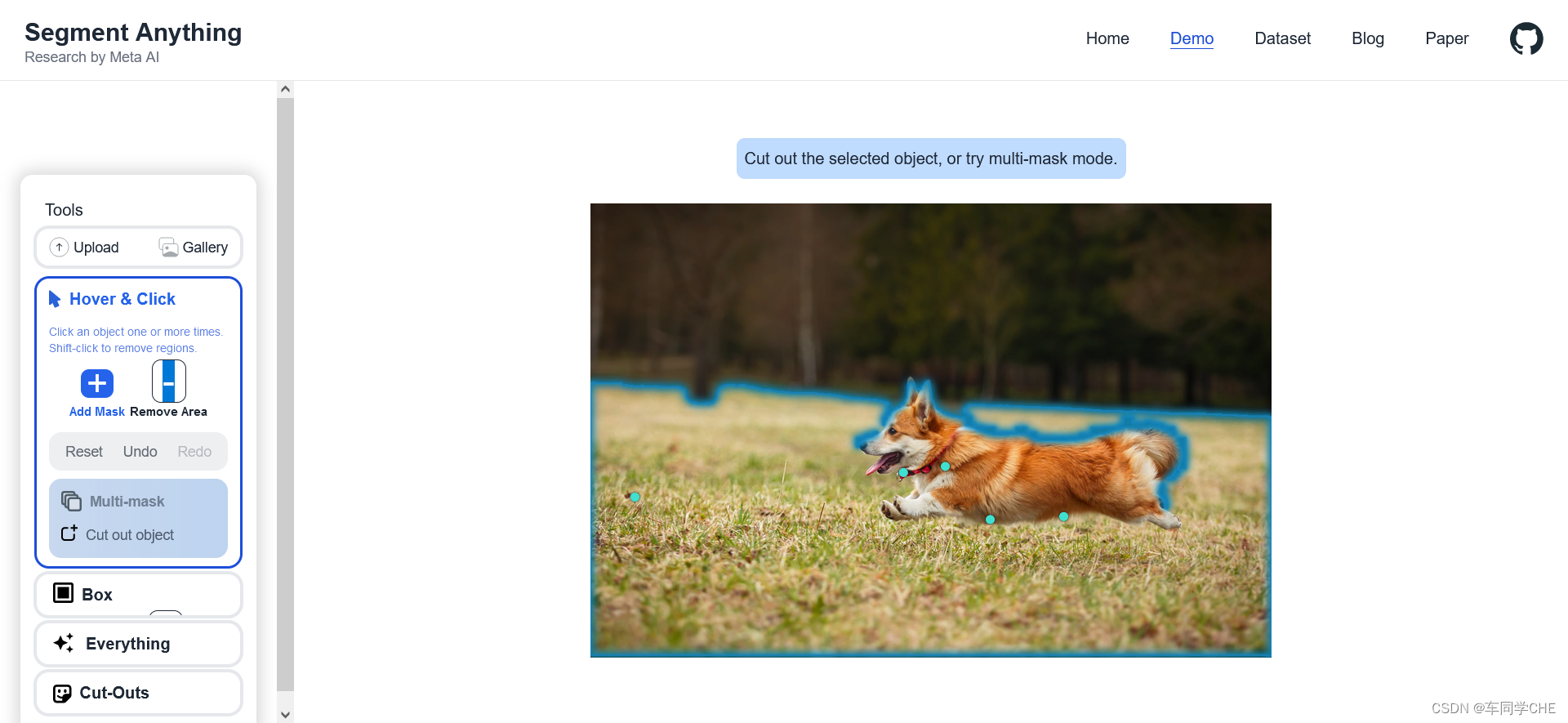
在上图中,我成功的分割出了前景区域,在分割过程中,我发现点击给一个点prompt的分割结果可能会出现多个区域,但是会更倾向于连通,也就是更倾向于分割出一个类别。

在上图中,如果我直接分出中间的红色椅子,是没有问题的,但如果我在点了左边两个点的基础上,在右边点了第三个点,则会出现问题——我试图只想分割出两个红色的椅子,但是它把中间的黄色椅子也给分割出来了,并且远出的木头椅子上也被分割出了一小块。可见SAM的点prompt功能对于单目标的分割是最好的,目标越单一效果越好。作者也是看重了这个特性。特别的,倘若希望识别多个区域,比如我希望将上图中的每一个椅子分别识别出来,可以分多次分别给每个椅子点个点,成功分割出一个椅子之后,再清空点prompt开始分割另一个椅子。在本研究的inference的时候,由于分割的是同一目标,所以最理想的情况下就是点一个点就能分出来,如果一个点差强人意的话,点三个点最好也能分割出来。

3、分割一个CT图像需要训练多少个模型?——应该只需要1个!
前文说到,在一张图上分割一个目标可以点多个点。但对于识别多个组织成分则需要重新加载prompt进行分割,而不能将每个目标上都点个点,并希望模型能够分别将其分割出来。但对于多个目标的识别,比如CT图像,对于不同组织成分只需要训练一个模型即可,不需要每个组织分别训练一个模型。





















 7126
7126










