快速体验
- 打开 InsCode(快马)平台 https://www.inscode.net
- 输入框内输入如下内容:
根据最新医学影像分割论文(UNet++架构),开发一个端到端的COVID-19肺部CT影像分析系统。要求:1) 使用PyTorch实现UNet++模型 2) 集成DICOM图像读取接口 3) 实现基于Grad-CAM的可视化解释 4) 提供Flask API接口供医生上传影像获取分析结果。训练数据使用公开的COVID-19 CT数据集。 - 点击'项目生成'按钮,等待项目生成完整后预览效果
最近在做一个医疗影像分析的项目,目标是开发一个能够自动分析COVID-19肺部CT影像的系统。这个系统的核心是基于UNet++架构的深度学习模型,能够自动分割CT影像中的病灶区域,并提供可视化的解释。整个过程从论文阅读到最终产品部署,经历了不少挑战和收获,今天就来分享一下我的实战经验。
- 项目背景与需求分析
医疗影像分析在COVID-19诊断中具有重要意义。医生通过CT影像可以快速判断患者的肺部感染情况,但手动分析耗时且容易遗漏细节。我们的目标是开发一个自动化工具,帮助医生快速定位病灶并提供辅助诊断建议。系统需要满足以下功能: - 支持DICOM格式的CT影像读取
- 使用UNet++模型进行病灶分割
- 提供基于Grad-CAM的可视化解释
-
通过API接口供医生上传影像并获取分析结果
-
技术选型与模型实现
选择UNet++架构是因为它在医学影像分割任务中表现优异,尤其是对细小结构的捕捉能力。实现过程中,我们使用PyTorch框架搭建模型,主要包括以下几个步骤: - 数据预处理:对公开的COVID-19 CT数据集进行标准化和增强处理
- 模型训练:使用交叉熵损失函数和Adam优化器进行训练
-
可视化解释:集成Grad-CAM技术,生成模型关注区域的热力图
-
DICOM图像读取与处理
DICOM是医学影像的通用格式,但直接处理起来比较复杂。我们使用pydicom库读取DICOM文件,并将其转换为适合模型输入的张量格式。需要注意的是,CT影像的像素值(Hounsfield单位)需要经过特定的窗宽窗位调整,以突出肺部组织的对比度。 -
API接口与部署
为了让医生能够方便地使用这个系统,我们开发了一个基于Flask的API接口。医生可以通过网页上传CT影像,系统会返回分割结果和可视化解释。部署时,我们使用了InsCode(快马)平台的一键部署功能,省去了服务器配置和环境搭建的麻烦。整个过程非常流畅,从代码上传到服务上线只用了不到10分钟。
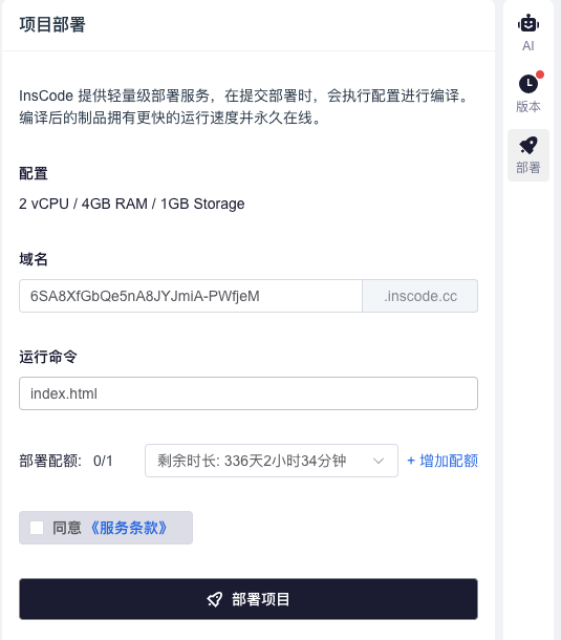
- 实际应用与效果
在测试阶段,我们的系统表现良好,能够准确分割出COVID-19的典型病灶(如磨玻璃影)。Grad-CAM的可视化解释也帮助医生理解模型的决策依据,增强了系统的可信度。未来,我们计划进一步优化模型性能,并扩展支持更多的疾病类型。
总结一下,从论文到产品的过程中,选择合适的工具和平台非常重要。InsCode(快马)平台的便捷部署功能让我能够快速将研究成果转化为实际应用,省去了很多不必要的麻烦。如果你也在做类似的AI项目,不妨试试这个平台,相信会给你带来不少便利。
快速体验
- 打开 InsCode(快马)平台 https://www.inscode.net
- 输入框内输入如下内容:
根据最新医学影像分割论文(UNet++架构),开发一个端到端的COVID-19肺部CT影像分析系统。要求:1) 使用PyTorch实现UNet++模型 2) 集成DICOM图像读取接口 3) 实现基于Grad-CAM的可视化解释 4) 提供Flask API接口供医生上传影像获取分析结果。训练数据使用公开的COVID-19 CT数据集。 - 点击'项目生成'按钮,等待项目生成完整后预览效果
创作声明:本文部分内容由AI辅助生成(AIGC),仅供参考





















 552
552

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










