实现功能:
输入:Uniprot中的accession number
输出:与输入蛋白相关的蛋白交互对(左列是原列表中的蛋白,右侧是查询到的partner)
直接使用代码
https://github.com/JulseJiang/Code_for_blog/blob/main/Python_ExtractPartnerFromIntAct/exTMPnonTMPpiar20201201.py
或者看看处理过程:
数据下载
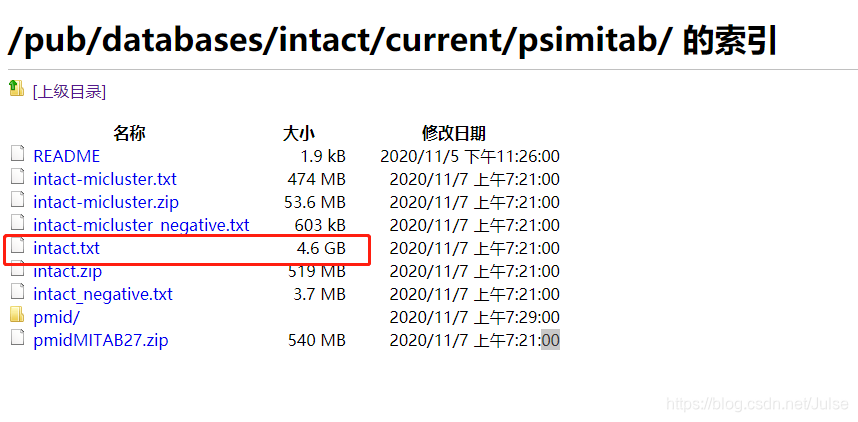
数据集下载链接:这里下载mitab格式,方便解析
ftp://ftp.ebi.ac.uk/pub/databases/intact/current/psimitab/
下载intact.txt或者intact.zip 自行解压都行
提取IntAct中的全体蛋白对
实现效果
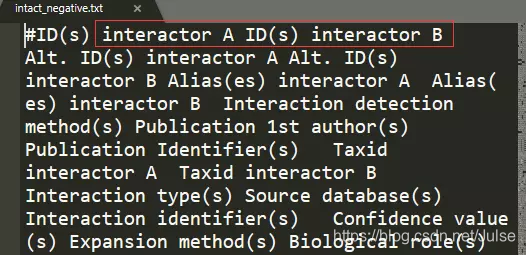
文件中部分蛋白是Uniprot中的,部分蛋白是EBI中的,目前只考虑Uniprot中的
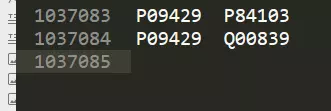
提取到103w对来自uniprot的蛋白,去重之后剩余673577对
20201201 下载的数据,也就是2020 年11月7日发布的数据,里面有673577对的蛋白来自uniprot标注
blog/blob/main/Python_ExtractPartnerFromIntAct/pair_norepeat.txt
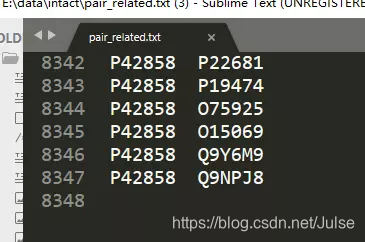
输入某个蛋白质accession 列表,查找到8k条相关的蛋白对
相关代码
- 提取intact.txt中用Uniprot ID表示的蛋白对
def extractPPIFromIntAct(finPair,foutPair):
# finPair = r'E:\data\intact\intact.txt'
# foutPair = r'E:\data\intact\pair.txt'
with open(finPair, 'r', encoding='UTF-8') as fi, open(foutPair, 'w') as fo:
line = fi.readline()
while (line):
line =




 该博客介绍使用Python提取IntAct中蛋白交互对的方法。先从指定链接下载mitab格式数据集,再提取全体蛋白对,考虑Uniprot中的蛋白,去重后得到一定数量蛋白对。输入蛋白质accession列表可查找相关蛋白对,还给出了相关代码及需考虑的accession过期问题。
该博客介绍使用Python提取IntAct中蛋白交互对的方法。先从指定链接下载mitab格式数据集,再提取全体蛋白对,考虑Uniprot中的蛋白,去重后得到一定数量蛋白对。输入蛋白质accession列表可查找相关蛋白对,还给出了相关代码及需考虑的accession过期问题。
 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章

















 1171
1171

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








