在蛋白质科学领域,有两大“高峰”始终令人望而生畏:
● 其一为“预测”——给定氨基酸序列,预测其最终折叠形成的结构;
● 其二为“设计”——从目标功能出发,反向推导并构建出全新的蛋白质结构与序列。
前者的突破得益于AlphaFold 2,后者的关键转折点则是RFdiffusion的问世。短短三年内,从2023年的v1版本,到2025年的v2与v3版本,RFdiffusion系列持续刷新着人们的认知:
● v1版本证明了“AI可从零生成全新蛋白质骨架”,并在大规模任务中验证了这条全新设计路径的可行性;
● v2版本攻克了“酶活性位点设计”的难题,将这一领域的“圣杯”目标进一步推向现实;
● v3版本则向“全原子-多模态分子”方向迈进,目前已能生成侧链原子的3D坐标,但仍需借助序列设计工具(如LigandMPNN);
● 当然,我们更期待其下一版本能在不依赖额外序列设计工具的情况下,独立完成蛋白质设计。
我们的 SciMiner 平台已部署 RFdiffusion v1 与 v2,并将在 v3 开源后第一时间跟进,让更多科研人员能够直接体验这场“AI 造蛋白”的革命。
初登舞台:v1 打开生成式蛋白设计的大门
2021-2022 年,AlphaFold2 震撼发布,让蛋白结构预测的问题在很大程度上得到解决。可设计新蛋白,仍是一道几乎无法跨越的难题。
传统的酶或结合蛋白设计,要么依赖物理化学能量函数的暴力搜索,要么从已有模板出发“修修补补”。前者计算量大到令人绝望,后者缺乏真正创新。
Baker 实验室在 2023 年推出的 RFdiffusion v1,开辟了另一条道路:
● 扩散模型可以从随机噪声开始,逐步去噪,最终生成合理的蛋白骨架;
● 用户可以加条件,比如要求对称性、加入特定 motif、或设计结合某分子;
● 模型则会在这些约束下“生长”出满足条件的蛋白结构。
这意味着,科研人员不再需要穷举式搜索,而是能通过“生成”来探索新的结构空间。
案例与冲击
在 v1 的论文和实验中,研究人员展示了多个惊艳结果:
● de novo 折叠:完全没有模板的单链蛋白;
● 对称装配体:环状多聚体甚至笼状壳层;
● 结合界面:能与病毒蛋白或小分子贴合的蛋白骨架;
● motif scaffold:在新骨架中精准嵌入功能性基团。
更令人震撼的是,一些设计经实验验证与目标分子严丝合缝地结合,结构几乎与设计模型完全一致。
难怪当时社区流传一句话:
“以前要筛上万种突变才能碰到一个勉强可用的,现在 RFdiffusion 一次就能给你几十个靠谱方案。”
当然,v1 也有局限:它主要关注骨架层面,在更精细的功能上仍显得力不从心。
突破瓶颈:v2 让“设计催化酶”成为现实
酶的催化能力常被视为蛋白工程的“终极难题”。要让一组残基精确摆放在口袋中以催化反应,需要满足极为严格的空间几何约束。传统上,研究人员必须预先挑选残基并定义其编号和侧链取向,然后再嵌入骨架。这个过程费时费力,且效率极低。即便如此,设计出的酶通常活性也很有限,需要海量筛选。
v1 的瓶颈
在 RFdiffusion v1 中,要实现活性位点设计,研究者必须:
1. 明确指定哪些残基构成活性位点(带索引的 motif);
2. 提供侧链取向,告诉模型这些原子怎么摆放。
这意味着设计者在输入阶段已经预先固定了部分结果,费力不说,还削弱了模型自主探索的空间。
v2 的革命性突破
024-2025 年,Baker 团队推出 RFdiffusion v2,正是为解决这一瓶颈而来。
它最大的创新是:
● 无索引活性位点支架(unindexed motif scaffolding)
研究者只需提供活性位点原子的空间几何关系,模型会在生成过程中自动决定:这些原子属于哪些残基、应该放在蛋白序列的哪个位置、侧链该怎么构象。这相当于告诉模型“我要一个满足几何关系的活性中心”,而不是“请把残基 35、72、108 放在这里”。
为实现这一点,v2 引入了两项关键技术:
● 双扩散轨道:一条扩散主链骨架,另一条扩散活性位点原子,两者逐步融合,确保几何约束得到精确实现。
● Flow matching training:一种更高效的训练方法,能让模型更快、更稳定地学习如何把噪声转化为目标几何。
成果与影响
在包含 41 个挑战性酶任务的基准测试中:
● RFdiffusion v2 解出了全部 41 个,而此前最好的方法只能解决其中 16 个。
● 在实验中,研究团队仅筛选了不到 100 个设计,就获得了 5 个具备实际活性的全新酶。
要知道,传统流程常常需要筛选上万候选。效率提升可谓颠覆性。
更令人兴奋的是,在一个锌离子水解酶的案例中,v2 设计出的酶活性甚至超过了人类多年工程改造的版本。
这意味着,从化学反应出发设计酶的目标,第一次在 AI 辅助下得以实现。
全面升级:v3 走向全原子、多分子
2025 年 9 月,Baker 团队在预印本中推出了 RFdiffusion v3(简称 RFD3),提出了一个全新的思路:
● 全原子扩散:模型不再只生成骨架,而是直接在直角坐标系下同时建模主链、侧链和非蛋白原子,统一扩散出完整复合物的3D坐标。
● 多模态分子体系:支持小分子配体、DNA等异源分子,能够在一个框架内生成蛋白-配体、蛋白-核酸、乃至多链组装的复合结构的3D坐标。
● 更灵活的条件约束:用户可以在输入中显式指定氢键网络、残基环境、溶剂可及性或配体几何,使模型“长”出与目标高度契合的结合界面。
● 简化而高效的架构:研发团队舍弃了前代的 SE(3) 等变框架,转而采用类似 “AlphaFold3” 的直角坐标原子级建模,让网络通过大规模数据直接学习物理规律。这一改动减少了人工归纳偏置,换来了更大的扩展性与更快的迭代周期。
在论文和开发者的技术分享中,RFD3 展示了多个令人兴奋的案例:
● 设计全新的蛋白–小分子复合物;
● 在 DNA 上“长”出能识别特定序列的蛋白结合域;
● 为酶设计直接考虑底物或辅酶的存在,实现真正意义上的“反应导向”。
虽然 RFD3 目前仍处于学术探索阶段,尚未开源,但初步结果已经显示它让一系列此前困难重重的设计类别成为可能。社区普遍认为,随着 v3 的发布,蛋白设计已从“骨架—活性位点”迈向“全原子—多模态分子”的新纪元。一旦开放,SciMiner 平台将第一时间完成部署,继续为科研人员提供最前沿的工具。
SciMiner:让前沿工具真正成为“可用技术”
RFdiffusion 的突破固然令人兴奋,但对于大多数科研团队而言,模型复杂、依赖繁重、环境脆弱,使其往往停留在“看得到却用不上”的状态。SciMiner 的使命,就是打破这道天然的技术壁垒。我们将 RFdiffusion v1 与 v2 以工程化方式上线,使其变成任何科研人员都能直接调用的工具:无需部署、无需配置、无需高性能集群,一台电脑、一个浏览器就能完成设计尝试。
科研人员可以把更多时间投入到科学问题本身,而不是耗费精力在环境、依赖和脚本上。RFdiffusion 在 SciMiner 上变成了一种“开箱即用”的能力,而不是一座难以攀登的技术高峰。
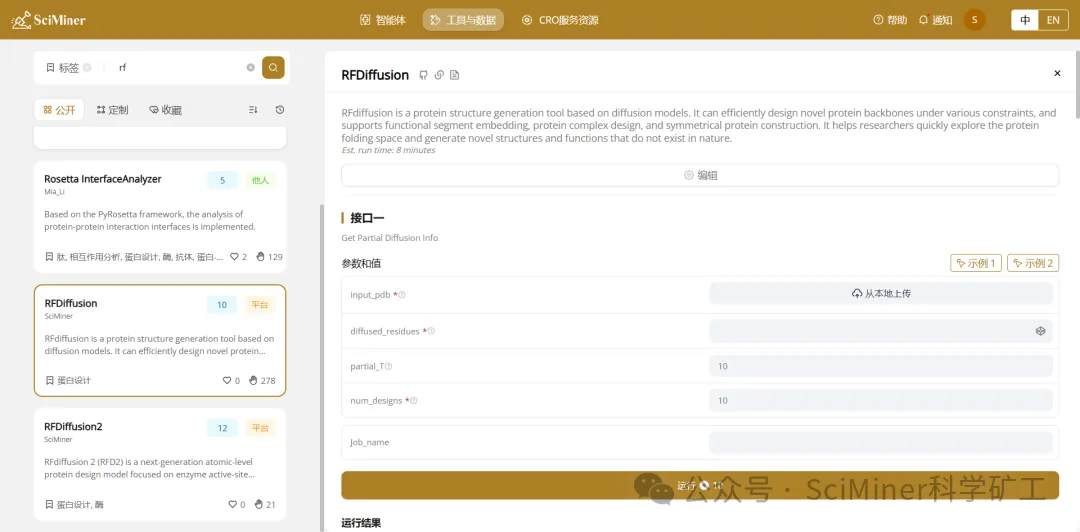
RFDiffusion输入界面图
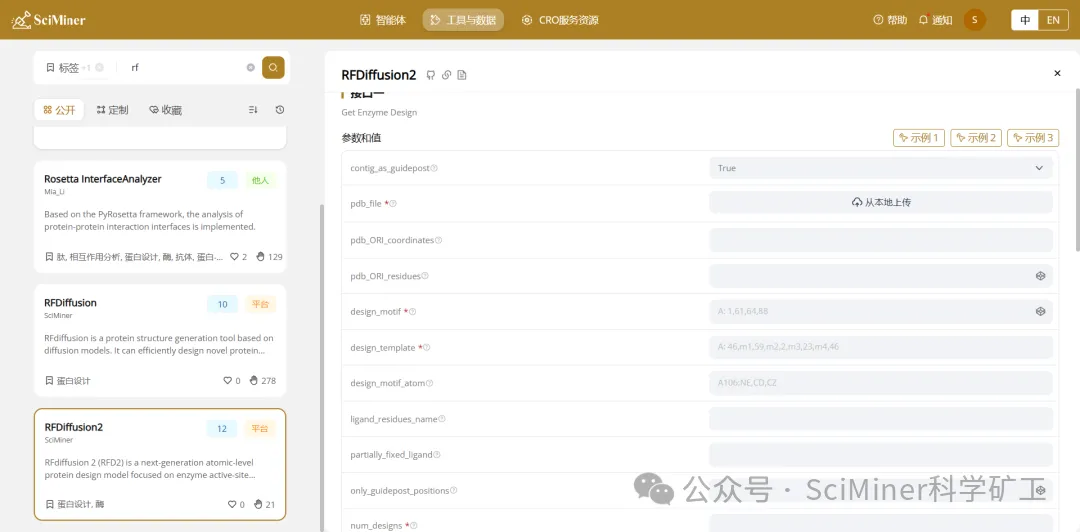
RFDiffusion2输入界面图
结语
RFdiffusion 系列见证了生成式 AI 在蛋白设计上的飞速进化,通过 SciMiner (http://sciminer.tech/),这些前沿工具已经从“实验室里的尖端模型”,变为科研人员日常可用的设计平台。我们相信,在不远的未来,“AI 按需造蛋白”将成为实验室里的日常操作,推动生命科学迈入一个真正的创造时代。






















 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








