阅读中产生的问题:
(1).mhd 文件和.raw 文件如何组成CT三维数组?形成过程是如何的? (来源:10.4知识点和10.5图左上部分)
解答:参考博客医学图像处理——数据预处理(.mhd+raw格式图像读取和显示)_mhd格式文件-优快云博客
(还是对逻辑形成过程理解不够清楚)
(2)从图10.5中候选结节位置为三维,到图10.7中数组坐标为二维,对结节的判断有影响吗?
解答:参考博客 医学影像中的坐标系_ct坐标系-优快云博客
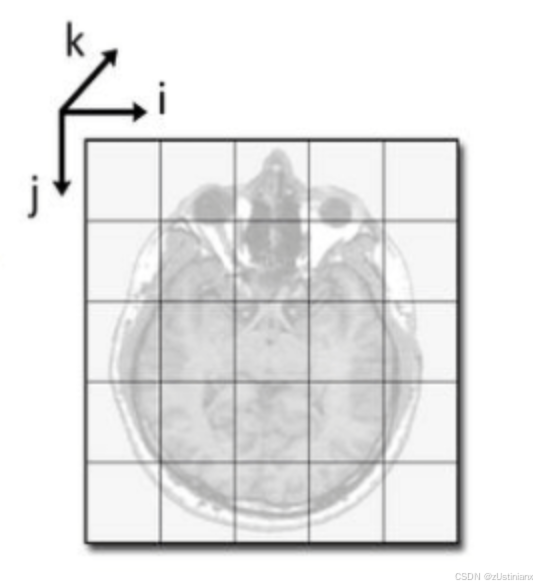
查找资料后,显示转化为病人坐标系后,仍是三维,与图10.7不符。
(3)从数组坐标系到病人坐标系:执行逆操作。逆操作的具体过程?
解答: (对10.4标红部分从病人坐标系到数组坐标系的逆向理解)
从数组坐标系到病人坐标系的逆操作:
1.使用方向矩阵的逆矩阵对数组坐标进行变换,
2.通过除以体素大小来调整尺度,
3.加上原点偏移量来完成整个转换过程。
ps:数学运算需要考虑到三维空间中每个维度(通常是x, y, z)上的不同值。
10.1 原始 CT 数据文件
CT 数据由两个文件组成:.mhd 文件包含元数据头信息,.raw 文件包含三维数组的原始数据。
实现一个 Ct 类,将这两个文件加载为三维数组,并进行矩阵转换,将病人坐标系转换为数组所需的索引、行和列坐标(用 _irc 表示)。
10.2 解析 LUNA 的标注数据
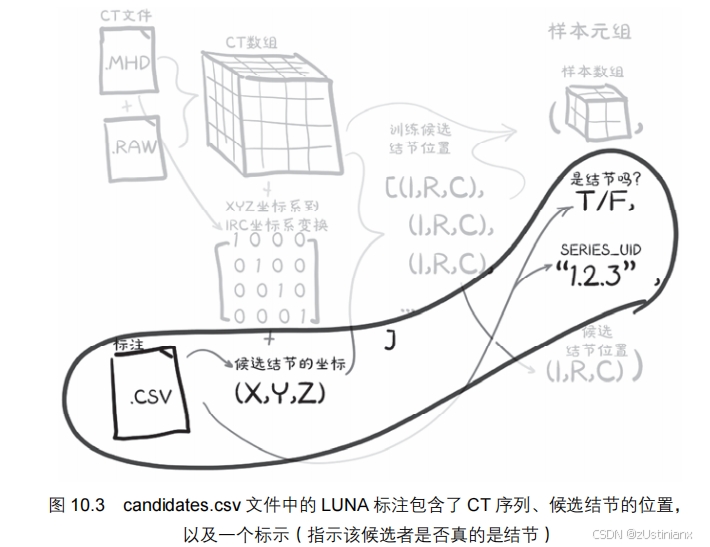
-
标注文件
LUNA 数据集提供了两个 CSV 文件:candidates.csv和annotations.csv。-
candidates.csv包含所有可能的结节候选信息,包括 CT 序列 UID、坐标和是否为结节的标记。 -
annotations.csv包含已标记为实际结节的信息,包括结节的直径。
-
-
数据处理
-
加载 CSV 文件:使用 Python 的
csv模块加载文件内容。 -
合并标注和候选数据:通过
getCandidateInfoList()函数,将两个文件中的信息合并为一个统一的列表。 -
处理坐标不匹配问题:两个文件中的坐标可能不完全对齐,通过一定的算法判断它们是否为同一个结节。
-
10.3 加载单个 CT 扫描
-
数据加载
使用 SimpleITK 库加载.mhd和.raw文件,将其转换为 NumPy 数组。-
文件路径通过
glob模块查找,确保文件存在。 -
使用
sitk.ReadImage()读取图像,并通过sitk.GetArrayFromImage()转换为 NumPy 数组。
-
-
数据清理
CT 数据以亨氏单位(HU)表示,范围为 -1000 到 +1000。-
将数据值限制在 [-1000, 1000] 范围内,以去除噪声和无关信息。
-
通过
clip()方法清理数据。
-
10.4 使用病人坐标系定位结节
-
病人坐标系与数组坐标系
病人坐标系以毫米为单位,而数组坐标系以体素为单位。
需要将病人坐标系中的结节中心坐标转换为数组坐标(I, R, C),以便从 CT 数据中提取感兴趣区域。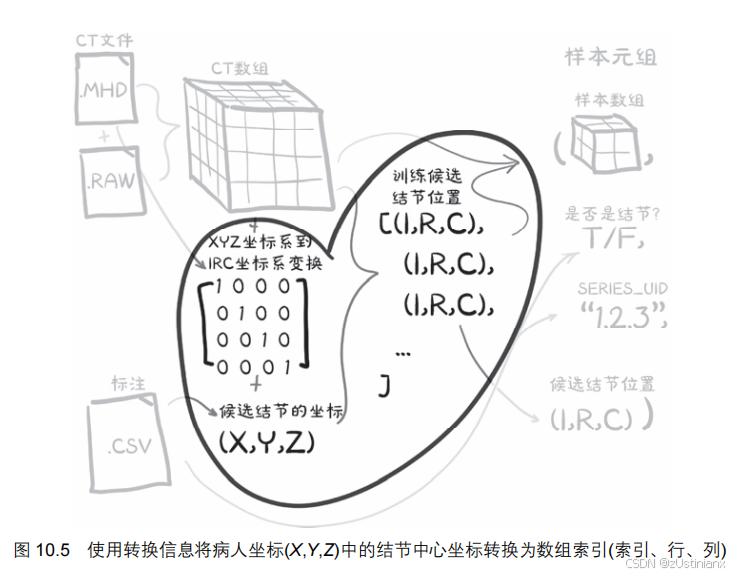
-
坐标转换
-
从病人坐标系到数组坐标系:通过方向矩阵、体素大小和原点偏移量进行转换。
-
从数组坐标系到病人坐标系:执行逆操作。
实现了xyz2irc()和irc2xyz()函数,用于完成这两种转换。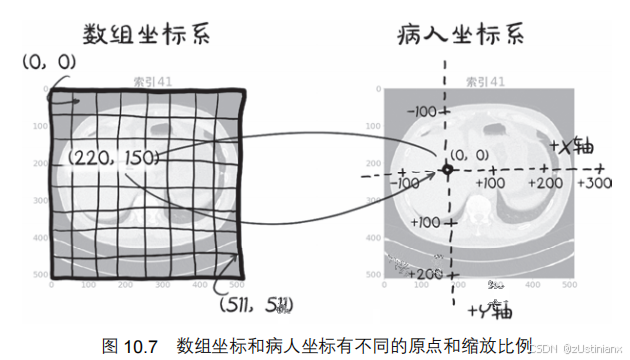
-
10.5 一个简单的数据集实现
-
PyTorch 数据集类
实现一个自定义的 PyTorch 数据集类LunaDataset,用于将 CT 数据和标注数据整合为模型训练所需的格式。 -
实现方法
-
__len__()方法:返回数据集的长度,即候选结节的数量。 -
__getitem__()方法:根据索引返回一个样本元组,包含:-
结节的 CT 数据块(三维数组)。
-
结节状态的分类张量(0 表示非结节,1 表示结节)。
-
序列 UID 和结节中心的数组坐标。
-
-
-
数据缓存
使用functools.lru_cache和diskcache对数据进行缓存,以提高数据加载效率。
缓存机制包括:-
内存缓存:缓存
Ct实例。 -
磁盘缓存:缓存提取的结节数据块。
-
-
训练集与验证集分离
通过val_stride参数将数据集划分为训练集和验证集。-
如果
isValSet_bool为True,则返回验证集。 -
如果
isValSet_bool为False,则返回训练集。
-
10.6 数据可视化
-
数据呈现
使用 Matplotlib 和 Jupyter Notebook 的内联魔法函数%matplotlib inline,展示 CT 扫描和结节切片的图像。
提供了findPositiveSamples()和showCandidate()函数,用于快速定位和展示正样本。





















 1634
1634

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








