这个目前还有点问题 没有很好的进行ROI分割提取肺部,而是单独把整个ct 做个肺部提取了, 正常应该提取左 右肺部2个分割,这个后面找时间再优化补充一下,这里先把已经搞的记录一下,我想 无非是先预处理一下图像 再提取?或者不用opencv 而用其他的lib 进行提取操作?或者参数有问题?
完整代码如下:
import pydicom
import numpy as np
import cv2
import matplotlib.pyplot as plt
from skimage import morphology
plt.rcParams['font.sans-serif'] = ['SimHei'] # 使用 SimHei 字体
plt.rcParams['axes.unicode_minus'] = False # 解决负号显示问题
plt.rcParams['font.size'] = 10 # 设置全局字体大小
def load_dicom_image(dicom_path):
"""
加载DICOM文件并转换为HU值
"""
ds = pydicom.dcmread(dicom_path)
pixel_array = ds.pixel_array
# 转换为HU值
if hasattr(ds, 'RescaleSlope') and hasattr(ds, 'RescaleIntercept'):
hu_image = pixel_array * ds.RescaleSlope + ds.RescaleIntercept
else:
hu_image = pixel_array
return hu_image, ds
def apply_window(image, window_width, window_level):
"""
应用窗宽窗位调整图像
"""
window_min = window_level - window_width / 2
window_max = window_level + window_width / 2
windowed_image = np.clip(image, window_min, window_max)
normalized = ((windowed_image - window_min) / (window_width)) * 255
return normalized.astype(np.uint8)
def segment_lung(hu_image, window_width=1642, window_level=-234, threshold=25, min_area_ratio=0.01):
"""
分割肺部区域 (ROI)
"""
# 应用肺窗增强肺部区域
lung_windowed = apply_window(hu_image, window_width, window_level)
# 可视化肺窗图像
plt.figure(figsize=(10, 5))
plt.subplot(1, 2, 1)
plt.imshow(lung_windowed, cmap='gray')
plt.title(f"肺窗图像 (WW={window_width}, WL={window_level})")
plt.axis('off')
# 二值化处理
_, binary = cv2.threshold(lung_windowed, threshold, 255, cv2.THRESH_BINARY)
plt.subplot(1, 2, 2)
plt.imshow(binary, cmap='gray')
plt.title(f"二值化图像 (阈值={threshold})")
plt.axis('off')
plt.show()
# 形态学操作 - 去除小噪点
kernel = cv2.getStructuringElement(cv2.MORPH_ELLIPSE, (5, 5))
cleaned = cv2.morphologyEx(binary, cv2.MORPH_OPEN, kernel, iterations=2)
filled = cv2.morphologyEx(cleaned, cv2.MORPH_CLOSE, kernel, iterations=3)
# 可视化形态学处理后的图像
plt.figure(figsize=(10, 5))
plt.subplot(1, 2, 1)
plt.imshow(cleaned, cmap='gray')
plt.title("形态学开运算")
plt.axis('off')
plt.subplot(1, 2, 2)
plt.imshow(filled, cmap='gray')
plt.title("形态学闭运算")
plt.axis('off')
plt.show()
# 提取连通区域
num_labels, labels, stats, centroids = cv2.connectedComponentsWithStats(filled)
# 计算最小面积阈值
min_area = min_area_ratio * hu_image.size
# 选择面积大于阈值的区域
valid_labels = []
for i in range(1, num_labels): # 跳过背景(0)
if stats[i, cv2.CC_STAT_AREA] > min_area:
valid_labels.append(i)
if not valid_labels:
# 显示连通区域分析结果
plt.figure(figsize=(12, 6))
plt.subplot(1, 2, 1)
plt.imshow(filled, cmap='gray')
plt.title("形态学处理后的图像")
plt.axis('off')
plt.subplot(1, 2, 2)
# 使用伪彩色显示连通区域
labels_display = labels.astype(np.float32) / num_labels
plt.imshow(labels_display, cmap='jet')
plt.title(f"连通区域分析 ({num_labels - 1}个区域)")
plt.axis('off')
plt.colorbar(label='区域ID')
plt.show()
raise ValueError(f"未检测到有效的肺部区域 (最小面积={min_area:.0f}像素)")
lung_mask = np.zeros_like(labels, dtype=np.uint8)
for label in valid_labels:
lung_mask[labels == label] = 255
# 显示最终肺部分割结果
plt.figure(figsize=(10, 5))
plt.subplot(1, 2, 1)
plt.imshow(lung_windowed, cmap='gray')
plt.title("肺窗图像")
plt.axis('off')
plt.subplot(1, 2, 2)
plt.imshow(lung_mask, cmap='gray')
plt.title(f"肺部ROI分割 (检测到{len(valid_labels)}个区域)")
plt.axis('off')
plt.show()
return lung_mask
def crop_and_scale_roi(hu_image, mask, padding=10, target_size=(256, 256)):
"""
裁剪并缩放感兴趣区域
"""
# 查找轮廓
contours, _ = cv2.findContours(mask, cv2.RETR_EXTERNAL, cv2.CHAIN_APPROX_SIMPLE)
if not contours:
# 显示原始掩码帮助调试
plt.figure(figsize=(8, 4))
plt.subplot(1, 2, 1)
plt.imshow(mask, cmap='gray')
plt.title("肺部掩码")
plt.axis('off')
plt.subplot(1, 2, 2)
plt.imshow(apply_window(hu_image, 400, 40), cmap='gray')
plt.title("原始CT图像")
plt.axis('off')
plt.show()
raise ValueError("未找到有效的轮廓")
# 获取所有轮廓的联合边界框
x_min, y_min, x_max, y_max = float('inf'), float('inf'), 0, 0
for contour in contours:
x, y, w, h = cv2.boundingRect(contour)
x_min = min(x_min, x)
y_min = min(y_min, y)
x_max = max(x_max, x + w)
y_max = max(y_max, y + h)
# 添加填充
x_min = max(0, x_min - padding)
y_min = max(0, y_min - padding)
x_max = min(hu_image.shape[1], x_max + padding)
y_max = min(hu_image.shape[0], y_max + padding)
# 确保边界框有效
if x_max <= x_min or y_max <= y_min:
plt.figure(figsize=(8, 4))
plt.subplot(1, 2, 1)
plt.imshow(mask, cmap='gray')
plt.title("肺部掩码")
plt.axis('off')
plt.subplot(1, 2, 2)
plt.imshow(apply_window(hu_image, 400, 40), cmap='gray')
plt.plot([x_min, x_max, x_max, x_min, x_min],
[y_min, y_min, y_max, y_max, y_min], 'r-')
plt.title(f"无效边界框: x={x_min}-{x_max}, y={y_min}-{y_max}")
plt.axis('off')
plt.show()
raise ValueError(f"无效的边界框坐标: x={x_min}-{x_max}, y={y_min}-{y_max}")
# 裁剪ROI
cropped_hu = hu_image[y_min:y_max, x_min:x_max]
cropped_mask = mask[y_min:y_max, x_min:x_max]
# 应用掩码 (可选)
# masked_roi = cv2.bitwise_and(cropped_hu, cropped_hu, mask=cropped_mask)
# 缩放图像
scaled_hu = cv2.resize(cropped_hu, target_size, interpolation=cv2.INTER_LINEAR)
scaled_mask = cv2.resize(cropped_mask, target_size, interpolation=cv2.INTER_NEAREST)
return scaled_hu, scaled_mask, (x_min, y_min, x_max, y_max)
def visualize_results(hu_image, mask, roi_coords, scaled_hu, scaled_mask):
"""
可视化处理结果
"""
plt.figure(figsize=(15, 10))
# 原始HU图像 (软组织窗)
plt.subplot(2, 3, 1)
soft_tissue = apply_window(hu_image, 400, 40)
plt.imshow(soft_tissue, cmap='gray')
plt.title("原始CT图像 (软组织窗)")
plt.axis('off')
# 肺部窗图像
plt.subplot(2, 3, 2)
lung_window = apply_window(hu_image, 1642, -234)
plt.imshow(lung_window, cmap='gray')
plt.title("肺窗图像")
plt.axis('off')
# 肺部掩码
plt.subplot(2, 3, 3)
plt.imshow(mask, cmap='gray')
plt.title("肺部ROI分割")
plt.axis('off')
# 原始图像上显示ROI框
plt.subplot(2, 3, 4)
plt.imshow(soft_tissue, cmap='gray')
x_min, y_min, x_max, y_max = roi_coords
cv2.rectangle(soft_tissue, (x_min, y_min), (x_max, y_max), (255, 0, 0), 2)
plt.imshow(soft_tissue, cmap='gray')
plt.title("ROI定位框")
plt.axis('off')
# 裁剪后的ROI (肺窗)
plt.subplot(2, 3, 5)
cropped_lung = apply_window(hu_image[y_min:y_max, x_min:x_max], 1670, -234)
plt.imshow(cropped_lung, cmap='gray')
plt.title(f"裁剪的ROI ({cropped_lung.shape[1]}x{cropped_lung.shape[0]})")
plt.axis('off')
# 缩放后的ROI
plt.subplot(2, 3, 6)
scaled_lung = apply_window(scaled_hu, 1670, -234)
plt.imshow(scaled_lung, cmap='gray')
plt.title(f"缩放后的ROI ({target_size[0]}x{target_size[1]})")
plt.axis('off')
plt.tight_layout()
plt.show()
# 主程序
if __name__ == "__main__":
# 配置参数
dicom_path = "Anonymized_20250720/series-00002/image-00130.dcm" # 替换为你的DICOM文件路径
padding = 15 # ROI周围的填充像素
target_size = (256, 256) # 缩放目标尺寸
# 可调整的分割参数
lung_window_width = 1670 # 肺窗宽
lung_window_level = -234 # 肺窗位
binary_threshold = 25 # 二值化阈值
min_area_ratio = 0.005 # 最小区域面积比例 (相对于图像总面积)
try:
# 1. 加载DICOM图像
hu_image, ds = load_dicom_image(dicom_path)
print(f"图像尺寸: {hu_image.shape}, HU值范围: {np.min(hu_image):.0f} 到 {np.max(hu_image):.0f}")
# 显示原始图像
plt.figure(figsize=(10, 5))
plt.subplot(1, 2, 1)
plt.imshow(apply_window(hu_image, 400, 40), cmap='gray')
plt.title("软组织窗 (WW=400, WL=40)")
plt.axis('off')
plt.subplot(1, 2, 2)
plt.imshow(apply_window(hu_image, lung_window_width, lung_window_level), cmap='gray')
plt.title(f"肺窗 (WW={lung_window_width}, WL={lung_window_level})")
plt.axis('off')
plt.suptitle("原始CT图像")
plt.tight_layout()
plt.show()
# 2. 分割肺部ROI
lung_mask = segment_lung(
hu_image,
window_width=lung_window_width,
window_level=lung_window_level,
threshold=binary_threshold,
min_area_ratio=min_area_ratio
)
# 3. 裁剪和缩放ROI
scaled_hu, scaled_mask, roi_coords = crop_and_scale_roi(
hu_image, lung_mask, padding, target_size
)
print(f"ROI坐标: x={roi_coords[0]}-{roi_coords[2]}, y={roi_coords[1]}-{roi_coords[3]}")
print(f"缩放后尺寸: {scaled_hu.shape}")
# 4. 可视化结果
visualize_results(hu_image, lung_mask, roi_coords, scaled_hu, scaled_mask)
# 5. 可选: 保存处理后的图像
# 保存肺部窗的缩放ROI
output_image = apply_window(scaled_hu, lung_window_width, lung_window_level)
cv2.imwrite("lung_roi_resized.png", output_image)
# 保存掩码
cv2.imwrite("lung_mask.png", scaled_mask)
except Exception as e:
print(f"处理过程中出错: {str(e)}")
# 如果可能,显示HU直方图帮助诊断
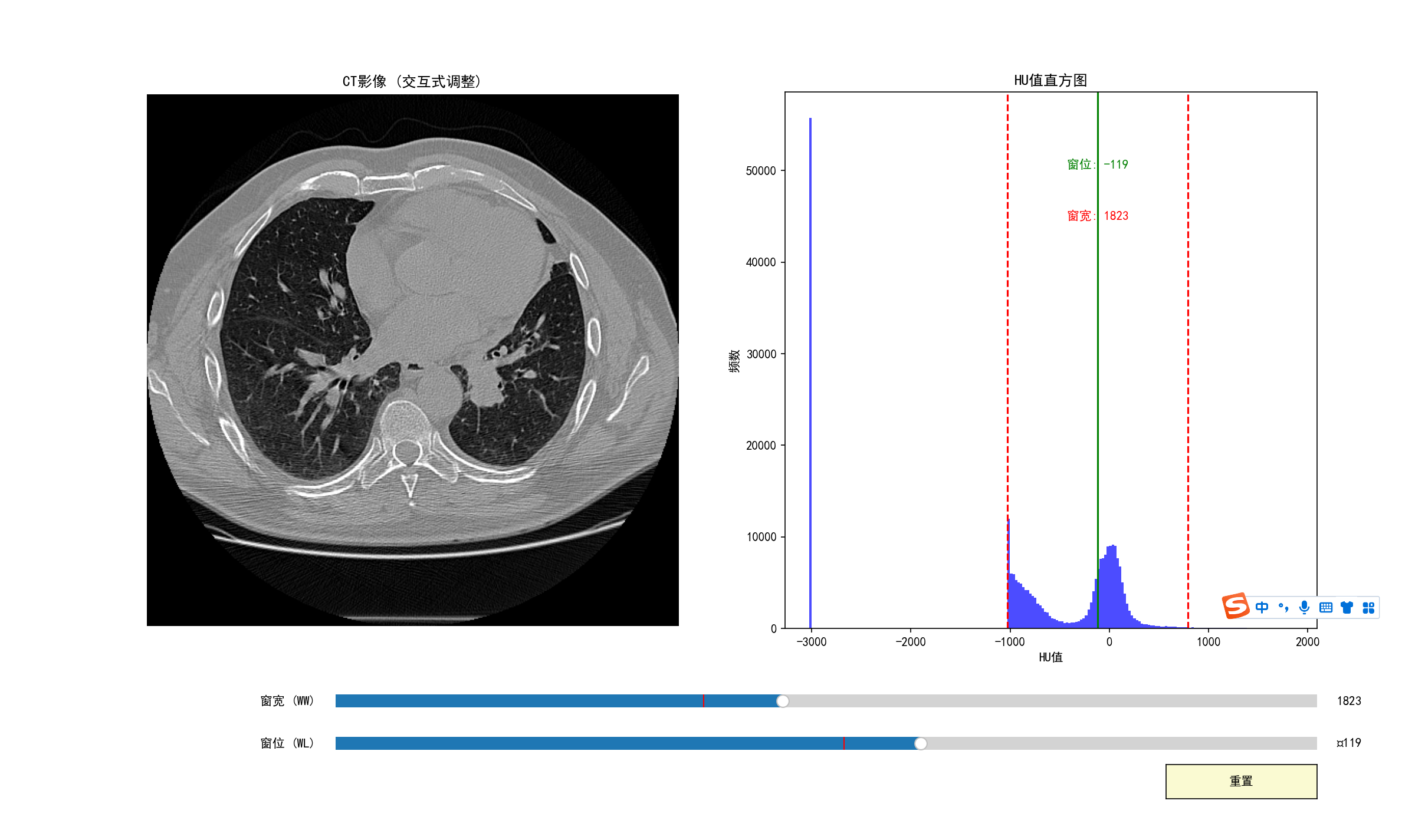
if 'hu_image' in locals():
plt.figure(figsize=(10, 4))
plt.hist(hu_image.flatten(), bins=200, color='blue')
plt.axvline(x=lung_window_level, color='red', linestyle='--', label=f'窗位={lung_window_level}')
plt.axvline(x=lung_window_level - lung_window_width / 2, color='green', linestyle='--', label='窗宽下限')
plt.axvline(x=lung_window_level + lung_window_width / 2, color='green', linestyle='--', label='窗宽上限')
plt.title("HU值直方图")
plt.xlabel("HU值")
plt.ylabel("频数")
plt.legend()
plt.show()
运行结果如图:
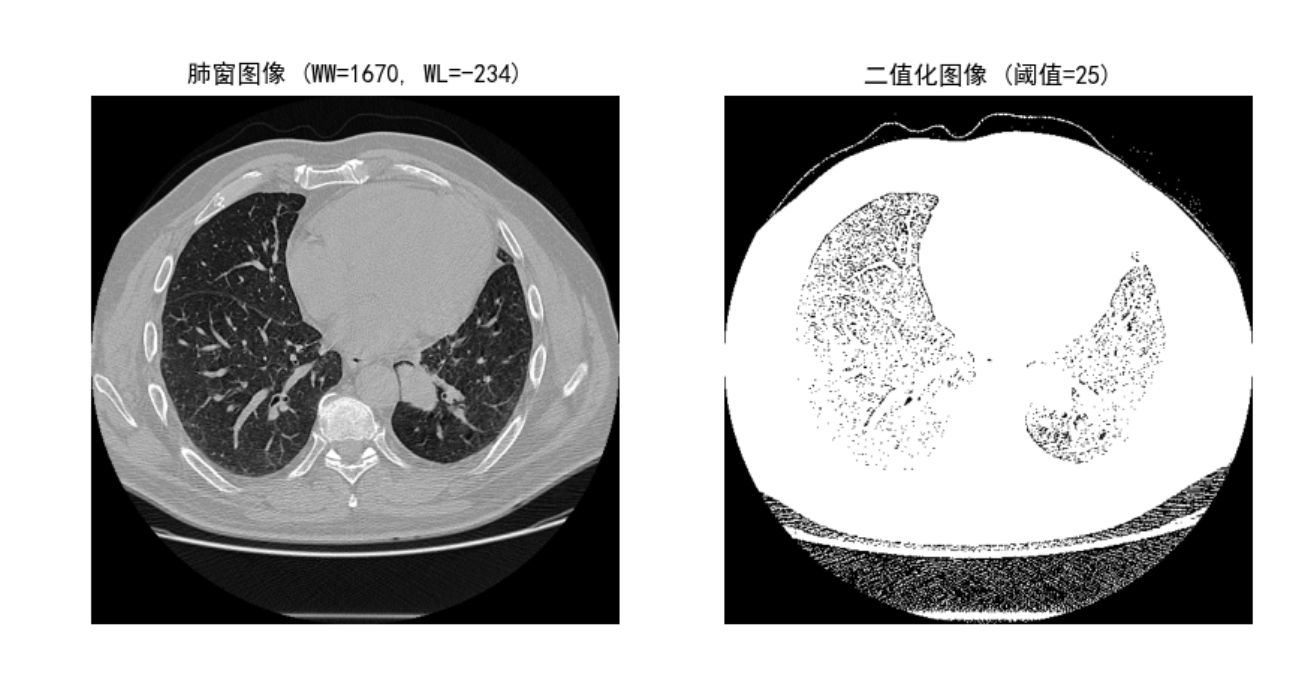

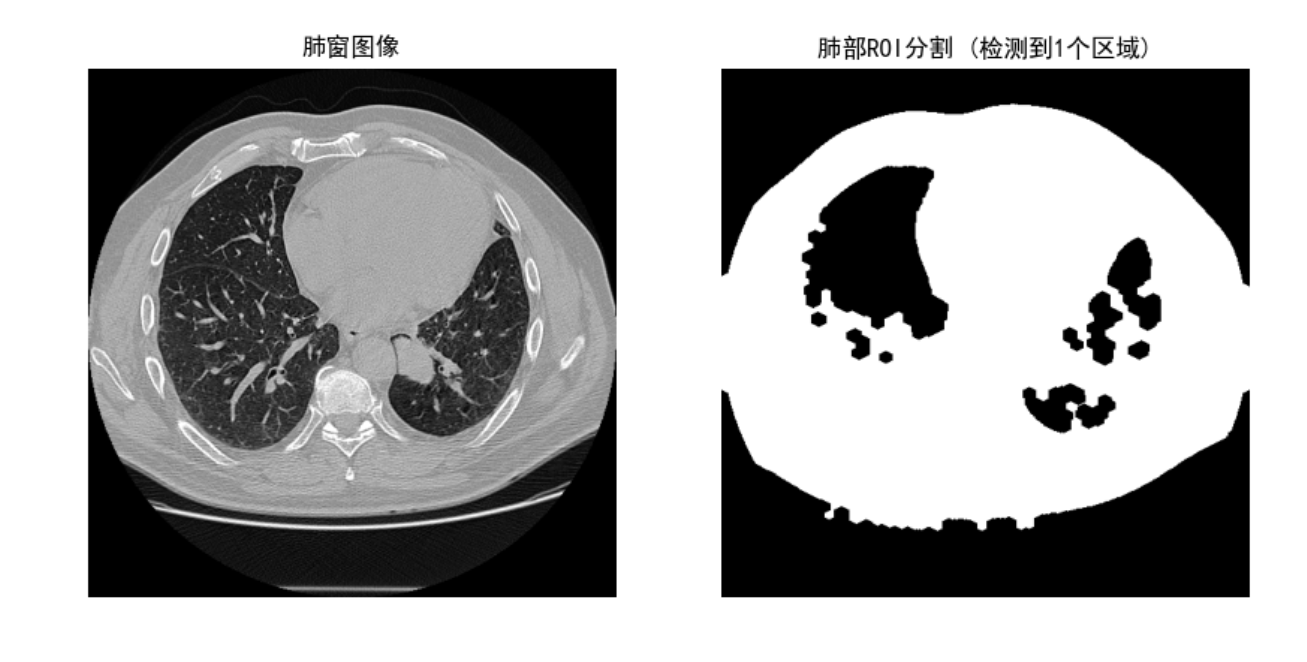
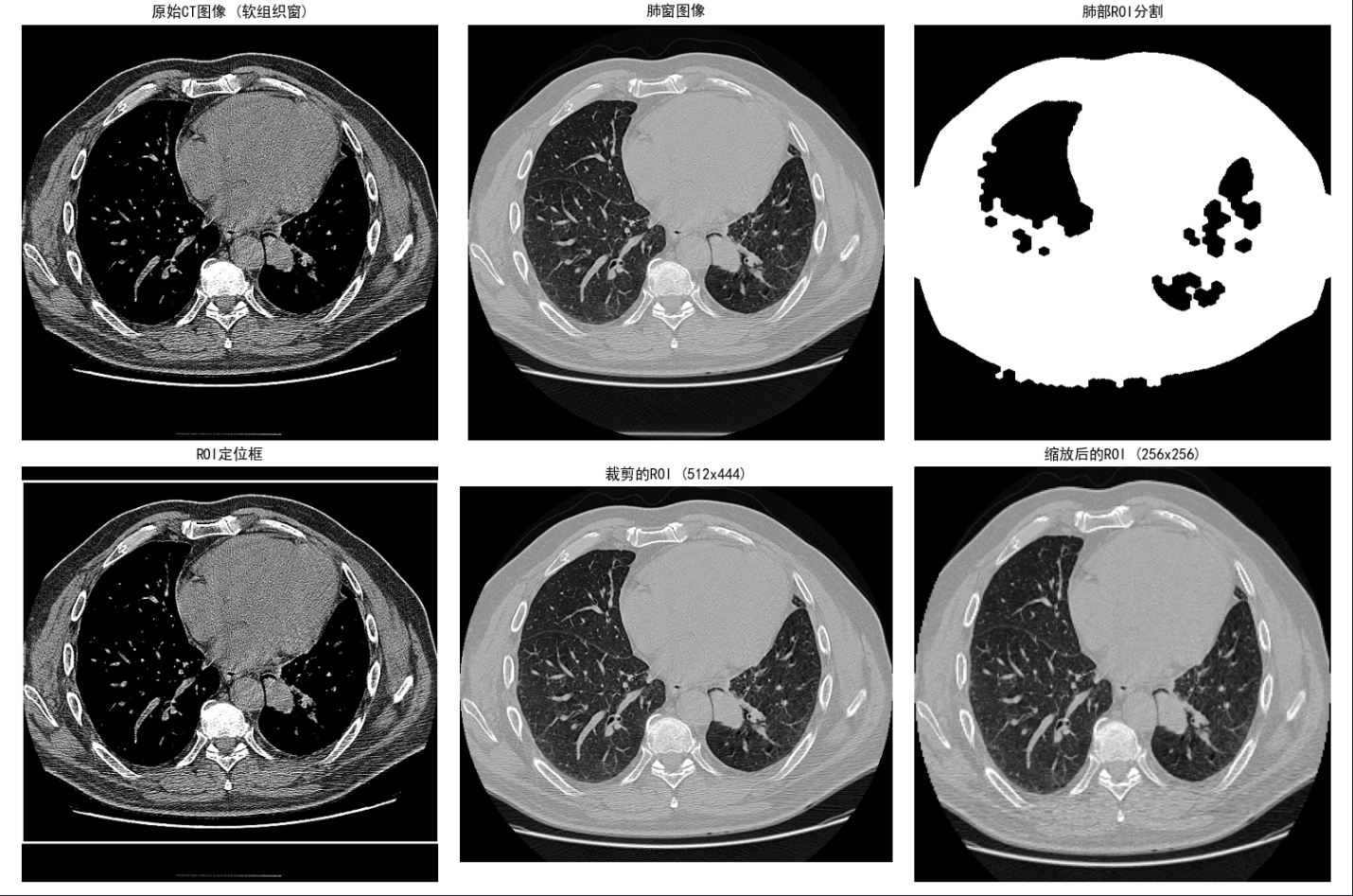
这里 roi 定位框有问题
1. 环境设置与字体配置
import pydicom
import numpy as np
import cv2
import matplotlib.pyplot as plt
from skimage import morphology
plt.rcParams['font.sans-serif'] = ['SimHei'] # 使用 SimHei 字体
plt.rcParams['axes.unicode_minus'] = False # 解决负号显示问题
plt.rcParams['font.size'] = 10 # 设置全局字体大小
-
作用:导入必要的库并设置中文字体显示
-
关键点:
-
pydicom:用于读取DICOM格式的医疗影像 -
numpy:处理图像数据 -
cv2:图像处理和计算机视觉功能 -
matplotlib:图像可视化 -
skimage.morphology:形态学操作 -
字体设置确保中文标签正常显示
-
2. DICOM图像加载函数
def load_dicom_image(dicom_path):
ds = pydicom.dcmread(dicom_path)
pixel_array = ds.pixel_array
# 转换为HU值
if hasattr(ds, 'RescaleSlope') and hasattr(ds, 'RescaleIntercept'):
hu_image = pixel_array * ds.RescaleSlope + ds.RescaleIntercept
else:
hu_image = pixel_array
return hu_image, ds
-
功能:加载DICOM文件并转换为Hounsfield单位(HU)
-
关键点:
-
dcmread()读取DICOM文件 -
使用
RescaleSlope和RescaleIntercept将原始像素值转换为HU值 -
HU值是CT影像的标准单位,其中:
-
水 = 0 HU
-
空气 = -1000 HU
-
骨骼 > 400 HU
-
-
3. 窗宽窗位调整函数
def apply_window(image, window_width, window_level):
window_min = window_level - window_width / 2
window_max = window_level + window_width / 2
windowed_image = np.clip(image, window_min, window_max)
normalized = ((windowed_image - window_min) / (window_width)) * 255
return normalized.astype(np.uint8)
-
功能:应用窗宽窗位调整图像显示范围
-
关键点:
-
window_width:窗宽,决定图像对比度 -
window_level:窗位,决定图像亮度中心 -
将HU值映射到0-255灰度范围
-
窗宽窗位原理:
-
窄窗宽 → 高对比度(适合观察组织间微小差异)
-
宽窗宽 → 低对比度(适合观察大范围密度差异)
-
-
4. 肺部ROI分割函数
def segment_lung(hu_image, window_width=1642, window_level=-234, threshold=25, min_area_ratio=0.01):
# 应用肺窗
lung_windowed = apply_window(hu_image, window_width, window_level)
# 二值化处理
_, binary = cv2.threshold(lung_windowed, threshold, 255, cv2.THRESH_BINARY)
# 形态学操作
kernel = cv2.getStructuringElement(cv2.MORPH_ELLIPSE, (5, 5))
cleaned = cv2.morphologyEx(binary, cv2.MORPH_OPEN, kernel, iterations=2)
filled = cv2.morphologyEx(cleaned, cv2.MORPH_CLOSE, kernel, iterations=3)
# 提取连通区域
num_labels, labels, stats, centroids = cv2.connectedComponentsWithStats(filled)
# 计算最小面积阈值
min_area = min_area_ratio * hu_image.size
# 选择面积大于阈值的区域
valid_labels = []
for i in range(1, num_labels):
if stats[i, cv2.CC_STAT_AREA] > min_area:
valid_labels.append(i)
# ... [错误处理和可视化] ...
return lung_mask
-
功能:分割肺部区域
-
关键参数:
-
window_width=1670,window_level=-234:肺窗参数 -
threshold=25:二值化阈值 -
min_area_ratio=0.005:最小区域面积比例
-
-
处理流程:
-
应用肺窗突出肺部区域
-
二值化分离肺部组织
-
形态学操作(开运算去噪,闭运算填充)
-
连通区域分析
-
筛选面积足够大的区域作为肺部ROI
-
5. ROI裁剪与缩放函数
def crop_and_scale_roi(hu_image, mask, padding=10, target_size=(256, 256)):
# 查找轮廓
contours, _ = cv2.findContours(mask, cv2.RETR_EXTERNAL, cv2.CHAIN_APPROX_SIMPLE)
# 计算边界框
x_min, y_min, x_max, y_max = float('inf'), float('inf'), 0, 0
for contour in contours:
x, y, w, h = cv2.boundingRect(contour)
# ... 更新边界坐标 ...
# 添加填充
x_min = max(0, x_min - padding)
# ... 其他边界处理 ...
# 裁剪ROI
cropped_hu = hu_image[y_min:y_max, x_min:x_max]
# 缩放图像
scaled_hu = cv2.resize(cropped_hu, target_size, interpolation=cv2.INTER_LINEAR)
return scaled_hu, scaled_mask, (x_min, y_min, x_max, y_max)
-
功能:裁剪感兴趣区域并进行缩放
-
参数:
-
padding=15:ROI周围的填充像素 -
target_size=(256, 256):目标缩放尺寸
-
-
处理流程:
-
查找ROI轮廓
-
计算包含所有ROI的边界框
-
添加填充避免裁剪边缘
-
裁剪ROI区域
-
缩放至目标尺寸
-
6. 结果可视化函数
-
功能:可视化处理全过程
-
展示内容:
-
原始CT图像(软组织窗)
-
肺窗图像
-
肺部ROI分割结果
-
原始图像上的ROI定位框
-
裁剪后的ROI
-
缩放后的ROI
-
7. 主程序流程
if __name__ == "__main__":
# 配置参数
dicom_path = "Anonymized_20250720/series-00002/image-00130.dcm"
padding = 15
target_size = (256, 256)
# 分割参数
lung_window_width = 1670
lung_window_level = -234
binary_threshold = 25
min_area_ratio = 0.005
try:
# 1. 加载DICOM图像
hu_image, ds = load_dicom_image(dicom_path)
# 2. 显示原始图像
# 3. 分割肺部ROI
lung_mask = segment_lung(...)
# 4. 裁剪和缩放ROI
scaled_hu, scaled_mask, roi_coords = crop_and_scale_roi(...)
# 5. 可视化结果
visualize_results(...)
# 6. 保存处理后的图像
cv2.imwrite("lung_roi_resized.png", ...)
except Exception as e:
# 错误处理和诊断
print(f"处理过程中出错: {str(e)}")
# 显示HU直方图帮助诊断
plt.hist(hu_image.flatten(), bins=200, color='blue')
# ... 标记窗宽窗位 ...
plt.show()
-
主要步骤:
-
加载DICOM图像
-
显示原始图像(软组织窗和肺窗)
-
分割肺部ROI
-
裁剪和缩放ROI
-
可视化处理结果
-
保存处理后的图像
-
-
错误处理:
-
捕获并显示异常信息
-
显示HU直方图帮助诊断问题
-
一般参数
1. 肺窗参数 (lung_window_width, lung_window_level)
-
典型值:WW=1500, WL=-600 我这里设置的是根据图像情况调整了的结果
2. 二值化阈值 (binary_threshold)
-
典型值:30-40
-
调整建议:
-
如果肺部区域未完全提取 → 降低阈值(如20)
-
如果背景噪声过多 → 提高阈值(如40)
-
您的设置:25(较低,可能包含更多噪声)
-
3. 最小区域面积比例 (min_area_ratio)
-
典型值:0.01(约占总像素1%)
-
调整建议:
-
如果小区域被忽略 → 降低比例(如0.005)
-
如果噪声区域被包含 → 提高比例(如0.02)
-
您的设置:0.005(较低,适合保留小区域)
-
常见问题诊断
错误:"未检测到有效的肺部区域"
可能原因:
-
肺窗参数不匹配
-
窗宽窗位设置不当,肺部区域未正确显示
-
解决方案:调整
lung_window_width和lung_window_level
-
-
二值化阈值不当
-
阈值过高或过低,肺部区域未正确分离
-
解决方案:调整
binary_threshold
-
-
图像不包含完整肺部
-
当前切片位于肺部区域外(如颈部或腹部)
-
解决方案:选择包含肺部的切片
-
-
最小面积设置过大
-
min_area_ratio设置过高,小区域被过滤 -
解决方案:降低
min_area_ratio
-
诊断工具:
-
HU直方图:显示图像中所有HU值的分布
-
肺部区域通常在-1000到-400 HU之间
-
窗宽窗位应在肺部区域中心
-
-
肺窗图像:查看肺窗调整后效果
-
二值化图像:检查肺部区域是否被正确提取
优化建议
-
自适应窗宽窗位:
# 根据图像特性自动计算窗宽窗位
def auto_lung_window(hu_image):
lung_mask = (hu_image < -400) & (hu_image > -1000)
if np.any(lung_mask):
lung_values = hu_image[lung_mask]
wl = np.median(lung_values)
ww = np.ptp(lung_values) * 1.5
return ww, wl
else:
return 1500, -600 # 默认值
-
自适应阈值:
# 使用Otsu自适应阈值
_, binary = cv2.threshold(lung_windowed, 0, 255, cv2.THRESH_BINARY_INV + cv2.THRESH_OTSU)
-
多切片处理:
# 处理整个DICOM系列
dicom_files = sorted(glob.glob("Anonymized_20250720/series-00002/*.dcm"))
for file in dicom_files:
try:
hu_image, ds = load_dicom_image(file)
# ... 处理逻辑 ...
except Exception as e:
print(f"处理 {file} 时出错: {str(e)}")
这个CT影像肺部ROI分割代码实现了完整的处理流程:
-
DICOM图像加载和HU值转换
-
肺窗调整优化肺部显示
-
二值化和形态学操作提取肺部区域
-
连通区域分析筛选有效ROI
-
ROI裁剪和缩放
-
结果可视化和保存
当出现"未检测到有效的肺部区域"错误时,应通过:
-
检查肺窗参数是否合适
-
调整二值化阈值
-
降低最小面积比例
-
验证图像是否包含肺部区域
通过HU直方图和中间处理步骤的图像,可以有效诊断问题原因并进行参数优化。






















 1万+
1万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








