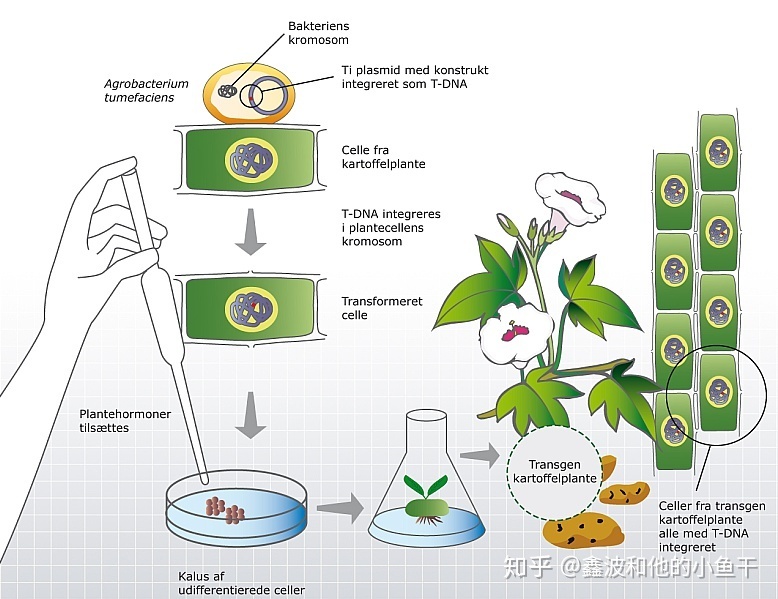
农杆菌作为一种天然高效的转基因载体,在植物的遗传转化上具有广泛的应用。它能在一定条件下趋化性地感染植物的受伤部位,并诱导产生冠瘿瘤或发状根。农杆菌转化时植物一定要受伤吗 农杆菌属的强毒株可以在自然条件下自发地遗传转化多种植物。在实验室条件下,它们还可以用于转化酵母,真菌和动物细胞。
农杆菌菌株将单链形式的T-DNA(T链)和毒力效应蛋白(Vir)转移到植物细胞中。转移后,T链可能与Vir和植物蛋白形成复合物,然后通过细胞质运输并进入细胞核。随后可以将T链随机整合到植物基因组(染色体上)并持续表达T链上编码的基因,这一过程称为稳定转化。T-DNA进入细胞后,即使未整合到宿主染色体,也能有T-DNA编码基因的瞬时表达,但转入基因的稳定表达(稳定转化)则需要T-DNA整合。
T-DNA整合到宿主基因组中的分子过程仍然未知,T-DNA分子的结构和整合基因组位置可能会影响转基因表达,研究T-DNA整合及其调控对于基础研究和农业生物技术有重要意义。
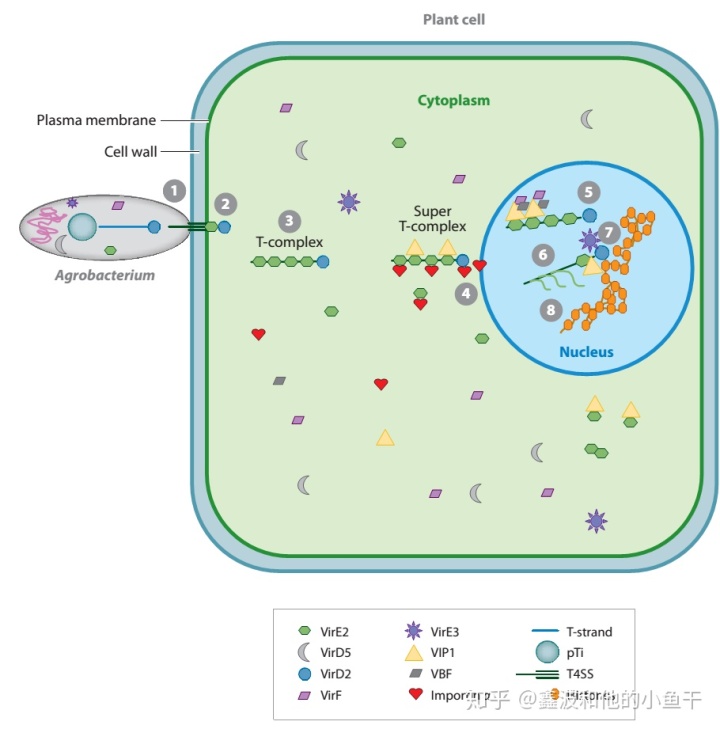
农杆菌介导的转化由多个阶段组成:1。农杆菌附着在植物细胞上;2.T链和毒力效应蛋白通过植物质膜进入植物细胞;3.T复合体和超T复合体的形成及其胞质转运;4. 进入细胞核;5. 超T复合物对染色质的靶向作用;6. 在T-DNA整合前从超T复合体中去除蛋白;7.T-DNA整合到植物基因组;8. 转基因表达。
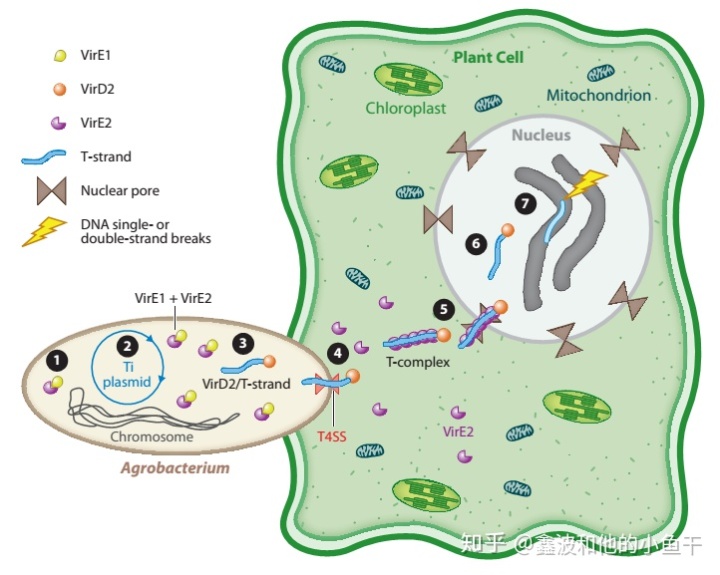
所有有毒农杆菌菌株都带有诱导冠瘿瘤的质粒(Ti)或诱导发状根的质粒(Ri)。Ti 质粒或Ri 质粒携带编码毒力蛋白(vir)的基因,这些毒蛋白基因的表达受到植物释放的酚类物质诱导。在细菌附着到植物细胞之前或附着时发生vir基因的诱导。
转化最初是农杆菌附着在植物细胞上。植物组织的受伤诱导产生多种防御化合物,包括一些酚类化合物,① 此时VirA-VirG两组分调节系统感知植物的酚和糖分子,从而诱导vir基因的表达和Vir蛋白的产生(包括解旋酶VirD1和核酸酶VirD2)。
随后完成T-DNA的切割。Ti/Ri质粒的T-DNA区域,通常位于左边界(LB)和右边界(RB)之间,左右两个边界是两个由特定核酸序列组成的25bp侧翼重复序列。② VirD1和VirD2协同作用在Ti/Ri质粒的LB和RB边界重复序列处均形成切口并切下T-DNA区域。产生的T-DNA通常包含LB的4–25bp和RB的1–3bp。在切刻过程中,③ VirD2共价附于一条T链RB的5'端,形成VirD2-T链复合物。
之后T链和毒力效应蛋白通过植物质膜进入植物细胞。④ VirD2-T链通过诱导的IV型分泌系统T4SS途径离开细菌,进入植物。该途径还输出其他几种Vir效应蛋白,如根癌农杆菌的VirD5、VirE2、VirE3和VirF。尽管VirE2在细菌中与其伴侣蛋白VirE1协同作用,但是只有VirE2被转运到植物中。
随后,转运进细胞的组分在植物体内形成T复合体和超T复合体。 ⑤ 在植物细胞内,单链DNA结合蛋白VirE2可能非共价包覆T链,形成VirD2/VirE2/T-DNA复合物,避免T-DNA被降解或截短。VirD2包含强大的C端核定位信号序列,能引导T复合物穿过植物细胞质,通过核孔进入细胞核。
进入细胞核以后,T-DNA将靶向植物基因组进行整合,整合前会从超T复合体中去除蛋白,⑥ VirE2从T链解离,而后 ⑦ T-DNA整合到植物基因组。T-DNA的整合是随机的,不会针对基因组的特定区域进行整合。
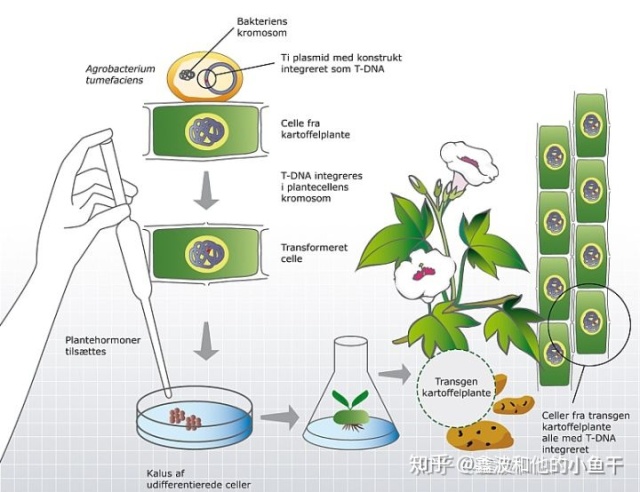
实验室条件下,利用农杆菌侵染获得的转基因后代,通常要经过选择标记筛选(抗性、荧光等),获得阳性转化苗。实际上,许多转基因后代植物都含有沉默的转基因。如果T-DNA整合到基因组转录活性较低的区域(如着丝粒,端粒,异染色质等),转入的基因(包括选择标记)可能不会正常表达。尽管T-DNA整合确实是随机的,但参与染色质修饰的蛋白质可能在整合中起一定作用。
体外实验中,VirD2不能将单链DNA连接到模拟靶序列,但可以介导T-DNA切割反应的逆反应,表明VirD2不具有通用的连接活性。 植物提取物可以以ATP依赖的方式将T-DNA连入模拟靶序列,表明T-DNA整合取决于植物连接酶。但是,进入植物细胞的T链可能没有直接连接到植物基因组的断裂上。相反,T链可以被宿主DNA聚合酶复制,并且可以在复制过程中连接到基因组。
T-DNA在整合到植物基因组时可能发生单插入(在一个基因座上)或多插入(在多个基因座上),这些基因座可能会在后代中分离。整合的T-DNA拷贝数在单个到几十个不等。这种拷贝数差异并非总是由于农杆菌菌株、植物物种或转化方案不同而引起,因为即使在同一次转基因事件中,转入受体的T-DNA拷贝数也会发生变化。单插入(单个T-DNA整合)的重要性不仅在于分析的相对简便性,还在于单拷贝整合子被转基因沉默的可能性降低。
农杆菌介导转化的另一个限制是转基因表达的可变性和稳定性。相同农杆菌菌株产生的独立转基因事件通常显示出很广的转基因表达范围,从很高的表达量到完全沉默。转基因表达的变异性使转基因事件的评估变得困难,通常需要产生并检测数百株转基因植物,以鉴定出几株具有高表达水平且表达稳定的转基因后代。

T-DNA已用于在拟南芥、水稻、玉米和短枝曲霉中产生较大的插入突变(敲除)文库。在这些文库中对T-DNA侧翼序列进行测序发现,在连接的T-DNA部分中,RB通常比LB更精确完整。RB连接序列的相对精度归因于与RB共价连接的VirD2蛋白可以保护它免受核酸外切酶消化。而较大的LB侧翼缺失被归因于LB末端区域缺乏保护。
T-DNA与植物DNA的连接通常不干净,整合到植物序列中的T-DNA边界往往不精确,在T-DNA和植物DNA连接处也经常发现植物染色体出现删除、插入和重排等异常。转基因株系T-DNA整合位点以外的基因组区域也经常见到缺失。然而尚不清楚这些整合位点以外的缺失是由T-DNA整合造成的还是由于植物组织培养过程造成的。染色体倒位和易位通常与T-DNA插入有关,从Salk收集的T-DNA插入序列中有19%具有染色体易位。这些发现表明,T-DNA整合可能与多个位点的双链DNA断裂有关。
T-DNA的整合类似于通过非同源末端连接(NHEJ)或微同源介导的末端连接(MMEJ)过程进行的DNA断裂修复。双链DNA断裂被错误修复是很常见的分子事件,这将导致断裂位点的缺失、插入、重排甚至易位。X射线辐照的植物原生质体具有增加的双链DNA断裂,也显示出更高的转化率,表明DNA双链断裂(或诱导损伤修复酶)增强了外源DNA的整合。
实际上,在植物中利用农杆菌介导基因靶向整合的效率非常低,T-DNA不必与靶整合位点具有同源性。NHEJ似乎是T-DNA整合的首选机制,但是尽管与DNA修复和重组相关的蛋白可能会影响T-DNA整合,这些途径都不是必不可少的。
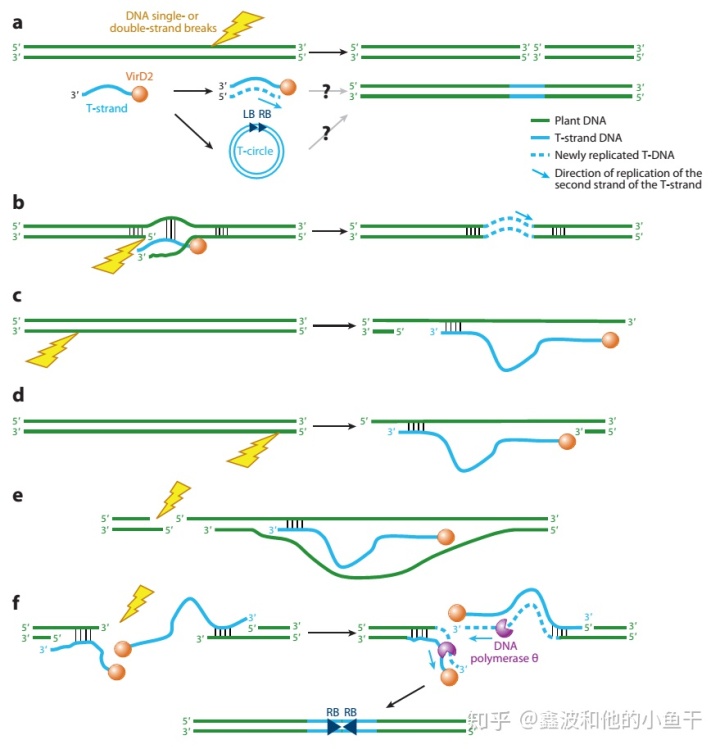
T-DNA以单链形式进入植物细胞,该单链分子的5'末端被VirD2蛋白保护,VirE2蛋白覆盖并保护T链,但可能无法保护T链的3'末端。在T-DNA整合之前或整合期间,必须将VirE2从T链上剥离,使T链序列不被掩盖,这时候它们便对核酸酶的降解变得敏感。
通常,T-DNA的整合过程可以分为两种情况:1. T链直接侵入基因组DNA,通过T链与植物染色体双链DNA靶位点的微同源性侵入;2. T链转化为DNA双链,然后通过连接进入植物基因组。
VirE2蛋白的去除还将为T链序列与互补植物靶DNA序列的基于同源性(或微同源性)的匹配提供可能。T链的互补配对可能不会恰好发生在T链3'末端,并且在整合的过程中进一步修剪同源位点的T链序列。基于同源性的单链入侵发生后,核酸酶降解或T链3'末端的修整将导致整合的T-DNA分子在3'末端(LB区)附近缺少序列。因此,基于同源性的单链入侵整合模型最初获得了科学家的青睐。但是,单链入侵模型不能轻易解释经常发现的头对头(RB-RB)或尾对尾(LB-LB)整合的T-DNA二聚体。
T链在核中形成双链T-DNA分子似乎是整合到植物基因组双链断裂处的有利机制。农杆菌传递的T链可能通过细胞核中的寡核苷酸转化为双链形式,由于寡核苷酸引物可能在内部位置与T链结合,因此会导致LB附近的T-DNA序列缺失。由这些引物的复制性延伸而合成的双链DNA将缺少T链的3'末端,而悬挂在外的单链末端将被核酸外切酶去除。
T链也可以转化为双链环状DNA分子。这些T-DNA环在农杆菌介导的转化过程中起什么作用尚不清楚。有学者推测它们可能是不整合的末端中间体,也有可能是整合的底物。
DNA聚合酶θ介导的T-DNA整合模型认为,DNA聚合酶θ对于T-DNA的整合至关重要。DNA聚合酶θ在植物基因组DNA双链断裂处的3'末端或附近,通过微同源性捕获T链的3'末端。在每个断裂的植物DNA末端捕获两个T-DNA可能会产生头对头T-DNA二聚体,尽管尾对尾或头对尾二聚体的产生没有那么简单。
这些整合方式或途径不一定是互斥的,可能同时存在。
要点小结
- 农杆菌介导的植物遗传转化涉及许多细菌和植物编码的蛋白质。
- T-DNA的整合和转基因表达是农杆菌介导稳定转化的最后步骤。
- T-DNA在植物基因组中的整合是随机的,基因座可能影响转基因表达。
- T-DNA整合经常伴随着缺失、插入、DNA填充和染色体易位。
- T-DNA整合类似于DNA修复过程,但不需要这些修复过程涉及的蛋白。
- DNA聚合酶θ是拟南芥中T-DNA整合所必需的。
参考文献
- Gelvin S B . Finding a way to the nucleus [J]. Current Opinion in Microbiology, 2010, 13(1):53-58.
- Gelvin S B . Plant Proteins Involved inAgrobacterium-Mediated Genetic Transformation [J]. Annual Review of Phytopathology, 2010, 48(1):45-68.
- Gelvin S B . Integration of Agrobacterium T-DNA into the Plant Genome [J]. Annual Review of Genetics, 2017, 40(1):19A-19A.







 农杆菌通过T-DNA随机整合到植物基因组中,实现遗传转化。T-DNA切割、转运、进入细胞核,然后通过一系列复杂过程整合,可能涉及DNA修复机制。整合过程可能不精确,可能导致基因组变异。DNA聚合酶θ在T-DNA整合中起关键作用。
农杆菌通过T-DNA随机整合到植物基因组中,实现遗传转化。T-DNA切割、转运、进入细胞核,然后通过一系列复杂过程整合,可能涉及DNA修复机制。整合过程可能不精确,可能导致基因组变异。DNA聚合酶θ在T-DNA整合中起关键作用。
















 2万+
2万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








