这个工具整合了目前流行的Primer3软件,再加上
Primer-BLAST的输入
Primer-BLAST界面包括了Primer3和BLAST的功能。提交的界面主要包括三个部分:target template(模板区), the primers(引物区), 和specificity check(特异性验证区)。跟其它的BLAST一样,点击底部的“Advanced parameters”有更多的参数设置。
模板(Template)
在“PCR Template”下面的文本框,输入目标模板的序列,FASTA格式或直接用Accession Number。如果你在这里输入了序列,是用于引物的设计。Primer-BLAST就会根据你输入的序列设计特异性引物,并且在目标数据库(在specificity check区选择)是唯一的。

引物(Primers)
如果你已经设计好了引物,要拿来验证引物的好坏。可以在Primer Parameters区填入你的一条或一对引物。并且选择好验证的目标数据库(在specificity check区选择)。根据需要可设置产物的大小,Tm值等。
特异性(Specificity)
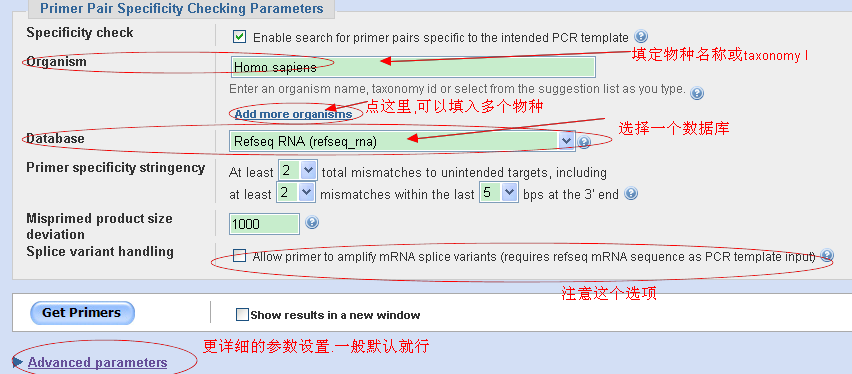
在specificity check区,选择设计引物或验证引物时的目标数据库和物种。这一步是比较重要的。这里提供了4种数据库:RefSeq mRNA, Genome (selected reference assemblies), Genome (all chromosomes), and nr (the standard non-redundant database)。前两个数据库是经过专家注释的数据,这样可以给出更准确的结果。特别是,当你用NCBI的参考序列作为模板和参考序列数据库作为标准来设计引物时,Primer-BLAST可以设计出只扩增某一特定剪接变异体基因的特异引物。selected reference assemblies 包括以下的物种: human, chimpanzee, mouse, rat, cow, dog, chicken, zebrafish, fruit fly, honeybee, Arabidopsis, 和 rice。Nr数据库覆盖NCBI所有的物种。
实例分析
用人尿嘧啶DNA糖基化酶(uracil-DNA glycosylase genes, UNG, GeneID: 7374)的两个转录本序列作为一个例子来分析。UNG1的序列长一点(NM_003362),UNG2的序列短一点(NM_080911,注:拿这两个基因的序列ClustalW一下就可以了)。这里用UNG2的序列设计引物,选择RefSeq mRNA database,物种是Human,其它默认。结果如下图A-B所示,设计的引物只能扩增出UNG2。看上面的图,把“Allow primer to amplify mRNA splice variants”这个选项给勾上,出现的结果如下图-C所示,新的引物也可以扩增出UNG1(注:我试了一下,不能得到预期的结果,可能参数没设对)。
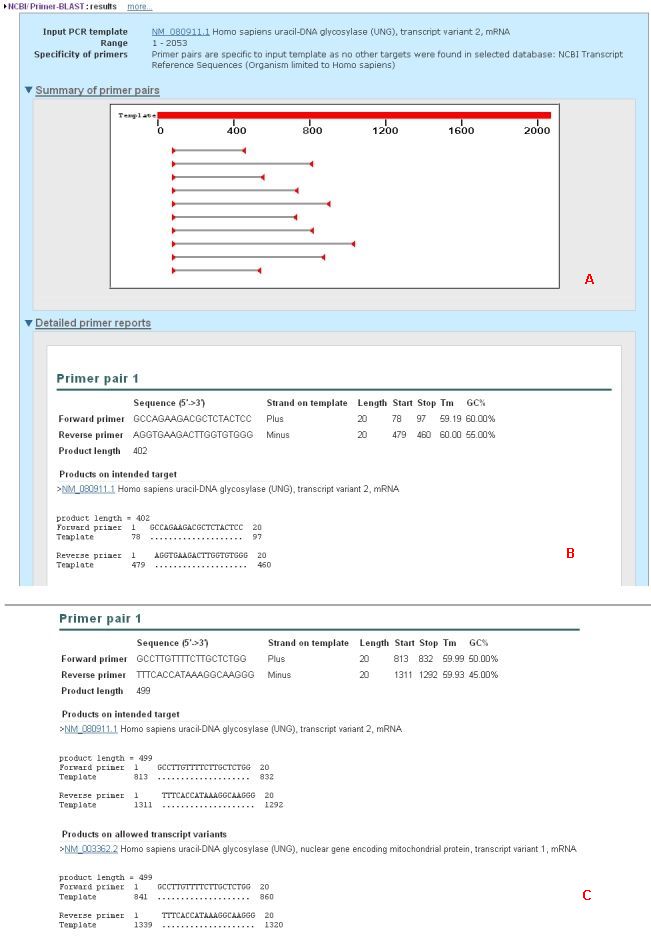
Figure. Primer-BLAST results for UNG transcript variant 2. The NCBI Reference sequence NM_080911 was used as a template. Top panel: Primers specific to the single splice variant are reported by default with the mRNA RefSeq database limited to human sequences. Bottom panel: Primers that amplify both splice variants are found with the option to allow splice variants.(点击看大图)
一些Tips
1,在任何时候都要优先使用参考序列的Gi号或Accession 号(尽量不要Fasta格式的序列)。另外,确保你的序列是最新版本的(在填Accession Number时后面不加版本号就会自动拿最新的序列)
2,就算你对整个序列的某部分感兴趣(如某条染色体上的某个区域),你也应该优化使用Gi号或Accession 号(Primer-BLAST有参数可以设置设计引物的范围,”Form-To”,如上面的第一幅图所示)。因为用Gi号或Accession 号,NCBI会自动读取该序列的一些注释数据,对引物的设计更加有利。
3,尽量使用没有冗除的数据库(如refseq_rna 或 genome database),nr数据库包括了太多的冗除的序列,会干扰引物的设计。
4,请指定一个或几个PCR扩增的目标物种。如果不指定在所有的物种搜索,将会使程序变得很慢,引物的结果也会受其它不相关的物种影响。
参考文献
1. Steve Rozen and Helen J. Skaletsky (2000) Primer3 on the WWW for general users and for biologist programmers. In: Krawetz S, Misener S (eds)







 介绍了Primer-BLAST工具的使用方法,包括模板输入、引物设计与验证及特异性检查等功能。通过实例展示了如何设计针对特定剪接变异体的特异性引物。
介绍了Primer-BLAST工具的使用方法,包括模板输入、引物设计与验证及特异性检查等功能。通过实例展示了如何设计针对特定剪接变异体的特异性引物。
















 1078
1078

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








