摘自大佬博客
https://www.cnblogs.com/TheRoadToTheGold/p/6290732.html
给出n个单词和m个询问
1.查询某个前缀是否出现过
2.查询某个单词是否出现过
3.查询前缀出现的次数
例如:
cat,cash,app,apple,aply,ok 建一颗字典树
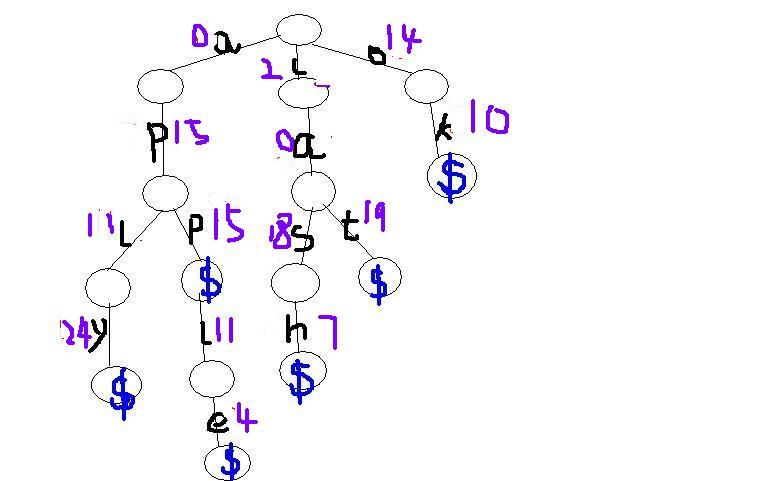
这里有两种编号:
1.相对整棵树而言(根据单词insert顺序 dfs序)
2.相对于每个节点而言
也就是说每个节点其实都开了26条分支
k=trie(i,j): 表示i号节点第j号分支的值为k
sum[i]:记录i节点被多少个单词经过(且记录的是后一个节点的位置!)
建树,插入函数
int tot=0;//记录1类编号
void insert(char *s){
len=strlen(s);
root=0;
For(i,0,len-1){
int id=s[i]-'a';
if(!trie[root][id])trie[root][id]=++tot;
sum[trie[root][id]]++; //注意是在后一个点位置+1
root=trie[root][id];
}
vis[root]=1;
}
查询前缀或者单词是否出现
//查单词要多用个vis[]标记下结束点
bool find(char *s){
int rt=0;
For(i,0,strlen(s)-1){
int id=s[i]-'a';
if(!trie[rt][id])return false;
rt=trie[rt][id];
}
//if(vis[rt])return true;//查单词
return true;//查前缀
}
查询前缀出现次数
int search(char *s){
int rt=0;
For(i,0,strlen(s)-1){
int id=s[i]-'a';
if(!trie[rt][id])return 0;
rt=trie[rt][id];
}
return sum[rt];
}























 1265
1265

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








