Problem UVA1533-Moving Pegs
Accept:106 Submit:375
Time Limit: 3000 mSec
 Problem Description
Problem Description
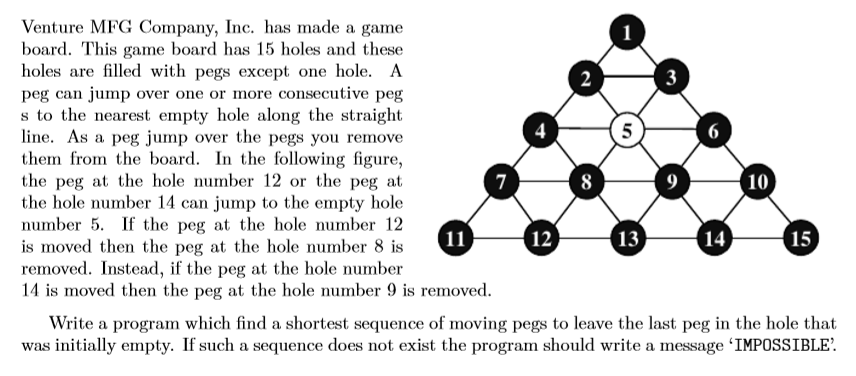
 Input
Input
The input consists of T test cases. The number of test cases (T) is given in the first line of the input file. Each test case is a single integer which means an empty hole number.
 Output
Output
For each test case, the first line of the output file contains an integer which is the number of jumps in a shortest sequence of moving pegs. In the second line of the output file, print a sequence of peg movements. Apegmovementconsistsofapairofintegersseparatedbyaspace. Thefirstintegerofthe pair denotes the hole number of the peg that is moving, and the second integer denotes a destination (empty) hole number.
 Sample Input
Sample Input
 Sample Ouput
Sample Ouput
10
12 5 3 8 15 12 6 13 7 9 1 7 10 8 7 9 11 14 14 5
题解:15个洞,二进制存储状态是比较正的思路。接下来就是水题了,只不过是把矩形地图换成了三角形地图,预处理一个临接表,存一下对于每个点能到哪些点。因为要字典序,因此顺序很重要,稍加分析就知道周围6个位置的大小关系,注意对于15个记录相邻点的数组,一定要统一顺序,除了字典序,还因为有可能要顺着一个方向走几格,这时顺序一致就很方便。
1 #include <bits/stdc++.h> 2 3 using namespace std; 4 5 const int maxn = 15, maxm = 6; 6 const int dir[maxn][maxm] = 7 { 8 {-1,-1,-1,-1, 1, 2}, {-1, 0,-1, 2, 3, 4}, { 0,-1, 1,-1, 4, 5}, {-1, 1,-1, 4, 6, 7}, 9 { 1, 2, 3, 5, 7, 8}, { 2,-1, 4,-1, 8, 9}, {-1, 3,-1, 7,10,11}, { 3, 4, 6, 8,11,12}, 10 { 4, 5, 7, 9,12,13}, { 5,-1, 8,-1,13,14}, {-1, 6,-1,11,-1,-1}, { 6, 7,10,12,-1,-1}, 11 { 7, 8,11,13,-1,-1}, { 8, 9,12,14,-1,-1}, { 9,-1,13,-1,-1,-1} 12 }; 13 14 15 int s; 16 bool vis[1 << maxn]; 17 pair<int, int> path[1 << maxn]; 18 int pre[1 << maxn]; 19 20 struct Node { 21 int sit, time; 22 int pos; 23 Node(int sit = 0, int time = 0, int pos = 0) : 24 sit(sit), time(time), pos(pos) {} 25 }; 26 27 int bfs(int &p) { 28 int cnt = 0; 29 int ori = (1 << maxn) - 1; 30 ori ^= (1 << s); 31 queue<Node> que; 32 que.push(Node(ori, 0, 0)); 33 vis[ori] = true; 34 while (!que.empty()) { 35 Node first = que.front(); 36 que.pop(); 37 if (first.sit == (1 << s)) { 38 p = first.pos; 39 return first.time; 40 } 41 42 int ssit = first.sit; 43 for (int i = 0; i < maxn; i++) { 44 if (!(ssit&(1 << i))) continue; 45 46 for (int j = 0; j < maxm; j++) { 47 int Next = dir[i][j]; 48 if (Next == -1 || !(ssit&(1 << Next))) continue; 49 50 int tmp = ssit ^ (1 << i); 51 while (Next != -1) { 52 if (!(ssit&(1 << Next))) { 53 //printf("%d %d\n",i, Next); 54 tmp ^= (1 << Next); 55 if (!vis[tmp]) { 56 Node temp(tmp, first.time + 1, ++cnt); 57 pre[cnt] = first.pos; 58 path[cnt] = make_pair(i, Next); 59 que.push(temp); 60 vis[tmp] = true; 61 } 62 break; 63 } 64 tmp ^= (1 << Next); 65 Next = dir[Next][j]; 66 } 67 } 68 } 69 } 70 return -1; 71 } 72 73 void output(int pos) { 74 if (!pre[pos]) { 75 printf("%d %d", path[pos].first + 1, path[pos].second + 1); 76 return; 77 } 78 output(pre[pos]); 79 printf(" %d %d", path[pos].first + 1, path[pos].second + 1); 80 } 81 82 int main() 83 { 84 int iCase; 85 scanf("%d", &iCase); 86 while (iCase--) { 87 scanf("%d", &s); 88 s--; 89 memset(vis, false, sizeof(vis)); 90 memset(pre, -1, sizeof(pre)); 91 int pos; 92 int ans = bfs(pos); 93 if (ans == -1) { 94 printf("IMPOSSIBLE\n"); 95 } 96 else { 97 printf("%d\n", ans); 98 output(pos); 99 printf("\n"); 100 } 101 } 102 return 0; 103 }




 本文详细解析了ProblemUVA1533-MovingPegs问题,通过二进制存储状态的方法,使用BFS算法在三角形地图上寻找最短的跳棋移动路径。代码中包含了一个预处理的临接表,用于记录每个点能够到达的点,以及具体的实现细节。
本文详细解析了ProblemUVA1533-MovingPegs问题,通过二进制存储状态的方法,使用BFS算法在三角形地图上寻找最短的跳棋移动路径。代码中包含了一个预处理的临接表,用于记录每个点能够到达的点,以及具体的实现细节。
















 16
16

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








