文章题目:Neural Architecture Search for Gliomas Segmentation on Multimodal Magnetic Resonance Imaging
链接:link https://arxiv.org/pdf/2005.06338
论文内容
论文阅读笔记,帮助记忆的同时,方便和大家讨论。因能力有限,可能有些地方理解的不到位,如有谬误,请及时指正。
近年来,随着深度学习的发展,医学影像和AI的结合也在飞速进步。胶质瘤作为脑部的常见疾病,MRI是其通用的诊断途径。常规的操作是采用Unet训练网络,本文提出了NAS在胶质瘤中的应用,分别搜索上采样和下采样的结构,在BraTS 2019数据集上进行了大量对比实验,其结果表明NAS不仅可以减轻块结构的制造压力,而且具有良好的可行性和扩展性。
BraTS 2019中的训练集( training set) 有200多个病例,每个病例有四个模态:T1、T2、flair、t1ce——T1看结构,T2看病变,t1ce显示瘤的位置。每个病例需要分割三个部分:whole tumor(WT), enhance tumor(ET), and tumor core(TC)。
为了模型表现稳定,作者进行的均一化处理,除了均值方差手段外,还加了一个非线性函数:
作者从xyz三个方向,立体的对数据进行复合采样,并在Unet的基础上分别搜索Up-Sample Cell和Down-Sample Cell,将他们以类似于U-Net的方式堆砌起来:
每个Cell都有三个Node:
其搜索空间:上下采样dilated convolution、depthwise separable convolution、shaped conv,se conv;下采样多了max pool 和 avg pool;正则操作有conv和identity。
作者轮流训练混合参数和网络的内核参数,并额外设计了特定的loss函数。NAS搜出的结构最终在BraTS 2019数据集中的表现和人类设计的网络性能相当:
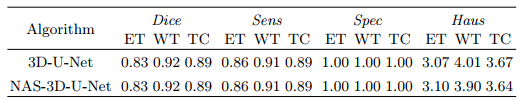










 本文介绍了一种利用神经架构搜索(NAS)优化深度学习模型,用于胶质瘤多模态磁共振成像(MRI)分割的方法。研究者在BraTS2019数据集上,通过NAS搜索上采样和下采样的最优结构,提高了模型的稳定性和性能,与人工设计的网络相媲美。
本文介绍了一种利用神经架构搜索(NAS)优化深度学习模型,用于胶质瘤多模态磁共振成像(MRI)分割的方法。研究者在BraTS2019数据集上,通过NAS搜索上采样和下采样的最优结构,提高了模型的稳定性和性能,与人工设计的网络相媲美。
















 2587
2587

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








