MIT6_0002F16_ProblemSet4
实验内容:
本次实验旨在设计并实现一个细菌种群动态的随机模拟模型,并根据模拟结果得出各种治疗方案如何影响细菌的传播的结论。
实验流程:
阅读实验pdf,可知实验具体要求,按照要求一步一步解决。
Problem 1: Implementing a Simple Simulation
该部分为最简易的细菌模拟,不添加抗生素,简单模拟细菌的种群增长情况。
主要任务是填写 SimpleBacteria类和Patient类。SimpleBacteria 类表示单个细菌的状态。Patient维护与病人相关的细菌种群的状态。
代码如下:(根据提示较易完成)
class SimpleBacteria(object):
def __init__(self, birth_prob, death_prob):
#max出生概率
self.birth_prob=birth_prob
#max死亡概率
self.death_prob=death_prob
def is_killed(self):
#利用随机的办法判断是否死亡
return random.random() <= self.death_prob
def reproduce(self, pop_density):
#利用随机的办法判断是否将繁殖,繁殖可能性为self.birth_prob * (1 - pop_density).
if random.random() < (self.birth_prob * (1 - pop_density)):
return SimpleBacteria(self.birth_prob, self.death_prob)
else:
raise NoChildException
class Patient(object):
def __init__(self, bacteria, max_pop):
#病人身上的细菌
self.bacteria=bacteria
#最大的细菌种群大小
self.max_pop=max_pop
def get_total_pop(self):
#返回细菌数目
return len(self.bacteria)
def update(self):
#根据提示即可完成
#判断一下细菌是否死亡或者产生新的细菌
surviving_bacteria = []
for bacterium in self.bacteria:
if not bacterium.is_killed():
surviving_bacteria.append(bacterium)
#计算当前种群密度,用细菌数/菌落数
pop_density = len(surviving_bacteria) / self.max_pop
#判断存活细菌是否将产生新细菌
child_bacteria = []
for bacterium in surviving_bacteria:
try:
child_bacteria.append(bacterium.reproduce(pop_density))
except NoChildException:
pass
self.bacteria = surviving_bacteria + child_bacteria
return len(self.bacteria)
Problem 2: Running and Analyzing a Simple Simulation (No Antibiotic Treatment)
模拟没有抗生素的情况下细菌的生长情况。需要完成两个函数:simulation_without_antibiotic 以及calc_pop_avg,calc_pop_avg用来求第n个时间点的细菌平均数,simulation_without_antibiotic 用来模拟细菌的生长情况。完成代码后,运行populations = simulation_without_antibiotic(100, 1000, 0.1, 0.025, 50) 可得具体生长图像。populations 是一个二维list,用于存取每个菌落与时间相关的性质,每个list元素中的元素表示一个菌落随时间step的变化
代码:
#返回第n步的细菌平均数
def calc_pop_avg(populations, n):
data=[]
#取每个菌落的第n步的菌落数
for i in range(len(populations)):
data.append(populations[i][n])
#返回其平均值即可
return np.mean(data)
def simulation_without_antibiotic(num_bacteria,max_pop,birth_prob,death_prob,num_trials):
#题目的图中steps达到了300,故这里选取300
time_steps = 300
populations = []
#模拟num_trials个菌落的生存情况,并将具体数据记录
for trial_i in range(num_trials):
bacteria = [SimpleBacteria(birth_prob,death_prob) for i in range(num_bacteria)]
patient = Patient(bacteria, max_pop)
trial_data = [patient.get_total_pop()]
for time_step in range(time_steps-1):
trial_data.append(patient.update())
#模拟每个菌落n步后,将该list加入到population中
populations.append(trial_data)
# 画图
x_coords = list(range(time_steps))
y_coords = [calc_pop_avg(populations, n) for n in x_coords]
make_one_curve_plot(x_coords, y_coords, "Time Step", "Avg. Population", "Uninhibited Bacterial Growth")
#返回该二维list
return populations
运行结果:符合预期
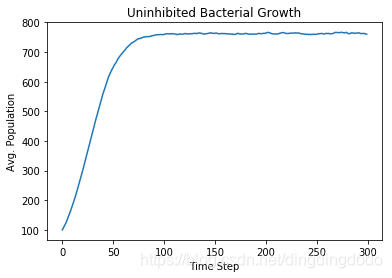
test函数也说明函数正确:
`test_calc_pop_avg (__main__.ps4_calc) ... ok`
Problem 3: Calculating a Confidence Interval
这一部分我们需要以问题2模拟中收集到的数据为样本,构建一个95%置信区间,估计在某个时间步长(零索引)下的平均细菌数量。根据数学公式提示较易完成:
def calc_pop_std(populations, t):
#将第t步的数据记录
data = [populations[i][t] for i in range(len(populations))]
#调用库返回std
return np.std(data)
def calc_95_ci(populations, t):
#平均数
mean = calc_pop_avg
#根据公式求1.96*SEM
width = calc_pop_std(populations, t)/(len(populations)**0.5) * 1.96
#返回mean+-SEM
return (mean, width)
运行test函数:
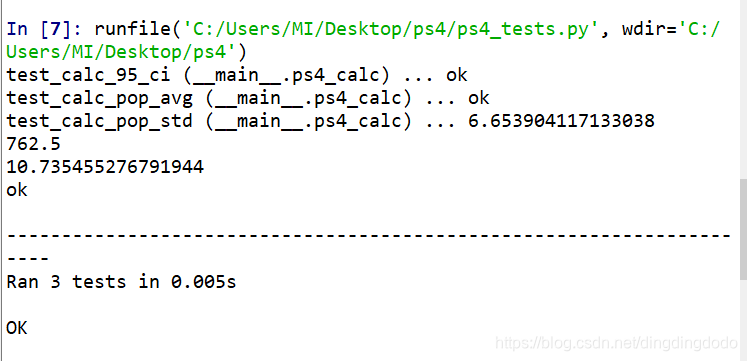
利用上述给的population,运行calc_95_ci(populations,299)并打印结果,如下:
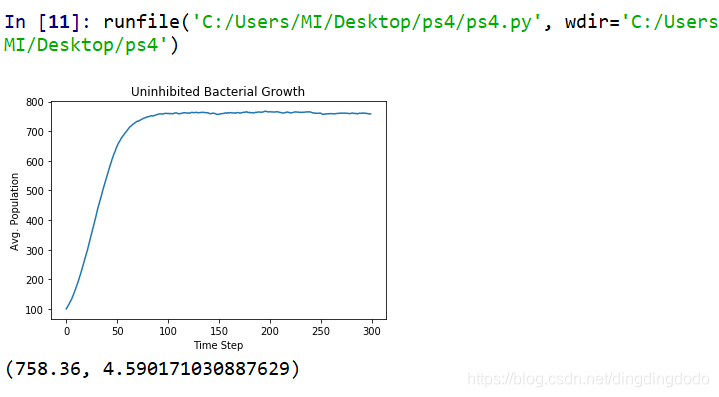
Problem 4: Implementing a Simulation with an Antibiotic
这部分填写ResistantBacteria 抗药性细菌类,是SimpleBacteria 的一个子类。
耐药性细菌有特殊的性质,对抗生素耐药的细菌死亡率固定,没抗生素耐药的细菌死亡率为death_prob/4。
class ResistantBacteria(SimpleBacteria):
def __init__(self, birth_prob, death_prob, resistant, mut_prob):
#调用父类初始函数
SimpleBacteria.__init__(self,birth_prob,death_prob)
#细菌突变概率,后代获得抗药性的概率
self.mut_prob = mut_prob
#是否是抗药性
self.resistant=resistant
def get_resistant(self):
return self.resistant
def is_killed(self):
#如果是抗药性的话,以故定概率死亡
if self.get_resistant():
return random.random() < self.death_prob
#否则以death_prob/4概率死亡
else:
return random.random() < self.death_prob/4
def reproduce(self, pop_density):
#判断是否可以繁殖
if random.random() < (self.birth_prob * (1 - pop_density)):
#父代有抗药性,则子代具有抗药性
if self.get_resistant():
return ResistantBacteria(self.birth_prob, self.death_prob,
self.resistant, self.mut_prob)
#父代有抗药性,子代有可能有抗药性
elif random.random() < (self.mut_prob * (1 - pop_density)):
return ResistantBacteria(self.birth_prob, self.death_prob,
True, self.mut_prob)
#子代没有抗药性
else:
return ResistantBacteria(self.birth_prob, self.death_prob,
False, self.mut_prob)
TreatedPatient 类,是Patient的子类,表示接受抗生素的病人。
class TreatedPatient(Patient):
def __init__(self, bacteria, max_pop):
#调用父类构造函数
Patient.__init__(self, bacteria, max_pop)
#默认为未注射抗生素
self.on_antibiotic = False
def set_on_antibiotic(self):
#注射抗生素
self.on_antibiotic = True
def get_resist_pop(self):
#返回细胞数
counter = 0
for bacterium in self.bacteria:
if bacterium.get_resistant():
counter += 1
return counter
def update(self):
#判断一下细菌是否死亡
surviving_bacteria = []
for bacterium in self.bacteria:
if not bacterium.is_killed():
surviving_bacteria.append(bacterium)
#判断在有抗生素情况下细菌生存情况
if self.on_antibiotic:
surviving_bacteria = [bacterium for bacterium in
surviving_bacteria if bacterium.get_resistant()]
#细菌繁殖情况
pop_density = len(surviving_bacteria) / self.max_pop
child_bacteria = []
for bacterium in surviving_bacteria:
child_bacteria.append(bacterium.reproduce(pop_density))
#总细菌数目=没死的细菌加上繁殖的细菌
self.bacteria = surviving_bacteria + child_bacteria
return len(self.bacteria)
Problem 5: Running and Analyzing a Simulation with an Antibiotic
该部分用于模拟研究耐药细菌以及抗生素治疗的效果。
def simulation_with_antibiotic(num_bacteria,max_pop,birth_prob,death_prob,
resistant, mut_prob,num_trials):
#根据注释确定需要的变量
time_steps_before_antibiotic = 150
time_steps_after_antibiotic = 250
populations = []
resistant_pop = []
#每过一个时间片
for trial_i in range(num_trials):
#实例化相应的细菌以及病人
bacteria = [ResistantBacteria(birth_prob, death_prob, resistant,
mut_prob) for i in range(num_bacteria)]
patient = TreatedPatient(bacteria, max_pop)
trial_data = [patient.get_total_pop()]
trial_data_resist = [patient.get_resist_pop()]
# 加入抗生素治疗
for time_step in range(time_steps_before_antibiotic - 1):
trial_data.append(patient.update())
trial_data_resist.append(patient.get_resist_pop())
patient.set_on_antibiotic()
for time_step in range(time_steps_after_antibiotic):
trial_data.append(patient.update())
trial_data_resist.append(patient.get_resist_pop())
populations.append(trial_data)
resistant_pop.append(trial_data_resist)
# 作图:
x_coords=list(range(time_steps_before_antibiotic + time_steps_after_antibiotic))
y_coords1 = [calc_pop_avg(populations, n) for n in x_coords]
y_coords2 = [calc_pop_avg(resistant_pop, n) for n in x_coords]
make_two_curve_plot(x_coords, y_coords1, y_coords2,
"Total",
"Resistant",
"Timestep",
"Avg. Population",
"Antibiotic Effect on Resistant Bacterial Growth")
return (populations, resistant_pop)
运行结果: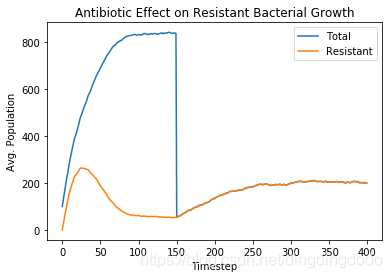
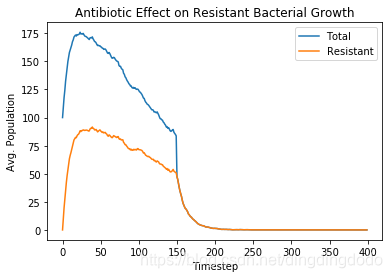
运行程序找对应的置信区间:
4个置信区间:
(196.8, 7.437677056716028)
(196.8, 7.437677056716028)
(0.0, 0.0)
(0.0, 0.0)
Problem 6 Write-up
A和B两种模拟只有一个区别,便是细菌的繁殖率,因此我们可以认为当繁殖率较低时在抗生素条件下很难维持增长,如果繁殖率较高,则随着突变的发生,产生突变的细菌也就越来越多,这样最终导致抗药菌很多,从而实现了增长。
-
What happens to the total population before introducing the antibiotic?
引入抗生素之前,细菌数量增长非常快。
-
What happens to the resistant bacteria population before introducing the antibiotic?
耐药菌数目也是增长迅速,占总菌种一定比例。
-
What happens to the total population after introducing the antibiotic?
A情景引入抗生素后细菌数量迅速下降,但之后恢复。B场景中下降至0
-
What happens to the resistant bacteria population after introducing the antibiotic?
在A情景下引入抗生素菌下降后又开始上升,而B情景则下降至0





 这篇博客详细介绍了MIT6_0002F16_ProblemSet4实验,涉及设计并实现一个细菌种群动态模拟,包括无抗生素和有抗生素的情况。实验通过模拟分析了不同治疗方案对细菌传播的影响,观察了抗生素使用前后细菌数量和耐药菌群体的变化。
这篇博客详细介绍了MIT6_0002F16_ProblemSet4实验,涉及设计并实现一个细菌种群动态模拟,包括无抗生素和有抗生素的情况。实验通过模拟分析了不同治疗方案对细菌传播的影响,观察了抗生素使用前后细菌数量和耐药菌群体的变化。
















 2405
2405

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








