
摘要
近年来,物理学家、化学家和地质学家纷纷宣称发现了新的进化原理,认为这些原理在一个多世纪以来被进化生物学家所忽视,甚至将这些新思想提升至与物理学基本定律同等的地位。然而,这些主张显然反映出对进化生物学数十年来所建立的理论框架缺乏基本了解。作者们所提出的数值指标在概念和定量分析上均存在严重问题,以至于失去实际意义,甚至未能区分突变与选择这样基础的概念。此外,这些“新定律”的倡导者还基于一个站不住脚的前提——即自然选择始终推动着有机体复杂性的增长。事实上,这一观点不仅缺乏实证支持,反而有大量证据指向相反的方向。进化生物学一贯欢迎来自其他学科的思想交流,但这并不意味着该领域应容忍那些在其他科学领域中无法被接受的未经证实的猜想。
关键词:进化、复杂性、选择、突变、遗传漂变

来源:集智俱乐部
作者:Michael Lynch
译者:赵思怡
审校:周莉

论文题目:Complexity myths and the misappropriation of evolutionary theory
论文地址:https://www.pnas.org/doi/abs/10.1073/pnas.2425772122
论文来源:PNAS
在过去的一个世纪中,生物科学取得了巨大进展,而在理论发展方面,没有任何一个分支学科能与进化生物学的成就相媲美。在遗传学基本原理的指导下,结合对适应性的广泛研究兴趣、对基因及基因组分子结构的深入理解,以及将随机过程纳入种群遗传学所带来的理论深化,进化理论如今已根深蒂固。如今,任何关于生物多样化的假说,若无法证明其与基本的种群遗传学原理相一致,都不应被认真对待。那种科学家仅引用达尔文、并简单地将一切现象归因于自然选择的时代,理应已经成为过去。
然而,我们如今正处在一个截然不同的时代。成功的政治家和夸饰的传教士频繁炮制虚假叙述——即所谓的“另类事实”——并通过反复传播,使那些缺乏判断力的人相信这些言论是绝对真理。这种策略与一些圈外人士试图重新定义进化理论的做法惊人地相似:他们往往宣称该领域仍处于一种“可悲的无知”状态,却几乎从不真正了解这一学科的核心内容。
“智能设计”论的支持者坚持认为,分子生物学的复杂程度之高,超出了地球自然进化过程所能解释的范畴。而一小部分声音响亮的倡导者则持续鼓吹所谓扩展进化综合(extended evolutionary synthesis),声称只要聚集起足够数量的追随者,一场科学革命便会随之而来[7,8,9]。但实际上,他们所指出的几乎所有“被忽视”的观点,早已在先前的研究中得到了充分探讨[6]。
不止一位物理学家曾断言,生物学本质上不过是物理学的一个分支。然而到了2023年,一小群物理学家、化学家和地质学家采取了更为激进的姿态,试图将进化科学从所谓的“混乱”中“拯救”出来,并通过提出一系列据称具有宏大统一潜力的新理论和“定律”,来实现这一目标。
当然,哲学中有一个专门的研究领域致力于界定一个观点何时应被视为“理论”或“定律”,但这并非本文的关注重点。关键问题是:与其他科学领域不同,进化生物学似乎缺乏应有的规范与边界。没有人会认真对待一位研究有机体的生物学家宣称,生物化学家长期以来误解了分子机制,并提出需要一种完全忽视氢键重要性的新框架;同样地,如果有人声称分子不存在背景热运动,或者原子理论本身存在根本缺陷,也绝不会被物理学家所接受。再比如,若有人建议地质学家应放弃均变原理以及对放射性衰变测年方法的依赖,这类主张也会遭到质疑和否定。然而在进化生物学领域,一些人却能够在高曝光度的平台上轻松提出荒诞不经的观点——不仅无视已有研究成果,甚至在这些主张明显站不住脚的情况下仍能获得关注与传播。下文中将重点分析两个典型代表作:Sharma 等人[10]和 Wong 等人[11],当然类似的例子还有很多[12]。

组装理论
Sharma 等人声称提出了一种全新的理论,并引入了一个单一指标,能够以前人数十年研究所未能实现的方式,解释并量化自然选择与进化过程。然而,在此过程中,他们显然对进化生物学领域缺乏基本了解,或未表现出说明当前理论究竟存在哪些问题的意愿。正如其他研究者所指出的那样[13],该论文的观点曾伴随着大量自我宣传,但这并非本文关注的重点。真正值得关注的是,这一理论在科学上存在诸多严重缺陷。下文将仅指出其中三个关键问题。
作者提出的理论核心是一个复合的组装度量(assembly measure),其公式如下:

其中:
N:样本中分子种类数量
ai:第i类分子的组装指数(即从基本构件出发组装出该对象所需的最少步骤数)
ni:样本中第i类分子的数量
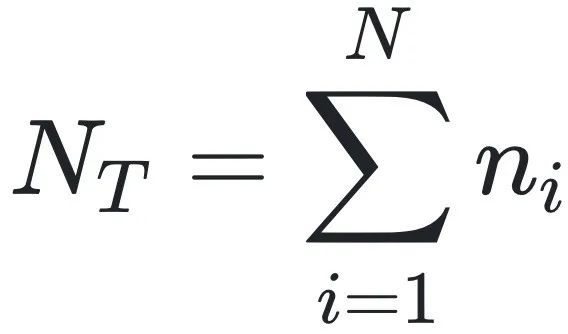 :样本中所有分子的总数
:样本中所有分子的总数
该指标并非基于第一性原理构建,而是源于作者对于“更复杂分子如何组装”的主观直觉。要使用这一分析方法,需要借助质谱等技术获取数据,并明确构成复杂结构的基本构件类型(例如原子或氨基酸)。理论上,该指标或许可用于分析来自外太空或远古岩石中的分子集合,从而推测生命存在的迹象——这是天体生物学中一个极具挑战性的问题。为此,作者试图设定一个阈值,以区分生物系统与非生物系统,并在多篇前期论文中推广了这些观点[14]。
一些问题从一开始就显而易见。第一个问题,为什么在分子数量项中要减去1?作者解释说,这是为了排除某些仅出现一次的分子种类可能由随机事件产生、因而无法代表真实复杂性的干扰因素。然而,任何对离群值的处理都与样本大小密切相关——更大的样本更可能包含更多“异常”类型。因此,减去1是否合理,并没有明确依据;即使将其替换为其他数值x,也缺乏一个客观的选择标准。第二问题,为什么要用最小组装步骤数进行指数加权?作者认为,研究者通常并不清楚某一分子实际的构建路径,但可以基于化学知识估计其所需的最少步骤。他们假设,某种组装指数所对应的组合可能性会以“超指数级”增长;但如果真是如此,为何不采用更强的加权方式?作者希望该指标能同时反映两个方面:一是“发现”一个对象的难度(即指数项),二是该对象被发现后“生产”的容易程度(即丰度项)。然而,生物体并非根据合成难易来决定分子的产量,而是通过复杂的调控机制精确控制各个基因和代谢通路产物的数量。因此,以丰度为基础进行加权的理由显得含混不清。
除了这些概念上的模糊之处,公式(1)在定量层面也存在明显缺陷。其中最显著的一点是,不同的ai、ni、N、x 和NT组合可能会得出相同的A值,从而导致该指标的解释高度不确定。例如:
考虑一个包含两种分子类型的样本,每种各有两个拷贝(即N=ni=2, NT=4),且ai=2,此时计算得:
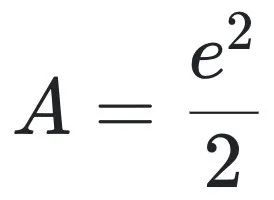
若将样本量翻倍(即N=2, ni=4,NT=8),尽管分子组成未变,结果却变为:
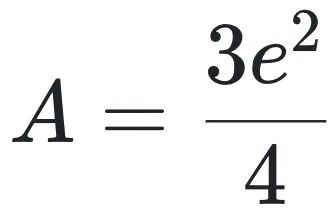
这意味着,在没有任何结构性变化的情况下,复杂度指标却提高了50%。
再看另一个例子:一个样本中仅含一种分子类型,共4个拷贝,其组装指数a=4,计算得:
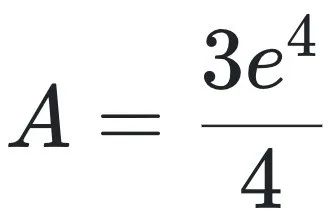
虽然该系统中的分子种类更少,但单个分子的组装步骤更复杂,最终复杂度却比前一系统高出约11倍。这些差异完全是由于公式设定所致,并不能反映真实的生物学复杂性。
此外,如果我们改变公式中两项的权重比例,也会得到完全不同的结果。而且由于分母中包含了NT,使得(ni−1)/NT这一项并非独立衡量某类分子“易于生产”的指标,而是受到整个样本组成的综合影响。关键在于:这一公式的任意性太强,使得单一指标A可以被赋予多种甚至相互矛盾的解释。
不过,这还不是组装理论最大的问题。更为根本的问题在于,作者反复误用“选择”这一术语,未能认识到即便是在最简单的模型中,进化也是突变偏差、自然选择与遗传漂变(genetic drift)共同作用的结果。他们混淆了“生成一个对象的突变过程”与“之后该对象是否被选择保留”这两个本质不同的机制。在进化生物学中,“漂变”指的是由一系列普遍存在的随机因素所产生的集体效应,例如有限的种群规模、家族大小的变异,以及多个突变同时存在时引发的背景干扰。这些因素通过引入“噪声”,影响选择的效率。在过去一个世纪中,这些机制构成了进化理论的核心内容[5,6]。
举一个简单的例子,设有两种对象类型:A和a,其正向与反向转化(或生成)的速率分别为u和ν。我们定义![]() 作为突变偏向于A的程度。在遗传学中,这可以类比为两个等位基因之间的可逆突变过程;而在物理系统中,也可以将其视为两种可互换粒子状态之间的转换。假设A类型处于持续的正向选择作用下,其选择优势为s,那么在长期稳态条件下,a类型的平均频率为:
作为突变偏向于A的程度。在遗传学中,这可以类比为两个等位基因之间的可逆突变过程;而在物理系统中,也可以将其视为两种可互换粒子状态之间的转换。假设A类型处于持续的正向选择作用下,其选择优势为s,那么在长期稳态条件下,a类型的平均频率为:
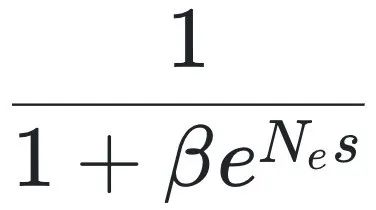
其中,Ne表示有效种群大小,它反映了进化过程中“噪声”的水平——Ne越大,随机漂变的影响越小,自然选择的效率越高[15, 16]。有趣的是,在物理学中的“两态系统”中也存在一个几乎完全相同的方程。只需将背景热运动的影响替代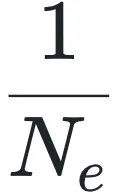 ,便可推导出著名的玻尔兹曼方程(Boltzmann equation)[17, 18]。
,便可推导出著名的玻尔兹曼方程(Boltzmann equation)[17, 18]。
这种公式形式可以扩展至任意数量的状态,包括 Sharma 等人所描述的由多个路径构成的复杂组装网络。然而,即便是这个简单的两态模型,也足以阐明两个关键问题。首先,在“完全不存在选择作用(s=0)的情况下,或当随机漂变的作用远大于选择强度(即Nes≪1)”时,A类型仍将具有一个非零的平衡频率:
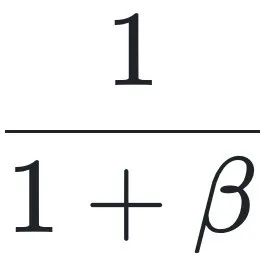
其中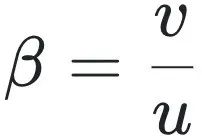 是正向与反向转化速率的比值。换句话说,即使没有选择压力,仅靠突变偏向就足以导致分子类型分布的偏差。这表明,选择并非产生分布不对称性的唯一机制。其次,由于整个过程仅依赖于一个复合参数
是正向与反向转化速率的比值。换句话说,即使没有选择压力,仅靠突变偏向就足以导致分子类型分布的偏差。这表明,选择并非产生分布不对称性的唯一机制。其次,由于整个过程仅依赖于一个复合参数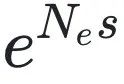 ,因此无法仅凭某一分子类型(如A)的出现频率来判断是否存在选择作用。高频出现的现象可能源于强烈的定向突变,也可能来自选择偏向——若无额外信息(例如生产偏差的具体程度),便无法区分这两种机制。
,因此无法仅凭某一分子类型(如A)的出现频率来判断是否存在选择作用。高频出现的现象可能源于强烈的定向突变,也可能来自选择偏向——若无额外信息(例如生产偏差的具体程度),便无法区分这两种机制。
事实上,进化生物学家(以及物理学家)早已认识到这些问题。要从频率数据中推断选择作用的存在,必须辅以关于突变或生产偏向的定量信息。Sharma 等人混淆了“突变”与“选择”这两个本质不同的概念,并忽略了可逆化学反应的可能性。在缺乏对生产速率进行量化测量的前提下,仅凭组装度量几乎无法提供有关选择作用的任何有效信息。换言之:
分子类型的显著偏向可以在没有选择作用的情况下出现;
即使存在选择作用,只要满足
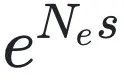 =1,其分布仍可能表现为看似均匀。
=1,其分布仍可能表现为看似均匀。
至于ai这个“组装指数”,它或许能在某些情境下作为衡量分子复杂性的一个参考指标——尽管这一想法并不新颖[13]。但考虑到上述种种理论缺陷,Sharma 等人提出的综合度量A在解释复杂性起源方面,似乎并无实质意义。
复杂性的适应性涌现的幻觉
接下来我们要讨论的是“组装度量是否具有信息性”这一概念的第三个问题,这也与 Wong 等人的观点相关。Sharma 等人隐含地假设:自然选择的主要目标之一是产生更高的复杂性。这种常见观点完全是人类中心主义的产物,并没有任何证据表明自然选择在持续追求更复杂的分子、细胞或生物体。当然,如今的生物确实比“最后的通用祖先”(LUCA)时期更为复杂,但在四十亿年前细胞刚刚出现时,演化只能朝一个方向发展——从无到有。微生物在这整个过程中一直存在,但它们并没有显著增加自身的复杂性。考虑到微生物庞大的种群规模和极短的世代时间,如果复杂性真的具有选择优势,那么显然已经存在足够多的机会去实现基因组、分子和细胞层面的复杂性演化。然而,通向真核生物的复杂形态细胞的起源是一个极为罕见的事件,生命之树的绝大部分仍然是原核生物。地球上目前大约有1030个原核生物个体,比单细胞真核生物高出三个数量级,比后生动物(metazoan)更是高出十个数量级[19]。这一事实显然并不支持“生命注定走向复杂”的说法。

基因组大小和基因结构复杂性的大幅扩展主要局限于后生动物和陆生植物——它们在整个生命之树中只是很小的一部分。而这两个类群恰好经历了有效种群规模的大幅下降[19, 20],正如我们之前所说,这会降低自然选择的效率。即使是在这些谱系中,基因数目的增长也通常仅为原核生物的两到五倍,这意味着自真核生物出现以来,平均每十万年只需新增一个新基因。如后文将进一步讨论的那样,多细胞生物基因组扩增的大部分内容其实来源于有害的可移动遗传元件以及蛋白编码基因中的非编码内含子插入[20]。
人们常认为,更多的重复基因和异源复合物分子有助于提升生物的稳健性(robustness),但这一点同样缺乏实证支持[19]。例如,尽管后生动物和陆生植物在DNA复制与修复机制上更为复杂,它们的有害突变率却显著高于原核生物。尽管真核生物拥有更为复杂的核糖体及其组装机制,它们的翻译速率或准确性并未提高,甚至可以说,其催化速率和酶反应的精确性反而低于原核生物(后者使用结构更简单的同源酶)。真核生物在生物能量方面的表现也弱于原核生物(即生长速率更低)[21, 22],这种差异在多细胞物种中尤为明显[23]。最后值得一提的是,许多生物(尤其是寄生生物,它们占生物多样性的一大部分)往往倾向于进化出简化的基因组,而在后生动物谱系中,许多用于氨基酸和辅因子合成的生物合成通路也已丢失。
生命体并不等同于具备自我复制能力的无生命物体。生命结构与功能的特殊细节,都是在LUCA之前就已经确立的历史偶然事件所遗留下来的遗产,它们决定了所有分子组装与分解的过程。这正是生物学不仅仅是化学或物理学的原因。即便真的存在某种适用于生命系统的组装规则,它也不一定与试管中、沉积物中或气体中的规则一致。已知不同系统发育谱系之间的生物合成机制存在显著差异[19],在另一个适宜生命存在的星球上,这些机制很可能完全不同。
那么,那些存在于所有细胞、尤其在多细胞生物中更为明显的“错误防御机制”和“遗传与代谢网络的扩展”,又该如何解释?这些现象是否真的与Wong等人提出的“功能性信息递增定律”(也称为“复杂性递增定律”[24])相一致?他们将这一所谓的定律赋予了类似于运动、重力、热力学、电学和磁学等物理基本定律的地位。答案似乎是否定的。理论模型和实际数据都表明,在某一特定生物通路中增加一层复杂性,往往会削弱对其他组成部分的选择压力,从而导致各个组件性能下降,整体系统的长期准确性并未提升[25, 26]。此外,一些关于复杂性产生并最终固化的经典观点,也在建构性中性演化理论 (constructive neutral evolution)中得到了体现[27, 28]。这些普遍原则不仅适用于生物学,也适用于许多人类发明,包括计算机架构设计中的某些特征[26]。
功能信息(functional information)到底指什么?Wong 等人并未明确给出定义,但他们显然心中有一个可以量化的概念,因为他们断言这一特征会随着时间增长。他们似乎将“功能”定义为处于选择作用之下的某种特性。从基因组和基因的角度来看——这些是负责构建细胞有机材料的基础分子——我们可以提出几个问题:
首先,所有基因组都会不断产生新的重复基因,这些通常是染色体复制过程中的偶然事件[20]。大多数重复基因要么部分冗余,要么完全冗余,并且通常只是短暂存在。在多细胞真核生物中,重复基因比在微生物中更容易被保留下来,不是因为它们获得了新功能,而是因为它们将原有功能进行了分割(即“亚功能化”,subfunctionalization)。亚功能化并不是因为自然选择主动追求这样的结果——这种结果本身对能量和突变来说都是一种负担——而是由于在有效种群规模较小的谱系中,选择效率有所下降。那么,我们又该如何将基因数量与功能信息联系起来呢?
其次,内含子很可能在最后的普遍共同祖先(LUCA)之前通过自我增殖的方式侵入了真核生物基因组,再次给宿主基因带来了原核生物所没有的能量消耗和诱变代价。尽管内含子序列确实具有一定的功能信息,因为每个内含子(及其邻近的外显子序列)都包含剪接复合体识别所需的关键基序,但没有任何证据表明祖先真核生物中这些基因结构修饰的出现是由选择增加信息含量推动的。相反,情况恰恰相反。
第三,由于遗传密码的冗余性,在某些密码子位置上的核苷酸替换并不会影响最终蛋白质的氨基酸序列。然而,在拥有极大有效种群规模的微生物中,特定位置的核苷酸可以通过提高转录或翻译效率而受到正向选择。那么,当“有效中性”效应在真核生物(尤其是那些Ne极低的多细胞类群)中越来越显著时,是否意味着功能信息正在逐渐丢失?我们又应该在什么时候停止将某个核苷酸位点或某个基因视为具有功能或信息内容?
最后,在他们为数不多的明确例子中,作者提出“核酶的信息含量取决于其大小”,尽管他们随后又声称:“某些功能的丧失仍可能代表所选功能的功能信息增加。”这句话含义不清。我们不妨再以核糖体为例,它本质上是一个核酶。核糖体是一个令人费解的大型分子机器,尤其值得注意的是在各种真核谱系中 rRNA 的显著扩张。这种随意的增长仅出现在 rRNA 中功能区域之外的扩展片段上。
同时,真核生物中核糖体蛋白的数量也有显著增加,参与核糖体生物合成所需的机制更是出现了更为剧烈的扩展[19]。然而,没有任何证据表明这些变化是由正向选择推动的。更合理的解释是:在地球上绝大多数生物(单细胞物种)中,自然选择足够高效,足以防止这类现象的发生。
这份清单还可以继续扩展,涵盖基因组演化的其他方面,例如在多细胞谱系中出现的、携带信息但具负担的寄生性遗传元件(如转座子和逆转座子)的大规模扩增。但关键论点已经阐明。对于敢于提出进化新定律的作者们而言,理应提供一些方法说明和支持数据。科学史上不乏昙花一现的理论风潮,其中偶尔也蕴含着潜在的重要思想(例如来自混沌理论、协同进化、可进化性、分形几何、网络科学和稳健性等领域的概念)。但通常这些理论至少有一个清晰的起点,而功能信息增长定律(Law of Increasing Functional Information)却连“功能”和“信息”的有用定义都没有给出。
生物体不仅仅是被美化的机器或计算机[29],生命系统中额外增加的复杂层次并非在绝对意义上是中性的。更复杂的系统更容易出错,同时构建和维持它们所需的能量成本也更高。只要一个简单的系统能够完成某项任务,自然选择在高效运作时会优先采用这种简单方案——换句话说,生物系统倾向于选择简单且有效的解决方式,除非复杂性带来了明显的优势。这一点与无生命的化学反应大不相同,后者只是按照物理化学规律,被动地沿着能量消耗最小、阻力最小的路径进行。总而言之,现有所有证据都表明:基因组和分子复杂性的扩展,主要局限于极少数谱系(其中包括我们人类),并非适应性过程的结果。相反,这些谱系中出现的细胞复杂性修饰,其实是随机遗传漂变增强、自然选择效率下降所带来的不可避免的副产物。
跨学科科学
一些读者(或许也包括前述论文的作者在内)可能会认为,前文的论述显得过于自信甚至傲慢,并主张应当为不同的观点留出讨论的空间。这一看法本身无疑是合理的,也值得提倡:科学正是在多元观点的碰撞中不断前行的。然而,当研究者忽视甚至拒绝参考已有的知识基础时,科学的发展就可能走向倒退;如果这种态度还伴随着过度自我宣传的行为,那么外界的质疑与批评也就不可避免。正如生物进化本身,科学也是一个“在继承中发生变异”的过程。真正能够颠覆我们对知识体系基本认知的重大突破,往往只在几个世纪这样漫长的时间尺度上才会出现。尽管在过去150年间,人类积累了大量科学成果,科研队伍也在持续壮大,但这类里程碑式发现之间的间隔却变得越来越长[30]。这恰恰符合我们对一个运行良好的科学体系所作的预期。
在进化生物学领域,不仅有容纳的空间,而且确实存在现实的需求,欢迎那些愿意以严肃方式参与该领域的跨学科研究者。事实上,在过去几十年中,进化理论(以及生态学)的一些最重要进展,恰恰来自于物理学家和概率论专家——他们意识到自己领域中的许多数学方法可以用来解决此前难以攻克的问题。地球化学家也开始帮助我们认识到,地球的基本物理结构及其元素组成,是如何对演化过程施加根本性约束的。进化生物学涵盖了生命科学的所有子学科,并高度依赖来自物理学、化学和地质学的知识。达尔文就是最早具有整合视野的生物学家之一。然而,正当跨学科合作日益受到重视之时,上述两篇论文却显得不合时宜:它们表面上聚焦于演化的关键问题,实则很大程度上忽视了生物学本身的内容。
进化生物学一直以一种非常特殊的方式被对待。外行人士,甚至包括在各自领域内极具建树的科学家,提出未经证实甚至离奇的观点,对于进化研究而言并非新鲜事。然而从逻辑上看,若想合理地否定整个学科,那些希望引发范式革命的人理应对他们试图推翻的领域有深入了解。如果你认为以往的研究完全无关,或者必须引入全新的普遍原理,那也可以接受——但负责任的做法是说明原因。任何一门成熟科学的终极目标,都是建立起坚实可靠的理论基础,并通过实证观察加以验证,而其余的工作不过是资料收集。在这方面,进化生物学或许已经比许多其他学科更接近这一理想状态。
参考文献
R. A. Fisher, The Genetical Theory of Natural Selection (Clarendon Press, Oxford, UK, 1930).
J. B. S. Haldane, The Causes of Evolution (Longmans, Green and Co., New York, NY, 1932).
S. Wright, Evolution and the Genetics of Populations, Vol. 1–4 (University of Chicago Press, Chicago, IL, 1969).
M. Kimura, The Neutral Theory of Molecular Evolution (Cambridge University Press, Cambridge, UK, 1983).
B. Charlesworth, D. Charlesworth, Elements of Evolutionary Genetics (W. H. Freeman and Co., New York, NY, 2010).
B. Walsh, M. Lynch, Evolution and Selection of Quantitative Traits (Oxford University Press, Oxford, UK, 2018).
E. Jablonka, M. J. Lamb, Evolution in Four Dimensions (The MIT Press, Cambridge, MA, 2006).
M. Pigliucci, G. B. Müller, Evolution: The Extended Synthesis (The MIT Press, Cambridge, MA, 2010).
N. Feiner, M. Feldman, S. F. Gilbert, K. N. Lala, T. Uller, Evolution Evolving (Princeton University Press, Princeton, NJ, 2024).
A. Sharma et al., Assembly theory explains and quantifies selection and evolution. Nature 622, 321–328 (2023).
M. L. Wong et al., On the roles of function and selection in evolving systems. Proc. Natl. Acad. Sci. U.S.A. 120, e2310223120 (2023).
T. Tlusty, A. Libchaber, Life sets off a cascade of machines. Proc. Natl. Acad. Sci. U.S.A. 122, e2418000122 (2025).
J. Jaeger, Assembly theory: What it does and what it does not do. J. Mol. Evol. 92, 87–92 (2024).
S. M. Marshall, A. R. G. Murray, L. Cronin, A probabilistic framework for identifying biosignatures using pathway complexity. Philos. Trans. R. Soc. A: Math., Phys. Eng. Sci. 375, 20160342 (2017).
W.-H. Li, Models of nearly neutral mutations with particular implications for nonrandom usage of synonymous codons. J. Mol. Evol. 24, 337–345 (1987).
M. Bulmer, The selection-mutation-drift theory of synonymous codon usage. Genetics 129, 897–907 (1991).
G. Sella, A. E. Hirsh, The application of statistical physics to evolutionary biology. Proc. Natl. Acad. Sci. U.S.A. 102, 9541–9546 (2005).
H. P. de Vladar, N. H. Barton, The contribution of statistical physics to evolutionary biology. Trends Ecol. Evol. 26, 424–432 (2011).
M. Lynch, Evolutionary Cell Biology (Oxford University Press, Oxford, UK, 2024).
M. Lynch, The Origins of Genome Architecture (Sinauer Associates, Inc., Sunderland, MA, 2007).
M. Lynch, G. K. Marinov, The bioenergetic costs of a gene. Proc. Natl. Acad. Sci. U.S.A. 112, 15690–15695 (2015).
M. Lynch, G. K. Marinov, Membranes, energetics, and evolution across the prokaryote–eukaryote divide. eLife 6, e20437 (2017).
M. Lynch, The bioenergetic cost of building a metazoan. Proc. Natl. Acad. Sci. U.S.A. 121, e2414742121 (2024).
D. W. McShea, R. N. Brandon, Biology’s First Law: The Tendency for Diversity and Complexity to Increase in Evolutionary Systems (University of Chicago Press, Chicago, IL, 2010).
M. Lynch, Evolutionary layering and the limits to cellular perfection. Proc. Natl. Acad. Sci. U.S.A. 109, 18851–18856 (2012).
S. A. Frank, Robustness and complexity. Cell Syst. 14, 1015–1020 (2023).
S. A. Muñoz-Gómez, G. Bilolikar, J. G. Wideman, K. Geiler-Samerotte, Constructive neutral evolution 20 years later. J. Mol. Evol. 89, 172–182 (2021).
L. Schulz, F. L. Sendker, G. K. Hochberg, Non-adaptive complexity and biochemical function. Curr. Opin. Struct. Biol. 73, 102339 (2022).
P. Ball, How Life Works (University of Chicago Press, Chicago, IL, 2023).
M. Park, E. Leahey, R. J. Funk, Papers and patents are becoming less disruptive over time. Nature 613, 138–144 (2023).
(参考文献可上下滑动查看)
阅读最新前沿科技趋势报告,请访问欧米伽研究所的“未来知识库”
https://wx.zsxq.com/group/454854145828

未来知识库是“欧米伽未来研究所”建立的在线知识库平台,收藏的资料范围包括人工智能、脑科学、互联网、超级智能,数智大脑、能源、军事、经济、人类风险等等领域的前沿进展与未来趋势。目前拥有超过8000篇重要资料。每周更新不少于100篇世界范围最新研究资料。欢迎扫描二维码或访问https://wx.zsxq.com/group/454854145828 进入。





















 7万+
7万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








