生信老学长
“师兄,老师让我先验证基因的作用才让我做实验,怎么解决啊”,看着师妹那渴求的眼神,你却在挠头?别怕,来找生信老学长。
TCGA数据库是做肿瘤绕不开的一个关键数据,自2006年开始至今,它已经包含了33种肿瘤类型和10000多例肿瘤患者的数据,包括转录组、表观修饰、基因组学等多种组学,主要的是包含全面的临床描述和专业的指征数据。以前有很多TCGA介绍的文章,但是很多都言之不清。本文将从数据库介绍和数据下载来为大家介绍这个数据库。
一、数据库简介
我们可以通过https://portal.gdc.cancer.gov/来访问,我们可以看到其包含了几乎所有器官的数据并且展示了总体数据的总结。注意,不要用网页翻译,否则会崩溃。
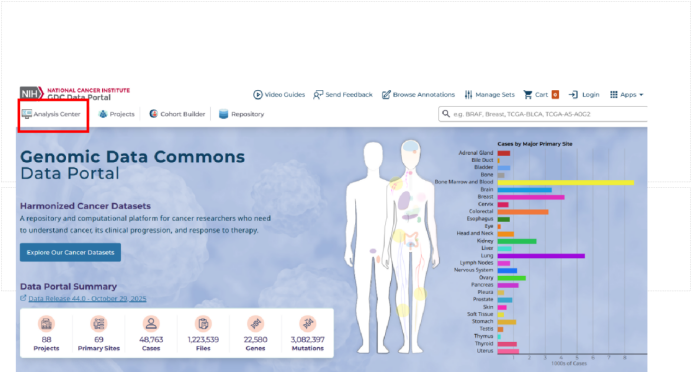
我们直接点击Analysis Center,这里介绍了可以下载的数据类型,不必停留,直接点击projects。
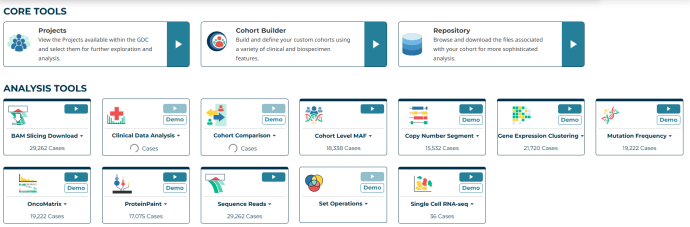
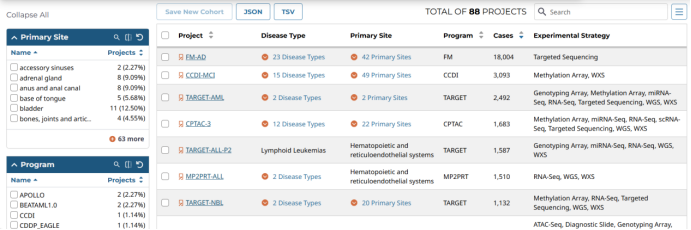
现在我们来到了数据下载和分析的主要页面。我们主要在左边栏选择数据,选择好后在右边栏查看详细数据。
二、数据类型简介
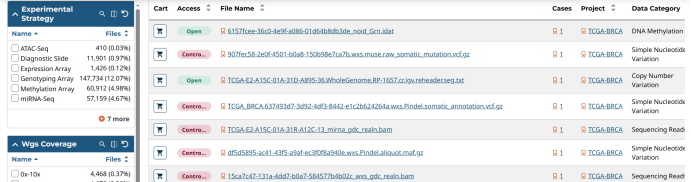
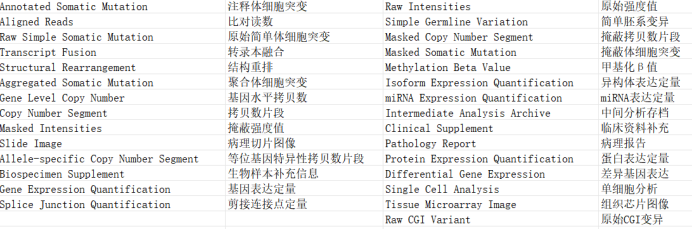
根据左边栏,我们可以发现数据被分为5个方面,分别为Primary Site发病位置、Program项目(可以理解为不同数据库)、Disease Type是疾病类型、Data Category为你要下载数据的类型,例如突变或者转录组。
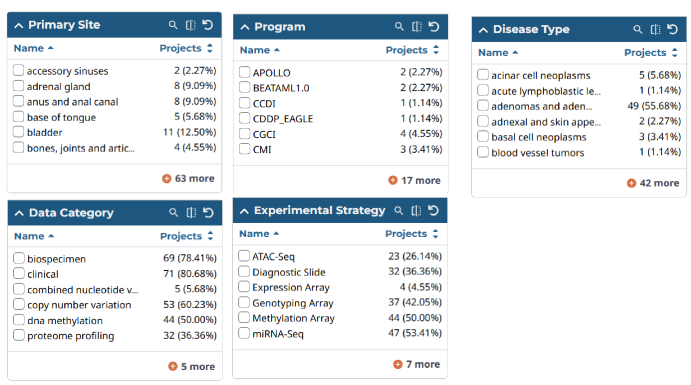
我们设置liver and intrahepatic bile ducts、TCGA、adenomas and adenocarcinomas、Transcriptome Profiling(转录组分析)、RNA-Seq来看看。我们发现最终有两个数据项目被选中,分别是TCGA-CHOL(胆管癌) 和 TCGA-LIHC(肝细胞肝癌)。Experimental Strategy为包含的可下载测序类型。
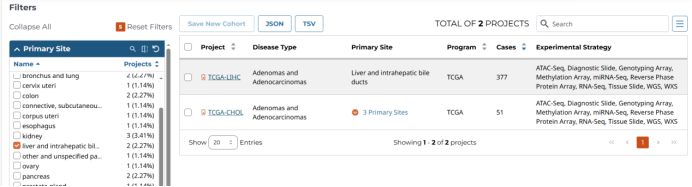
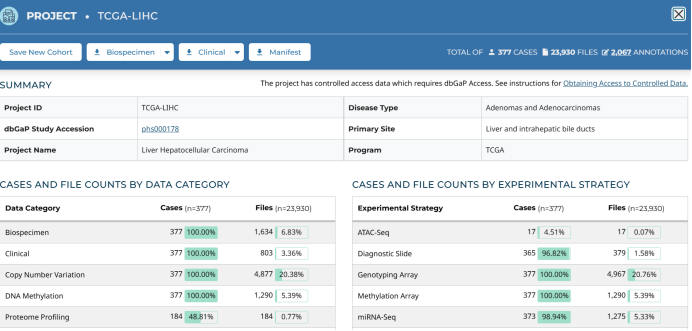
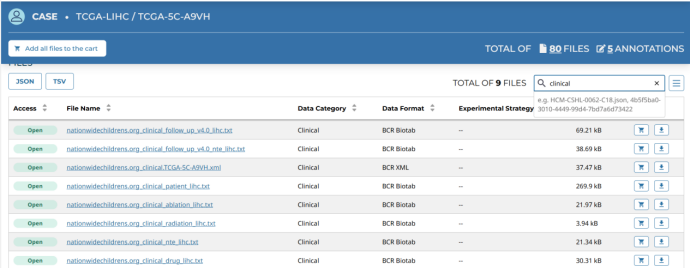
我们点击TCGA-lIHC看看,第一页基本是项目介绍,第二页是每个病人的介绍,这里我们可以点击一个case名来查看,滑到最下边,这里我们主要是可以下载一些隐藏的临床数据,例如乙肝丙肝的感染情况等。直接搜索clinical,例如nationwidechildrens.org_clinical_patient_lihc.txt这个文件,下载后我们可以看到表面抗体和核心抗体的情况。
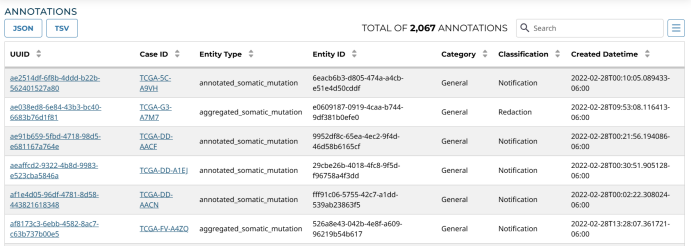
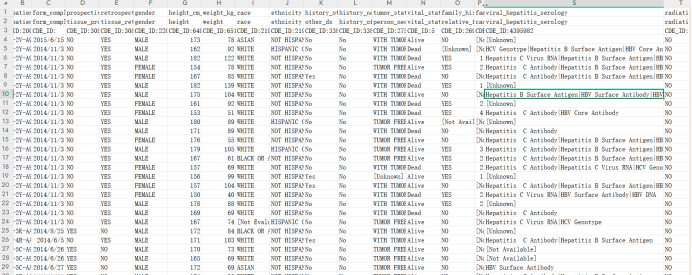
三、临床数据简介
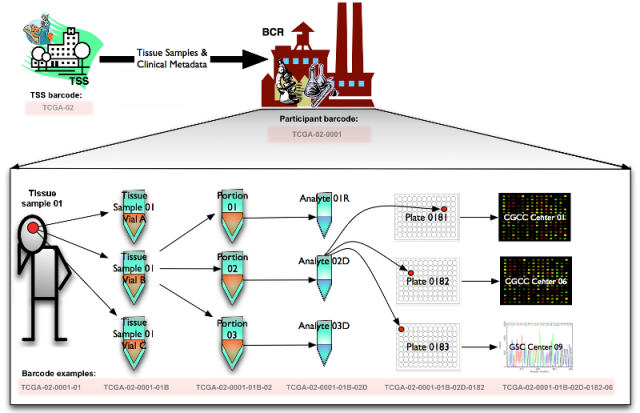
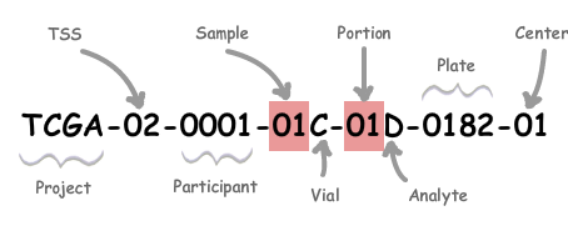
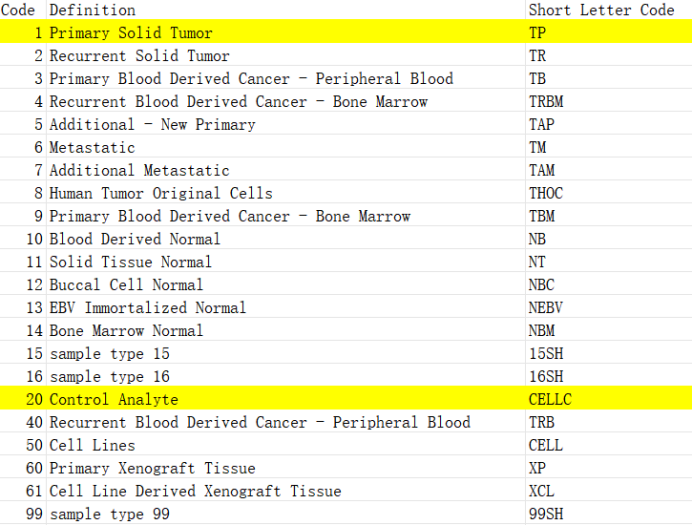
TCGA样本处理和命名方式参考https://docs.gdc.cancer.gov/Encyclopedia/pages/TCGA_Barcode/,代码表参考https://gdc.cancer.gov/resources-tcga-users/tcga-code-tables,我们可以发现一个病人的样本可能会分为多份进行测序,将命名TCGA-02-0001-01C-01D-0182-01分为六部分来看,TCGA为项目名,02为多形性胶质母细胞瘤,0001为病人号,01C为肿瘤的第三个样本(肿瘤类型范围为01至09,正常类型为10至19,对照样本为20至29),01D样本第一部分的DNA样本,0182为第182个96孔板,01为测序中心编号。其中,最终要的01C这个部分,我们用来判断是否为肿瘤,一般是01(原发性实体肿瘤)和20(对照)。
四、图像数据
回到最开始的地方,我们选择Repository。在这里我们可以发现更为详细的数据选择和类型。在这里主要包括Data Type(各种数据的类型和详细分类)、Workflow Type(使用软件)、Preservation Method(样本保存方式)等。
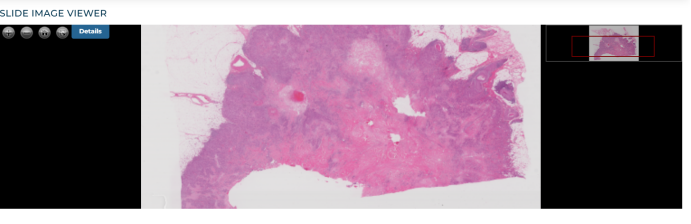
我们点击Data type下的slide image,这样会有幻灯片列表。

五、数据下载
这里推荐一个R包TCGAbiolinks。这里我们建议配合TCGA页面进行分析,包括你要下载的项目类型,数据类型,平台等https://github.com/BioinformaticsFMRP/TCGAbiolinks。
library(TCGAbiolinks)
library(SummarizedExperiment)
#查看可以下载的项目
> TCGAbiolinks:::getGDCprojects()$project_id
[1] "CTSP-DLBCL1" "TCGA-BRCA"
[3] "TCGA-LUAD" "CPTAC-3"
[5] "APOLLO-LUAD" "MATCH-B"
[7] "CMI-ASC" "MATCH-C1"
大家可以根据github上的示例进行下载,小编这里有一份注释好的教学代码,大家也可参考,关注微信号生信老学长,回复TCGAbiolinks_download领取。
VX公众号:生信老学长
专业项目指导和生信支持




















 1742
1742

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








