目前检测microRNAs的挑战是通常两个传统的PCR引物不适合miRNA目标,因为引物的组合长度几乎是microRNAs长度的两倍。现有的技术通过使用例如发夹引物,添加polyA尾,或通过连接添加片段延伸微microRNAs已经解决了这个问题,然而,这些方法缺损害了检测的灵敏度和特异性。此外,这些方法不能检测在3’端修饰的microRNAs,因为会干扰延伸过程。
BioVendor/欣博盛生物新推出检测miRNA的Two-Tailed RT-qPCR方法提供了一个优越的解决方案。Two-Tailed RT-qPCR不是使用单一的结合探针,而是使用两个半探针(hemiprobes),它们结合到microRNA的不同片段,并通过折叠的系绳连接。虽然每一个半探针都太短而不能结合microRNA,但当两者互补时,它们就会协同结合。
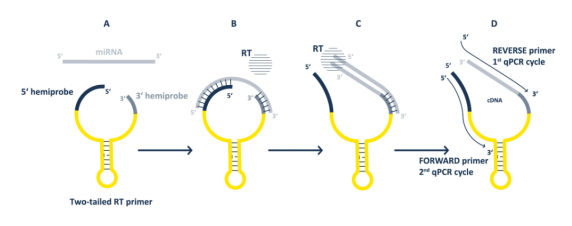
图1. Two-Tailed RT-qPCR检测原理
● Two-Tailed RT-qPCR检测优势:
1. 引物结合特异性强
2. 形成cDNA后可以使用2个序列特异性引物进行PCR扩增
3. SYBR以及水解探针均可以使用
4. 适用一管多色以及单色qPCR后的双管多色的反转录
● 产品列表
|
货号 |
产品名称 |
规格 |
|
RDTTRT50 |
Two-Tailed cDNA Synthesis System 50 rxn/欣博盛生物 |
50 rxn |
|
RDTTPCR150 |
Two-Tailed qPCR Master Mix 150 rxn/欣博盛生物 |
150 rxn |
|
RDTT000****PRI |
Two-Tailed RT/ PCR Primers/欣博盛生物 |
50 rxn/ 150 rxn |
****代表不同miRNA名称编号




 BioVendor/欣博盛生物新研发的Two-TailedRT-qPCR方法通过使用两个半探针提高microRNA检测的特异性,避免传统方法对灵敏度和3端修饰miRNA检测的限制。该技术允许高效扩增和适用于多种qPCR设置。
BioVendor/欣博盛生物新研发的Two-TailedRT-qPCR方法通过使用两个半探针提高microRNA检测的特异性,避免传统方法对灵敏度和3端修饰miRNA检测的限制。该技术允许高效扩增和适用于多种qPCR设置。
















 1万+
1万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








