作者 | 王汝恒编辑 | 龙文韬校对 | 李仲深
今天给大家介绍澳大利亚Monash大学的宋江宁教授与南京理工大学的於东军教授等人在Briefings in Bioinformatics期刊上发表的文章“SAResNet:self-attention residual network for predicting DNA-protein binding”。有关DNA-蛋白质结合特异性的知识对于理解基因表达、调控和基因治疗的机制至关重要。近年来,从序列数据出发的基于深度学习的预测DNA-蛋白质结合的方法取得了巨大的成功。尽管如此,目前的“S-O-T-A”计算方法仍存在一些缺点,这些缺点与使用实验数据不充足的有限数据集有关。为了解决这个问题,作者提出了一种新颖的基于迁移学习的方法,称为SAResNet,它结合了自注意力机制和残差网络结构,并在多个实验数据集上都取得了不错的效果。
一、研究背景
转录因子(TFs)是与DNA序列结合并调节基因表达的蛋白,在调节基因组功能中起主要作用,并且对个性化医学也具有重要意义。转录因子结合位点(TFBS)是TF结合的DNA片段,通常在4-30bp范围内。转录因子通常同时调节多个基因,在某种程度上,其在不同基因上的结合位点是保守的,但并不完全相同。因此,DNA-蛋白质结合的准确预测对于理解转录因子的生理作用,表征基因组的特定功能特征以及阐明在复杂生物体中如何精心编排高度特异性的序列表达程序非常重要。随着高通量测序技术的发展,多种实验方法可以在体内鉴定这些结合位点,例如ChIP-seq和SMiLE-seq。但是,这些方法昂贵且费时。在这种情况下,需要开发快速,准确的计算方法来鉴定DNA-蛋白质结合位点,因此,出现了许多基于机器学习的方法,而大多数方法使用浅层网络来拟合数据,因为某些数据集不足以支持深层网络的训练。其次,尽管这些算法使用的是浅层网络,但它们在小型数据集上的性能仍然不能令人满意。作者提出了迁移学习的解决方案,通过在大量相似任务的数据集上进行训练,然后在特定任务数据集上微调,并取得了显著成效。
二、模型与方法
2.1 特征表示
SAResNet的输入是由大小为L×4的二进制one-hot向量表示的DNA序列。L是DNA序列的长度(在实验中为101bp),“ 4”对应于碱基对的数量(A ,C,G,T)。在one-hot编码中,序列中的每个碱基对都表示为四个one-hot向量[1,0,0,0],[0,1,0,0],[0,0,1,0]和[0,0,0,1]中的一个,值1对应于该位置的核苷酸种类。如果将DNA序列视为具有四个通道(A,C,G,T)的一维序列,那么序列基序发现的任务就可以看作计算机视觉中的图像二分类任务。
2.2 Self-attention module
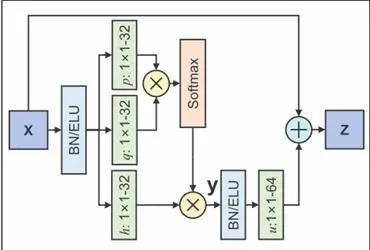
图1. Self-attention模块
由于通过卷积过程获得的信息通常仅限于局部邻域,因此仅使用卷积层来建模序列中的远程依存关系效率低下。在本文中,作者参考了非局部模型的思想,并提出了一个新的自我注意模块,该模块可以使模型有效地识别长距离分离区域之间的联系。
其中,x是前一个隐藏层的表示,y是注意力模块的输出特征。i和j分别是输入信号的序列x中某一位置的索引和枚举所有可能位置的索引。函数F计算i和所有j之间的注意力,函数h计算在位置j处的输入特征图表示(起到降维作用),N是x中的位置数。
在函数F中,序列特征通过函数p和q变换特征空间。在以上公式中,p、q和h是可学习的权重矩阵。可将序列特征由x的通道数变为1×1卷积核减少后的通道数。为了提高存储效率和模型精度,作者在这里均选择用32个卷积核卷积。最后,作者通过1×1卷积进一步扩充了注意力层输出的尺寸(64维),并将原来的x添加回特征表示中。因此,最终输出为:
2.3 Residual net module
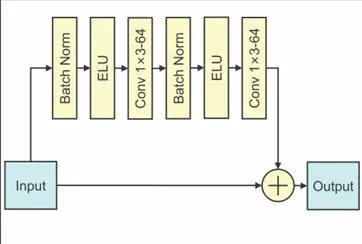
图2. Residual模块
通过增加神经网络的层数,可以增强模型的表达能力。然而,由于梯度更新的不稳定性,深层的传统前馈网络难以训练。在这方面,残差网络(ResNet)通过“短路连接”为该类问题提供了新的解决方案。本文采用的残差网络如下:
其中xl和xl +1分别表示第l个残差基本块的输入和输出,W1是与第l个残差块相关联的一组权重,而F代表残差函数。
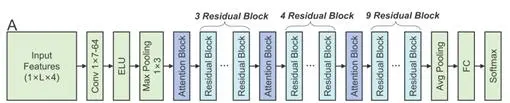
图3. SAResNet模型的网络结构
三、实验结果
3.1 迁移学习的效果
作者针对数据集中样本数量把众多数据集分为大中小三类,实验结果表明,在几乎所有测试数据集中,迁移学习的表现都优于直接学习(直接在某一个数据集上学习)和全局学习(在所有数据集上学习但不在单个数据集上微调)。图4显示,在不同规模的数据集上,迁移学习的性能优于直接学习和全局学习。
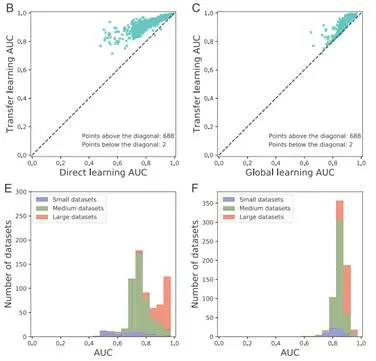
图4. 三类学习方式实验结果对比
3.2 Self-attention模块的消融实验
为了更好验证本文所提出的自注意机制的效果,作者在网络前三个残差模块前面添加了自注意模块,并与没有自注意机制的网络进行了对比实验。如表1所示,在添加了自注意机制之后,该模型几乎所有性能指标均得到了改进,从而说明了自注意机制的有效性和可靠性。
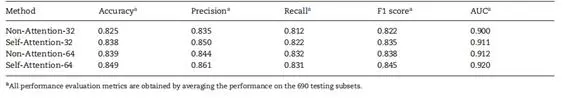
表1. 自注意模块消融实验
3.3 与“S-O-T-A”方法的比较实验
从表2中可以明显看出,SAResNet在三个不同规模数据集上的AUC(学生t检验,p <1.9×10-11)方面取得了统计学上显着的性能提升。与表现第二好的Expectation-Luo相比,SAResNet在三种类型的数据集上分别提高了4.9%,5.6%和2.0%。特别是在中小型数据集上,性能提升最为显著。
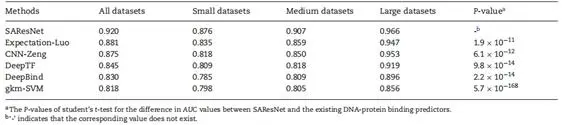
表2. SAResNet与其他方法的性能比较表
四、总结
在本文中,作者设计并实现了一种新颖的深度迁移学习方法SAResNet,以预测DNA序列中的DNA-蛋白质结合位点。特别是,作者结合了自注意力机制和残差结构来提出了一个新的深度学习架构,并通过迁移学习训练了网络模型。在基准数据集(690 ChIP-seq数据集)上的测试表明,作者提出的SAResNet的性能优于其他最新方法。
Web Server
http://csbio.njust.edu.cn/bioinf/saresnet/
参考文献
https://academic.oup.com/bib/advance-article/doi/10.1093/bib/bbab101/6218493




















 1531
1531

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








