本文内容速览:
在遗传信息从DNA转录为RNA再翻译为蛋白质的过程中,蛋白质能否正常发挥其生物学功能,不仅取决于遗传信息的准确传递,还受到DNA、RNA以及蛋白质本身多种化学修饰的协同调控。在分子生物学研究早期,DNA作为遗传信息的直接携带者,蛋白质作为细胞功能的主要执行者,两者成为最早受到广泛关注的研究对象。相比之下,RNA及相关化学修饰因检测手段和对其生物学意义认知的局限,长期以来受到的关注相对较低。随着检测技术和表观遗传领域的发展,RNA上的化学修饰逐渐被关注并成为研究热点。
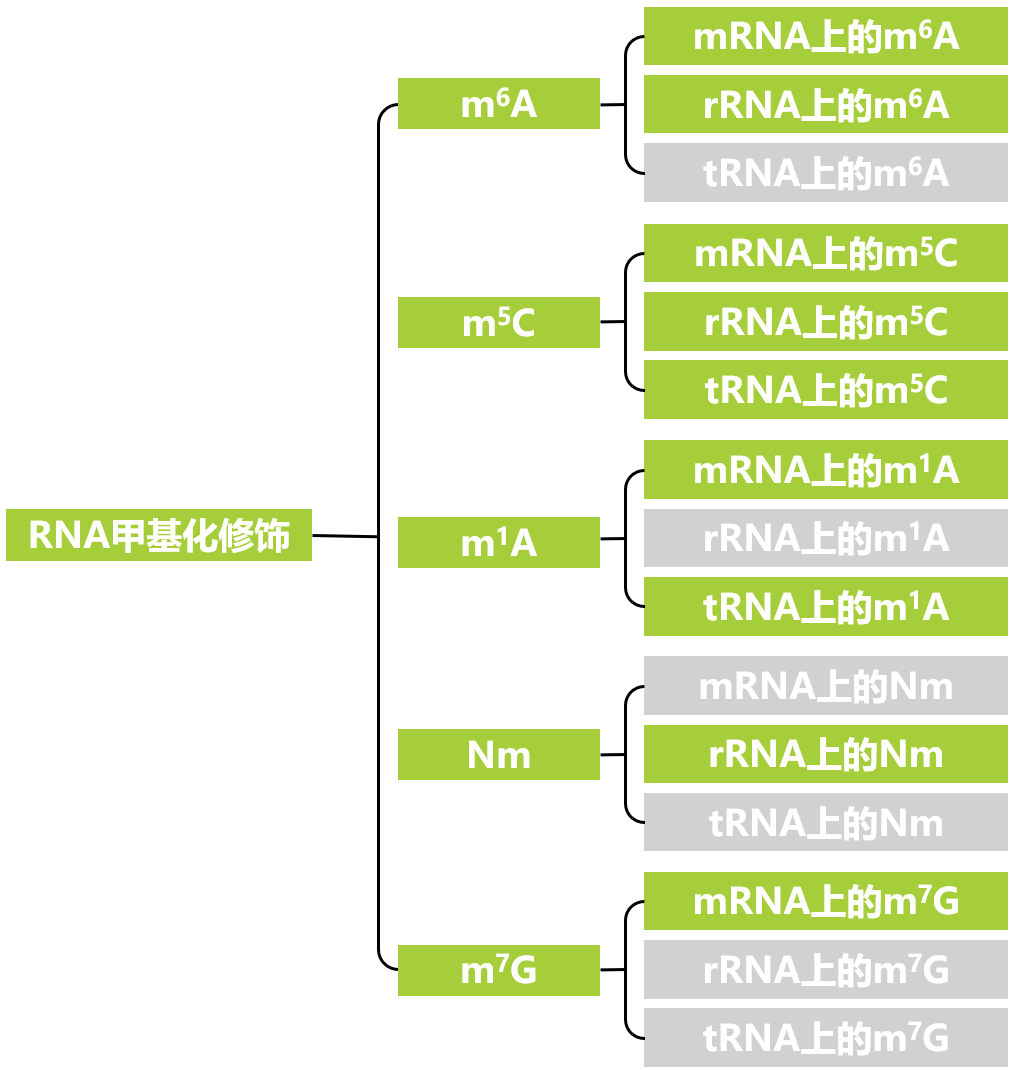
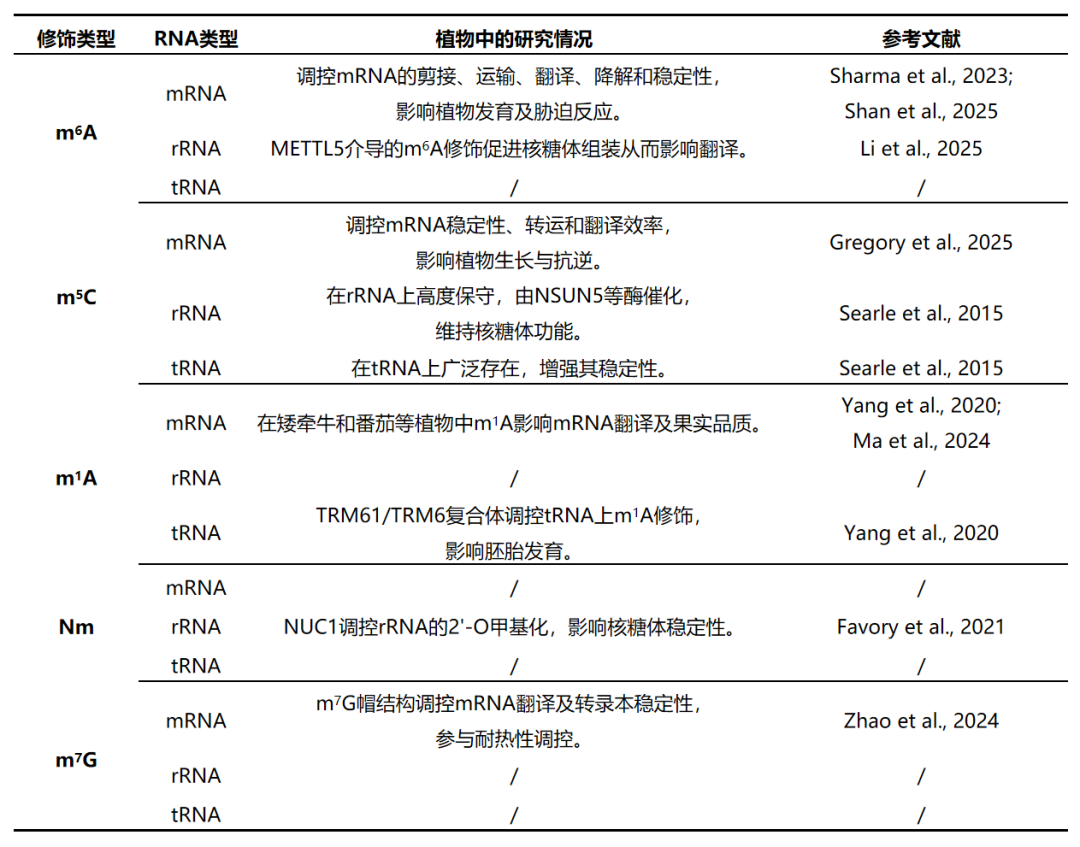
在“探索DNA甲基化:BS-seq技术的应用”以及“蛋白翻译后修饰研究套路”等往期推文中,已经介绍了有关DNA和蛋白修饰的相关内容,而RNA上的修饰介绍较少,因此,这篇推文小远将给大家分享RNA修饰,并着重介绍甲基化修饰相关的研究。在正式分享相关内容之前,小远统计了植物中研究过的RNA甲基化修饰类型以及对应的研究情况,以便大家对RNA甲基化修饰的研究情况有一个整体的了解(表1)。
表1 植物RNA甲基化修饰研究情况。
在我们阅读有关DNA甲基化和RNA甲基化的文章时,经常看到有各种各样的甲基化修饰简写的命名符号,例如m6A、6mA、m6A等,那这些简写中符号和数字的具体含义分别是什么?相信大家有时也很困惑。根据IUPAC命名规范,核酸甲基化修饰通常以小写“m”表示甲基,具体修饰位置用数字标注,可作上标或前缀(如m6A、6mA、m6A均为6-甲基腺嘌呤的规范简写)。该命名方式适用于DNA和RNA,并无数字前后与分子对象对应的规定。由此可见,甲基化的简写只要符合规范,不同的写法都是可以的,只是在文中我们最好需要单独说明一下,我们所用的简写具体表示的是哪种甲基化,以免造成误解。然而,在我们习惯的描述中,针对RNA甲基化的简写,我们还是更习惯将数字标注在“m”之后,如m6A;而DNA甲基化的简写,我们更习惯将数字标注在“m”之前,如6mA。
m6A
N6-甲基腺苷(N6-methyladenosine,m6A)是在RNA分子的腺苷(A,Adenosine)第6位氮原子上加上一个甲基基团(-CH₃),即m6A修饰(图1),该修饰是真核生物中最丰富的内源性修饰之一,过去几年间在各种植物中被广泛关注和研究(Sharma et al., 2023)。m6A作为一种可逆性修饰会受到特异性酶系统的调控,作为“写入器”的甲基转移酶可以为RNA加上m6A,作为“消除器”的去甲基酶可以去除RNA的m6A,作为“识别器”的结合蛋白可以识别RNA的m6A (Shan et al., 2025)。
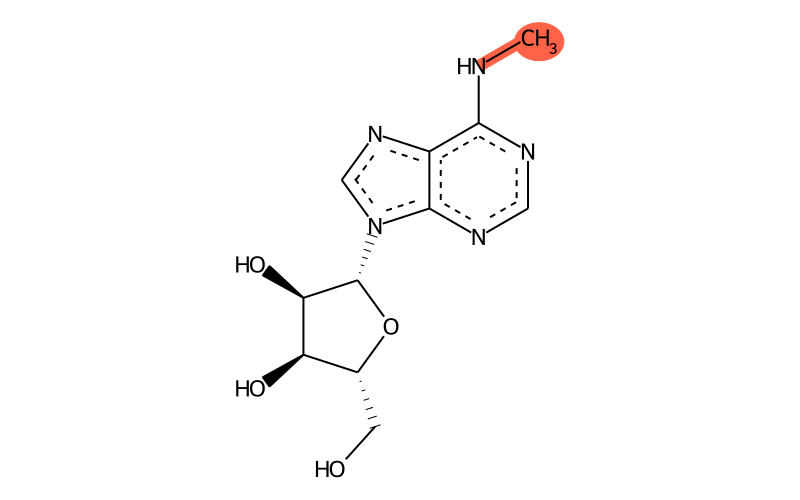
图1 m6A化学结构示意图(来源:Modomics)。
mRNA上的m6A
2025年5月,江苏大学曹军课题组在International Journal of Biological Macromolecules杂志上发表了一篇题为“A review of m6A modification in plant development and potential quality improvement”的综述文章,该文章中作者总结了m6A对mRNA的剪接、运输、翻译、降解和稳定性等方面的影响,进而在植物发育以及对生物和非生物胁迫的抵抗中发挥重要作用(图2)。
图2 m6A在植物发育和环境响应中的作用(Shan et al., 2025)。
rRNA上的m6A
2025年6月,新加坡国立大学申莉莎课题组在iMeta杂志上发表了一篇题为“METTL5-mediated 18S rRNA m6A modification enhances ribosome assembly and ABA response in Arabidopsis”的研究论文,该研究揭示了METTL5通过催化18S rRNA中A1771位点的m6A,促进RPL24A与核糖体的结合,进而促进80S核糖体的组装。这一过程对于解毒谷胱甘肽S-转移酶(GST)的mRNA翻译至关重要,有助于维持正常的活性氧(ROS)水平并确保对脱落酸(ABA)的正常反应。在mettl5突变体中,m6A1771的缺失破坏了RPL24A的掺入和核糖体组装,进而影响GST的翻译,导致ROS积累过多和对ABA过度敏感(图3)。
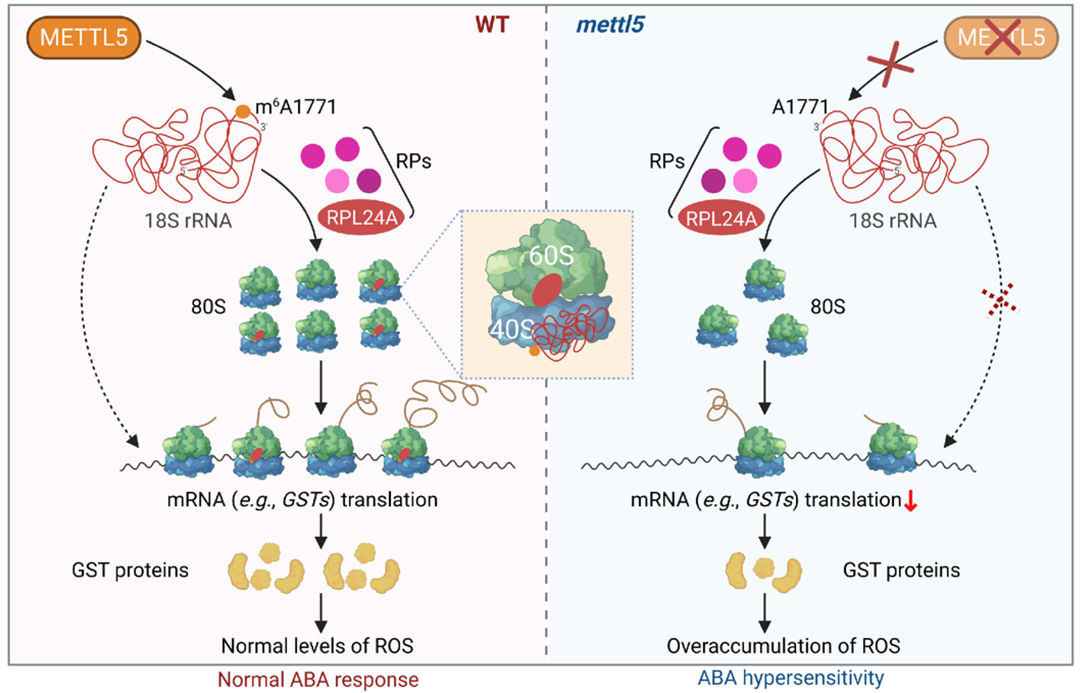
图3 rRNA的m6A促进RPL24A与核糖体的结合 (Li et al., 2025)。
m5C
5-甲基胞嘧啶(5-methylcytosine,m5C)是在RNA分子的胞嘧啶(C,Cytosine)第5位碳原子上加上一个甲基基团(-CH₃),即m5C修饰(图4)。m5C与m6A类似,也是真核生物中一种丰富的甲基化修饰,该修饰也是一种可逆修饰,同样可以被“写入器-消除器-识别器”这套酶系统调控 (Xue et al., 2020)。
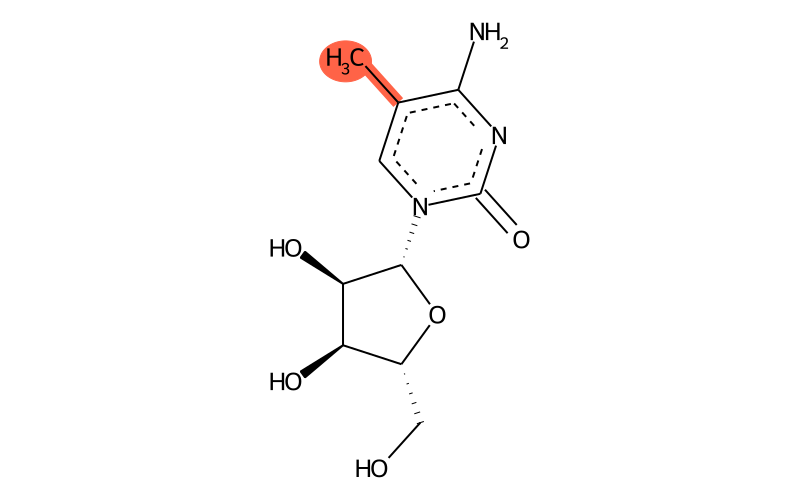
图4 m5C化学结构示意图(来源:Modomics)。
mRNA上的m5C
2019年8月,马克斯·普朗克分子植物生理学研究所Friedrich Kragler课题组在Current Biology杂志上发表了一篇题为“m5C Methylation Guides Systemic Transport of Messenger RNA over Graft Junctions in Plants”的研究论文,该研究发现m5C甲基化修饰能够促进mRNA在植物嫁接过程中跨越嫁接口实现系统性运输。缺乏m5C修饰的mRNA则无法高效地在砧木和接穗之间移动,说明m5C是mRNA实现长距离运输的关键决定因素(图5)。
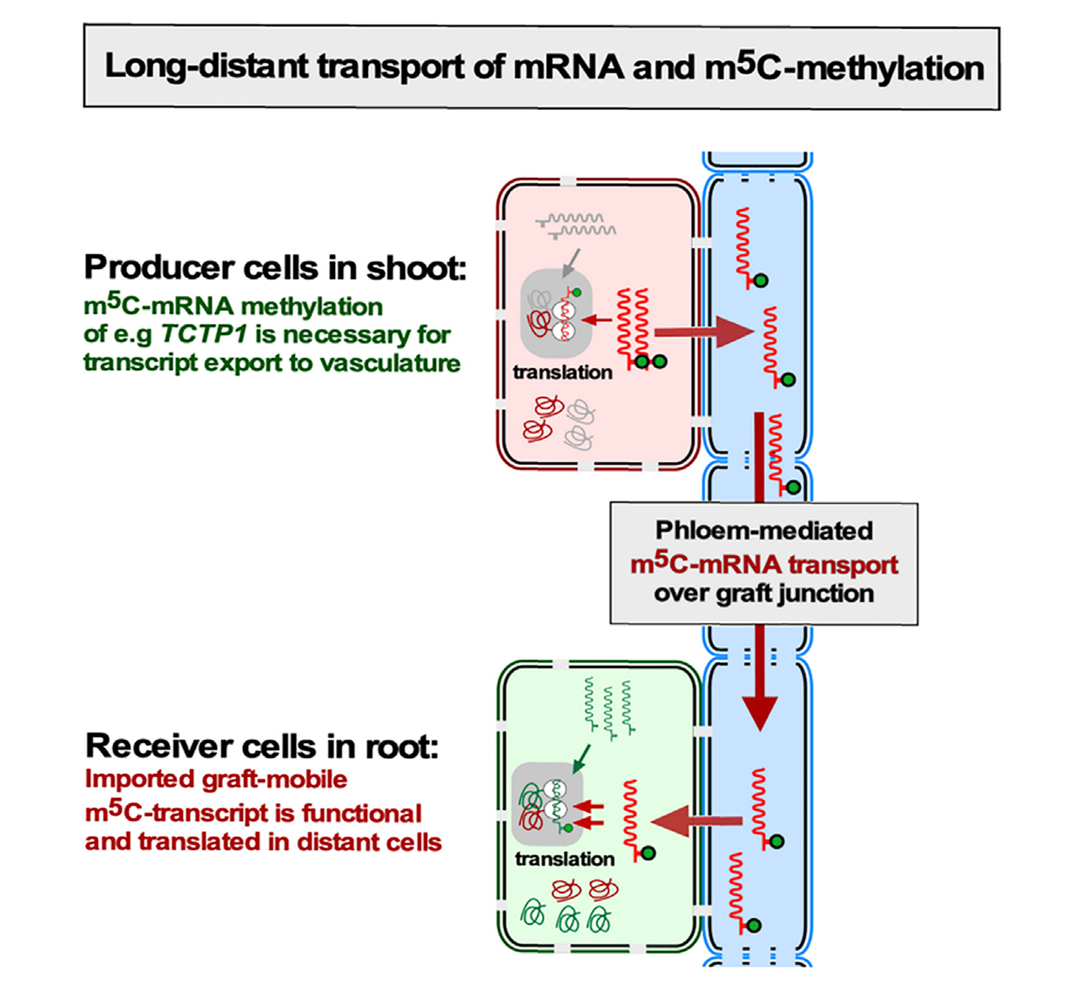
图5 m5C介导mRNA长距离运输示意图(Yang et al., 2019)。
2020年5月,中科院植物研究所种康课题组联合中科院北京基因组研究所杨运桂课题组在Developmental Cell杂志上发表了一篇题为“OsNSUN2-Mediated 5-Methylcytosine mRNA Modification Enhances Rice Adaptation to High Temperature”的研究论文,该研究发现水稻中mRNA的m5C修饰主要由甲基转移酶OsNSUN2介导。OsNSUN2的存在增强了水稻mRNA的m5C修饰水平,从而促进相关基因的稳定表达,提升了水稻对高温胁迫的适应能力。缺失OsNSUN2的水稻在高温下表现出更弱的适应性,表明OsNSUN2介导的m5C修饰对水稻耐高温具有重要作用(图6)。
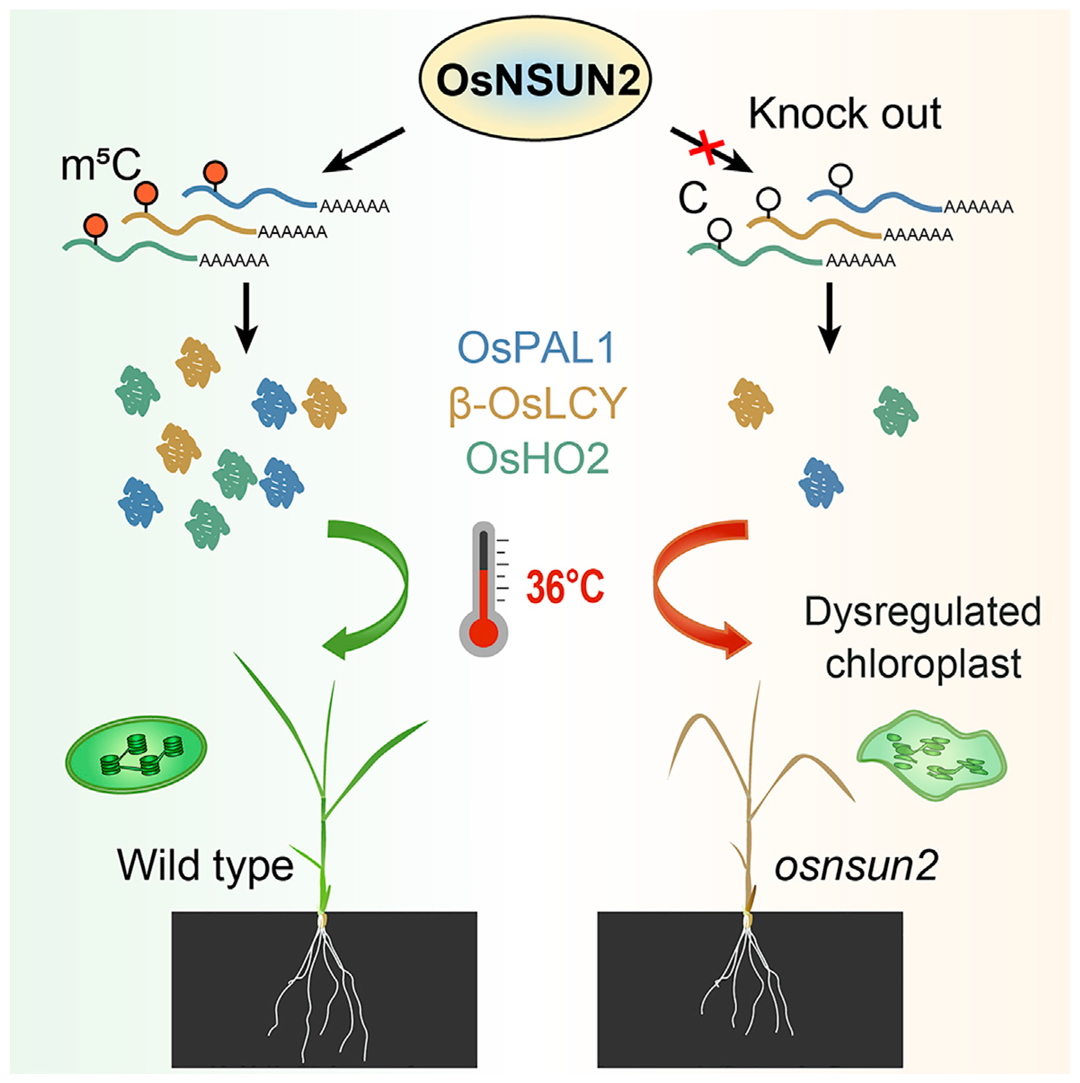
图6 m5C影响mRNA的翻译从而影响水稻对高温胁迫的适应(Tang et al., 2020)。
除了上面两个案例中提到的m5C影响植物mRNA的转运和翻译之外,David等人在“Transcriptome-Wide Mapping of RNA 5-Methylcytosine in Arabidopsis mRNAs and Noncoding RNAs”一文中还提及m5C可能影响mRNA的稳定性,不过作者没有深入研究,因此本文没有单独进行介绍。Bhat等人在“Epitranscriptomic modifications in plant RNAs”这篇综述文章中总结了m5C影响mRNA的稳定性、转运和翻译效率等方面,在植物生长发育以及抵抗非生物胁迫中起着重要作用。对这两篇文章感兴趣的读者可以自己去阅读原文。
rRNA和tRNA上的m5C
植物rRNA和tRNA上的m5C的相关研究较少,小远在查找资料的过程中发现有研究同时介绍了rRNA和tRNA上的m5C,因此这部分就一起介绍。2015年8月,阿德莱德大学Iain Robert Searle课题组在BMC Plant Biology杂志上发表了一篇题为“Conservation of tRNA and rRNA 5-methylcytosine in the kingdom Plantae”的研究论文,该研究在拟南芥中系统分析了m5C甲基化,发现核tRNA有39个位点、rRNA有7个位点,并且这些位点在植物界高度保守。与动物不同,植物线粒体和叶绿体tRNA普遍缺乏m5C。甲基转移酶TRDMT1和TRM4B负责核tRNA的m5C,甲基转移酶NSUN5负责rRNA的m5C(图7)。
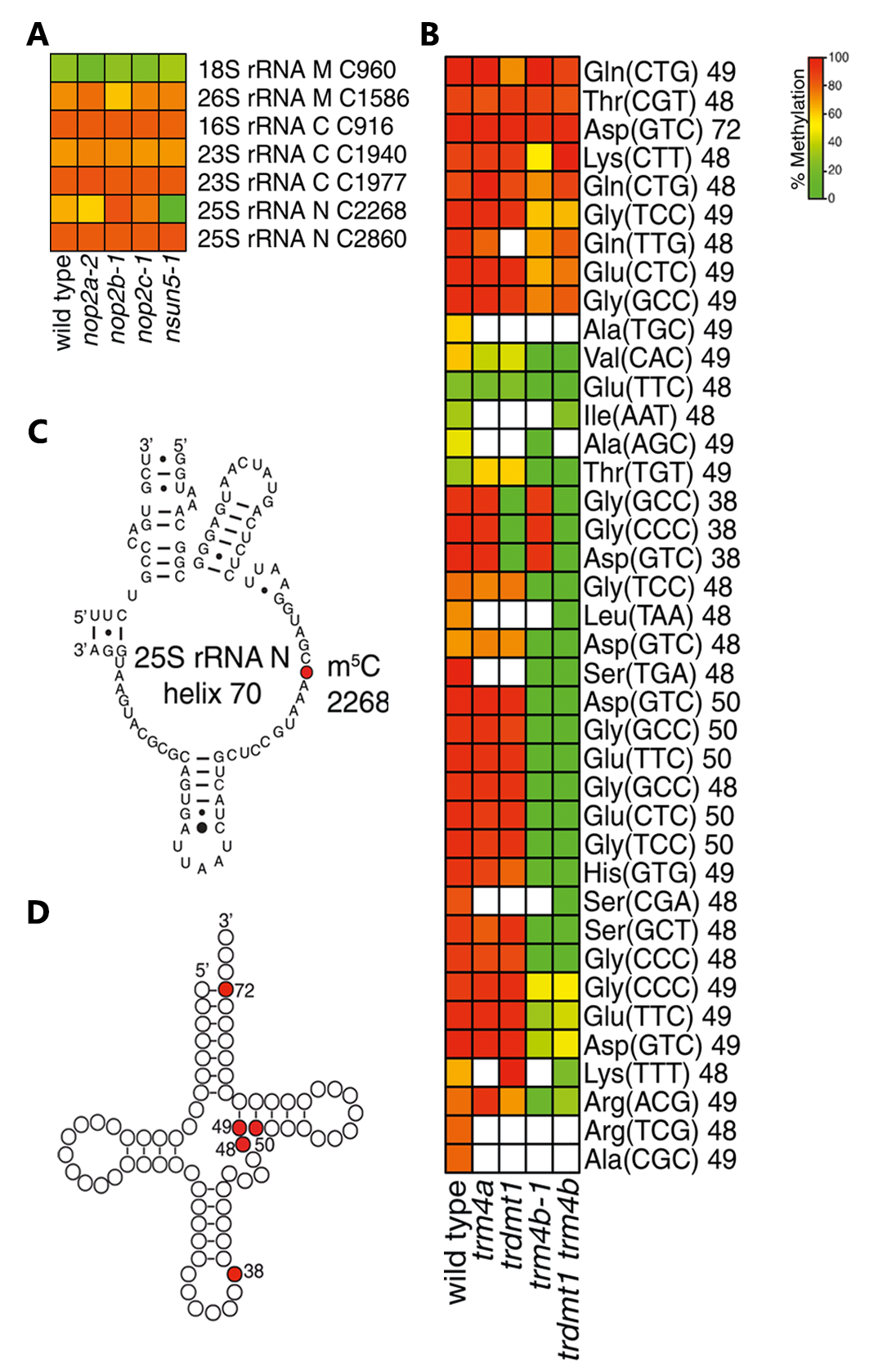
图7 野生型和甲基转移酶突变体中rRNA和tRNA上的m5C情况(Burgess et al., 2015)。(A)野生型及rRNA甲基转移酶突变体中rRNA 7个位点的m5C丰度差异;(B)野生型及tRNA甲基转移酶突变体中tRNA 39个位点的m5C丰度差异;(C)rRNA二级结构及m5C位点展示;(D)tRNA二级结构及m5C位点展示。
m1A
N¹-甲基腺苷(N¹-methyladenosine,m1A)是在RNA分子的腺苷(A,Adenosine)第1位氮原子(N¹)上加上一个甲基基团(-CH₃),即m1A修饰(图8)。该修饰同样属于可逆修饰,其“写入器”和“消除器”已有相关报道,但是“识别器”貌似还没有明确的报道(Sharma et al., 2023)。
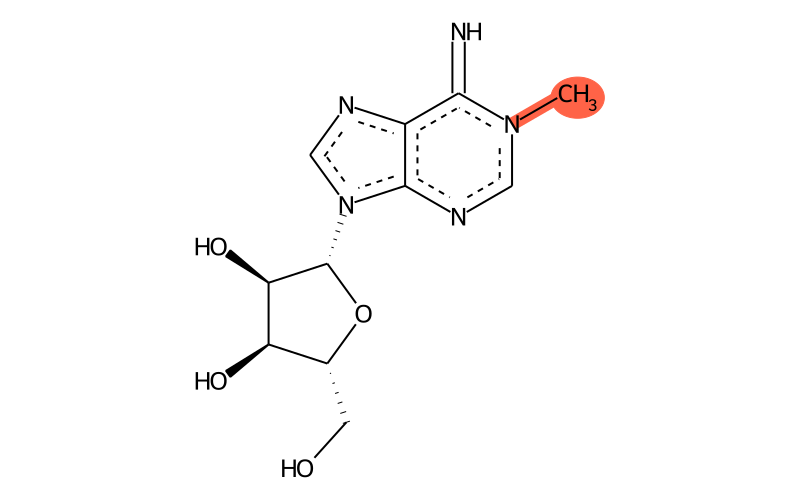
图8 m¹A化学结构示意图(来源:Modomics)。
mRNA上的m1A
m1A在植物mRNA中的丰度可能不如m6A与m5C,但是有研究发现在矮牵牛中m1A在mRNA上的丰度也很高(Yang et al., 2020)。目前m1A抗体在RNA修饰检测中被m⁷G干扰的风险,所以实际上m1A在mRNA上的丰度情况可能需要更精准和严谨的实验数据来说明(Grozhik et al., 2019)。不管实际情况如何,目前的相关研究显示m1A影响mRNA的翻译,在植物生长发育过程中发挥着重要作用(Ma et al., 2024; Yang et al., 2020; Zhao et al., 2025)。
2025年7月,浙江大学李栋栋课题组在Postharvest Biology and Technology杂志上发表了一篇题为“Dynamic abundances of m7G, m5C and m1A mRNA modifications are associated with tomato fruit quality following harvest”的研究论文,该研究中作者分析了几种RNA修饰在贮藏前后番茄果实中的丰度变化,结果发现,在贮藏后的番茄中m1A在mRNA中的丰度显著下降(图9)。作者分析,m1A可能影响果实成熟和品质相关基因的表达从而影响番茄果实在采后贮藏期间的生理变化。
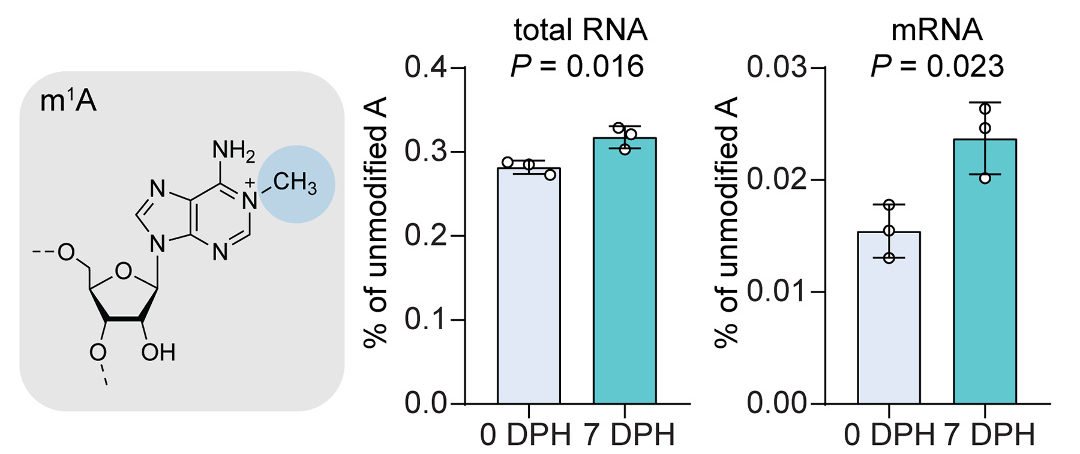
图9 番茄果实贮藏前后mRNA上m¹A的丰度变化情况(Zhao et al., 2025)。
tRNA上的m1A
2020年5月,中国科学院遗传与发育生物学研究所杨维才课题组在Journal of Experimental Botany杂志上发表了一篇题为“The Arabidopsis TRM61/TRM6 complex is a bona fide tRNA N1-methyladenosine methyltransferase”的研究论文,该研究发现,拟南芥中TRM61/TRM6复合体是tRNA上m1A修饰的专一甲基转移酶,其功能缺失会导致起始tRNA的m1A修饰水平显著下降,造成tRNA(tRNAiMet)稳定性下降,进而引发胚胎发育障碍和种子致死性表型(图10)。
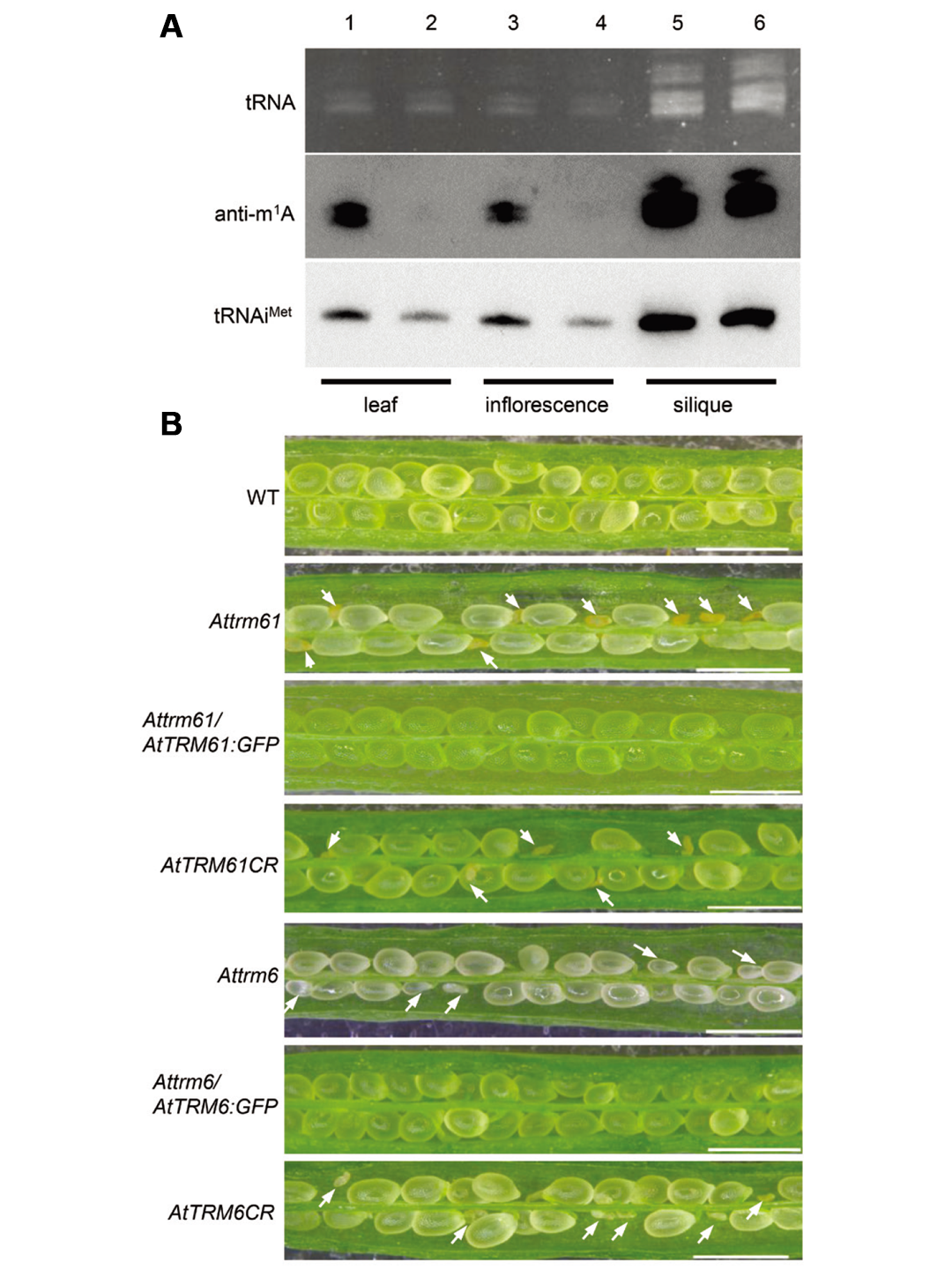
图10 TRM61/TRM6复合体通过介导m¹A修饰影响拟南芥tRNA的稳定性(Tang et al., 2020)。(A)不同组织中,野生型与Attrm61/LEC1pro::AtTRM61(Attrm61突变体材料中转入由胚胎特异性启动子驱动的AtTRM61)中的m¹A丰度差异;(B)野生型材料(WT)、突变体材料(Attrm61&Attrm6&ATTRM61CR&ATTRM6CR)、回补材料(Attrm61/AtTRM61:GFP& Attrm61/AtTRM6:GFP)长角果表型观察。
Nm
Nm(2’-O-甲基化,2’-O-methylation)是指在RNA分子的核糖核苷(N,Nucleoside)2’位羟基(2’-OH)上加上一个甲基基团(-CH₃),即Nm修饰(图11)。目前Nm修饰的“写入器”已有相关报道,而“消除器”和“识别器”还没有相关报道,因此按照目前研究的情况来看Nm可能是一种不可逆的修饰(Zhou et al., 2024)。
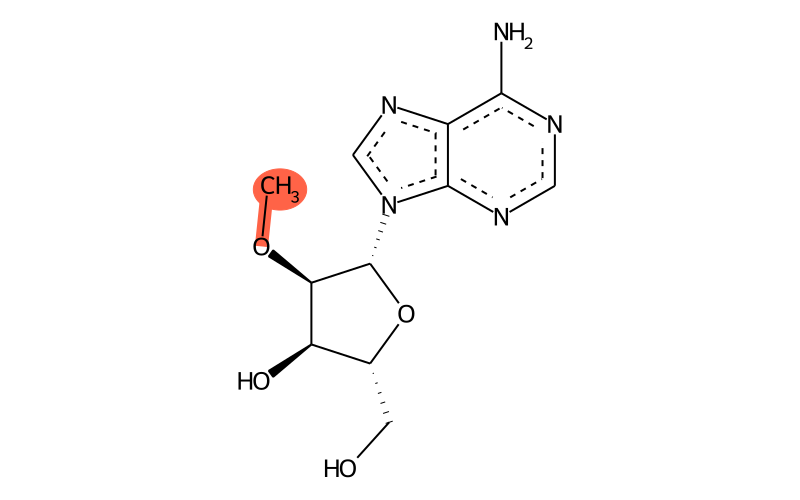
图11 Am化学结构示意图(来源:Modomics)。注:此处展示的为Nm中Am的化学结构,其余Nm修饰在核糖核苷相同位置加上-CH₃。
rRNA上的Nm
2021年2月,佩皮尼昂大学J Sáez-Vásquez课题组在RNA Biology杂志上发表了一篇题为“Mapping rRNA 2’-O-methylations and identification of C/D snoRNAs in Arabidopsis thaliana plants”的研究论文,该研究探究了拟南芥rRNA和snRNA上的Nm修饰位点,尤其是在rRNA的大亚基和小亚基中。NUC1是一种与RNA修饰相关的蛋白质,参与调控rRNA的Nm修饰(图12)。通过与C/D盒小核糖核酸(snoRNAs)协同作用,NUC1在特定rRNA位点的甲基化修饰中发挥着关键作用,这些修饰对于rRNA结构的稳定性和核糖体功能至关重要,进而影响植物的生长和发育。
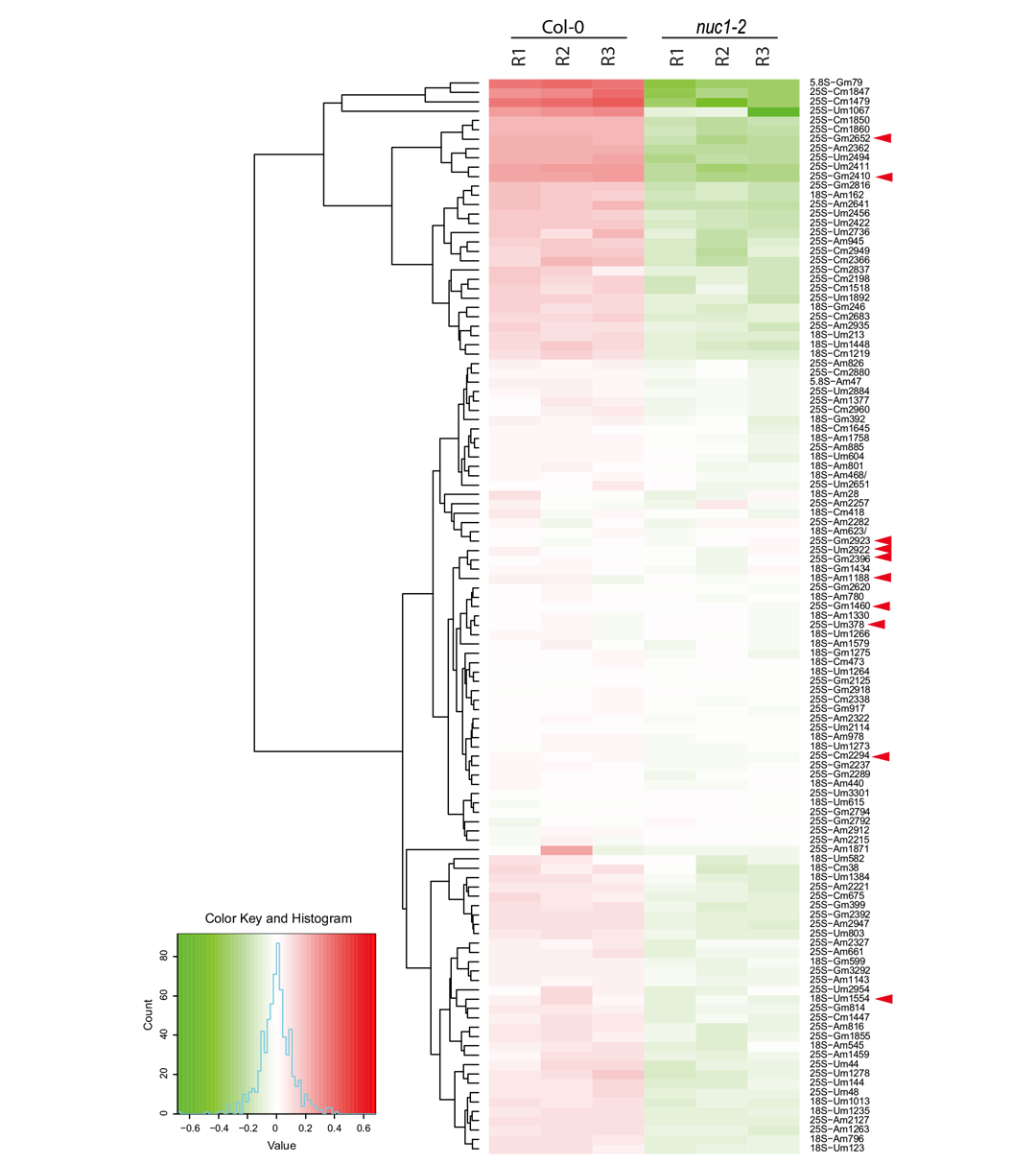
图12 NUC1参与调控rRNA的Nm修饰(Favory et al., 2021)。
m7G
m7G(7-甲基鸟嘌呤,7-methylguanosine)是指在RNA分子的鸟嘌呤(G,Guanosine)第7位氮原子(N7)上加上一个甲基基团(-CH₃),即m7G修饰(图13)。该修饰通常出现在真核生物的mRNA分子的5’端帽子结构中,也存在于tRNA和rRNA中,对于mRNA的翻译至关重要(Cowling 2009; Sharma et al., 2023)。m7G同样也可以被“写入器-消除器-识别器”所调控,例如鸟苷-7甲基转移酶(写入器)、去帽酶(消除器)以及eIF4E等结合蛋白(识别器)。
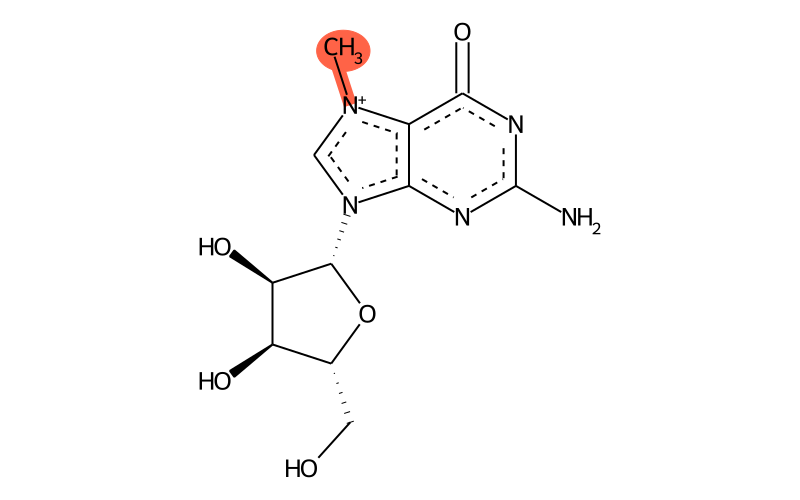
图13 m7G化学结构示意图(来源:Modomics)。
mRNA上的m7G
m7G帽子的形成主要与鸟苷-7甲基转移酶以及NAD-RNA去帽酶有关(Xiao et al., 2023)。有关m7G帽子介导的mRNA的翻译过程在往期推文“UTR中不可小觑的调控元件”有所介绍,感兴趣的读者可以回顾一下。
通常我们说到的m7G帽子都是指m7GpppN,即m7G的5’羟基与第一个转录的核苷酸(m7GpppN中的N)通过三磷酸链(m7GpppN中的ppp)连接。实际上,这种m7G帽子是Cap-0形式,还有两种其他类型的帽子形式分别是Cap-1(m7GpppNm)和Cap-2(m7GpppNmNm),主要区别在于m7G连接的第一个和第二个核苷酸是否存在Nm修饰。这两种类型的帽子在哺乳动物中占主导,而含Cap-0的mRNA会被识别成非自身RNA (Despic and Jaffrey, 2023)。有研究也探索了植物中是否存在Cap-1和Cap-2,研究发现并没有这两种Cap,说明不同物种中mRNA帽子修饰和功能存在进化分歧(Xiao et al., 2025)。
2024年10月,中国科学技术大学赵忠在Molecular Plant杂志上发表了一篇题为“Heat stress-induced decapping of WUSCHEL mRNA enhances stem cell thermotolerance in Arabidopsis”的研究论文,该研究揭示了MYB3R-like通过招募甲基转移酶RID2,直接激活WUSCHEL(WUS)基因,维持其mRNA的5’端m7G甲基化,防止其降解。进一步分析显示,WUS通过抑制蛋白质折叠相关基因,维持干细胞中蛋白质的高效合成。在热应激条件下,MYB3R-like/RID2模块的抑制导致WUS mRNA去帽,减少其转录本丰度,解除对干细胞蛋白质折叠相关基因的抑制作用,从而通过消除错误折叠的蛋白质聚集来增强细胞的耐热性(图14)。
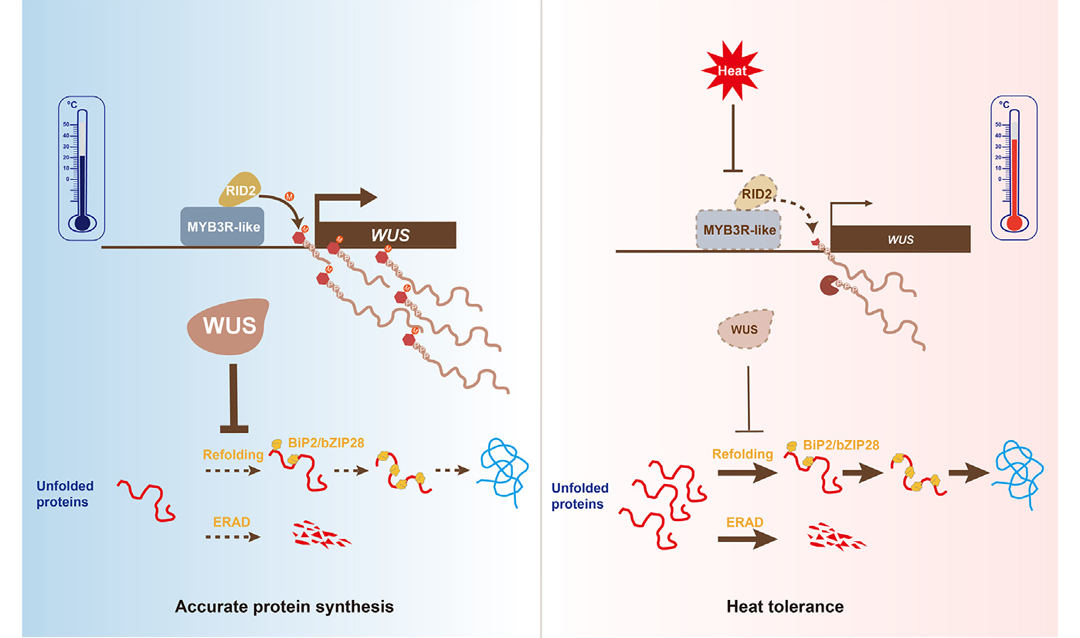
图14 MYB3R-like/RID2-WUS介导的干细胞耐热性(Liu et al., 2024)。
通过上述研究我们可以很清晰地感受到,植物中不同的RNA甲基化修饰在不同类型的RNA上的研究程度各有不同。m6A和m5C修饰虽然相关研究较多,但是这些研究也都主要聚焦在mRNA上,rRNA和tRNA上这两种修饰的研究还是相对比较少。相比之下,其他几种RNA甲基化修饰的研究就更少。在其他物种中,有关RNA甲基化修饰相关的研究有不少,有些甚至比植物中同类型修饰研究要更深入,这提示我们植物中RNA甲基化修饰还有很长的路要走、是一个值得研究的方向。
结合介绍的相关案例,小远对RNA甲基化修饰的研究思路进行了大致的总结,分为以下几个方面:
① 植物RNA甲基化研究首先聚焦于修饰类型的发现和修饰位点的精准鉴定。通过高通量测序技术(如MeRIP-seq、m6A-CLIP等),实现对mRNA、rRNA、tRNA等多种RNA分子的甲基化修饰在全基因组范围内定位,为深入研究甲基化的功能奠定基础;
② 在鉴定的基础上,进一步关注甲基化修饰的分子功能及作用机制。主要围绕“写入器”、“消除器”和“识别器”等关键蛋白,探究RNA甲基化对分子稳定性、剪接、翻译效率等多层次转录后调控的影响,明确其调控基因表达与表型的具体过程;
③ 系统分析RNA甲基化修饰在植物生长发育(如种子、花粉、胚胎等)和应对环境胁迫(如干旱、盐分、温度等)过程中的动态变化及功能意义,揭示其在植物适应性和抗逆性中的核心调控作用;
④ 拓展研究多种RNA修饰的协同调控,关注m6A、m5C等不同修饰类型在同一RNA分子或不同分子间的相互作用,探索其对转录后调控网络的复杂作用。
当前研究主要关注m6A、m5C、m¹A、Nm、m7G等修饰在植物中的作用,涉及mRNA、rRNA、tRNA等类型的RNA。这些修饰不仅在植物发育、环境应答及抗逆性中发挥重要作用,还调控RNA的稳定性、翻译效率、转运等。然而,RNA甲基化修饰的研究面临一些挑战,首先是识别修饰的复杂性,由于修饰在不同细胞类型、发育阶段和环境条件下的动态变化,甲基化图谱的精确构建仍然困难。其次,修饰间的交互作用尚未完全揭示,如何理解不同修饰在同一RNA分子中的协同作用是一个亟待解决的问题。功能验证不足也是当前研究中的一大难点,尽管有些修饰位点已确定,但缺乏对其具体生物学功能的深入研究。
References:
Azevedo-Favory J, Gaspin C, Ayadi L, et al. Mapping rRNA 2’-O-methylations and identification of C/D snoRNAs in Arabidopsis thaliana plants[J]. RNA biology, 2021, 18(11): 1760-1777.
Bhat S S, Paul M, Gregory B D. Epitranscriptomic modifications in plant RNAs[J]. RNA biology, 2025 (just-accepted).
Burgess A L, David R, Searle I R. Conservation of tRNA and rRNA 5-methylcytosine in the kingdom Plantae[J]. BMC plant biology, 2015, 15(1): 199.
Cowling V H. Regulation of mRNA cap methylation[J]. Biochemical Journal, 2010, 425(2): 295-302.
Despic V, Jaffrey S R. mRNA ageing shapes the Cap2 methylome in mammalian mRNA[J]. Nature, 2023, 614(7947): 358-366.
Grozhik A V, Olarerin-George A O, Sindelar M, et al. Antibody cross-reactivity accounts for widespread appearance of m1A in 5’UTRs[J]. Nature communications, 2019, 10(1): 5126.
Li P, Zhang Y, Zhang S, et al. METTL5‐mediated 18S rRNA m6A modification enhances ribosome assembly and ABA response in Arabidopsis[J]. iMeta, e70055.
Liu S, Wu H, Zhao Z. Heat stress-induced decapping of WUSCHEL mRNA enhances stem cell thermotolerance in Arabidopsis[J]. Molecular Plant, 2024, 17(12): 1820-1832.
Shan C, Dong K, Wen D, et al. A review of m6A modification in plant development and potential quality improvement[J]. International journal of biological macromolecules, 308(Pt 2): 142597.
Shan C, Dong K, Wen D, et al. Writers, readers, and erasers of N6-Methyladenosine (m6A) methylomes in oilseed rape: identification, molecular evolution, and expression profiling[J]. BMC Plant Biology, 2025, 25(1): 147.
Sharma B, Prall W, Bhatia G, et al. The diversity and functions of plant RNA modifications: what we know and where we go from here[J]. Annual Review of Plant Biology, 2023, 74(1): 53-85.
Shen L, Liang Z, Gu X, et al. N6-methyladenosine RNA modification regulates shoot stem cell fate in Arabidopsis[J]. Developmental cell, 2016, 38(2): 186-200.
Tang J, Jia P, Xin P, et al. The Arabidopsis TRM61/TRM6 complex is a bona fide tRNA N1-methyladenosine methyltransferase[J]. Journal of experimental botany, 2020, 71(10): 3024-3036.
Xiao C, Li K, Hua J, et al. Arabidopsis DXO1 activates RNMT1 to methylate the mRNA guanosine cap[J]. Nature Communications, 2023, 14(1): 202.
Xiao C, Li Q, Wu S, et al. mRNAs of plants and green algae lack the m7G cap‐1 structure[J]. The New Phytologist, 2025, 246(2): 396.
Xue C, Zhao Y, Li L. Advances in RNA cytosine-5 methylation: detection, regulatory mechanisms, biological functions and links to cancer[J]. Biomarker research, 2020, 8(1): 43.
Yang W, Meng J, Liu J, et al. The N1-methyladenosine methylome of petunia mRNA[J]. Plant physiology, 2020, 183(4): 1710-1724.
Zhao Z, Zeng S, Liu H, et al. Dynamic abundances of m7G, m5C and m1A mRNA modifications are associated with tomato fruit quality following harvest[J]. Postharvest Biology and Technology, 2025, 225: 113522.
Zhou K I, Pecot C V, Holley C L. 2’-O-methylation (Nm) in RNA: Progress, challenges, and future directions[J]. Rna, 2024, 30(5): 570-582.
Zhu C, Zhang S, Zhou C, et al. RNA methylome reveals the m6A-mediated regulation of flavor metabolites in tea leaves under solar-withering[J]. Genomics, Proteomics & Bioinformatics, 2023, 21(4): 769-787.




















 1164
1164

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








